Detta är anteckningar från föreläsning 4 i Harvard Extension’s Cell Biology course.
Den sekretoriska vägen hänvisar till det endoplasmatiska retikulumet, Golgiapparaten och vesiklarna som rör sig mellan dem samt cellmembranet och lysosomerna. Den kallas ”sekretorisk” för att den är den väg genom vilken cellen utsöndrar proteiner till den extracellulära miljön. Men som vanligt berättar etymologin bara en bråkdel av historien. Denna väg bearbetar också proteiner som kommer att vara membranbundna (antingen i cellmembranet eller i själva ER- eller Golgimembranen), liksom lysosomala enzymer och även alla proteiner som kommer att leva sitt liv i själva sekretoriska vägen. Den gör också en del andra saker än att bearbeta proteiner.
Cytosolen och ”lumen” (den vätska som fyller sekretorsgången) är olika kemiska miljöer och de blandas normalt aldrig. Cytosolen är reduktiv (när du befinner dig i cytosolen möter du hela tiden molekyler som vill erbjuda dig elektroner), och ER, Golgi och den extracellulära miljön är oxidativ (molekyler kommer hela tiden fram till dig och ber om elektroner). Se redox om du fortfarande är förvirrad. Detta ger olika förhållanden för proteinveckning: till exempel bildas disulfidbindningar vanligtvis endast under oxidativa förhållanden. Dessutom kan olika proteiner leva endast i den sekretoriska vägen eller endast i cytosolen. Den sekretoriska vägen ger cellen en väg att hantera saker som kanske inte är bra att ha i cytoplasman, och/eller som är mest användbara när de hålls koncentrerade i ett specialiserat fack med sina önskade interaktionspartners. Hepatocyter (i levern) binder läkemedel och gifter i det smidiga ER och bryter ner dem för att utsöndras från kroppen där. Den sekretoriska vägen är inte sammanhängande, utan varje rörelse mellan dess komponenter sker i små bubblande mikrokosmos av sin egen kemiska värld, så kallade vesiklar.
Många proteiner som går genom den sekretoriska vägen rör aldrig cytosolen – förutom de delar av membranproteiner som sticker ut på den cytosoliska sidan. Många av dem behöver chaperoner för att hjälpa till med veckningen och/eller en hel rad posttranslationella modifieringar för att vara redo för sin ursprungliga funktion, och den sekretoriska vägen är specialiserad på att förse dem med allt detta.
Dagens föreläsning kommer att fokusera på hur proteiner översätts till ER och hur de färdas (i vesiklar) mellan ER, Golgi och andra destinationer. Detta skildras vackert i videon Life of the Cell:
Det endoplasmatiska retiklet är det första steget i den sekretoriska vägen. Dess membran är kontinuerligt med det yttre kärnmembranet, även om det inte är klart varför det spelar någon roll, eftersom det inte är så att proteiner börjar sitt liv i kärnan. Snarare driver mRNA:erna runt i cytoplasman tills de plockas upp av en ribosom som är intresserad av att översätta dem. Vid ”posttranslationell translokation” flyttas det nya proteinet in i ER efter att det översatts. I det mer intressanta fenomenet som kallas ”cotranslational translokation” börjar ribosomen översättningen precis som vilket annat protein som helst, men någonstans i de första 16-30 aminosyrorna träffar den på en signalpeptid (även kallad signalsekvens). Signalmotivet är ofta en positivt laddad aminosyra följt av 6-12 hydrofoba aminosyror. Detta motiv känns igen av signal recognition particle (SRP, ett ”ribonukleoprotein” eller en hybridmolekyl av RNA och protein) som binder till det och hindrar ribosomen från att fortsätta översättningen. Översättningen stoppas tills ribosomen/SRP-komplexet möter en SRP-receptor på ER-membranet. När de möts binder SRP och dess receptor vardera en GTP-molekyl i ER-membranet, vilket tydligen stärker deras interaktion. Lyckligtvis sker allt detta i anslutning till en Sec61-translocon – ett proteinkomplex som bildar en kanal som korsar ER-membranet. Transloconen är egentligen ett komplex av tre olika proteiner (gener: SEC61A1 eller SEC61A2, SEC61B, SEC61G), varav Sec61a-underenheten har tio membranöverskridande a-helixer som bildar kanalen. När ribosomen väl är dockad vid membranet fortsätter den översättningen och driver signalpeptiden och så småningom hela proteinet genom kanalen in i ER-lumen. När översättningen upphör hydroliserar både SRP och SRP-receptorn sitt GTP för att frigöra varandra och ribosomens last (detta måste kräva energin från GTP, eftersom den ursprungliga bindningen skedde i nedåtgående riktning), ett signalpeptidas klyver signalpeptiden från det framväxande proteinet och proteinet är fritt för att börja veckas i ER.
Ett par andra aktörer är inblandade för vissa ER-proteiner. Oligosackaridtransferas, som lägger till glykosylgrupper till asparaginer i det nascenta proteinet, är en del av transloconkomplexet och utför faktiskt glykosylering medan det nya proteinet fortfarande översätts. Även om vi kallar glykosylering för en ”posttranslationell modifiering” görs den i det här fallet faktiskt under översättningen. För att vissa proteiner ska få sin rätta struktur måste de dessutom översättas fullständigt innan de tillåts börja veckas – om den N-terminala delen tillåts börja veckas så snart den kommer in i lumen skulle den få fel övergripande struktur. För att förhindra detta binder ibland Chaperon BiP proteinet för att hålla det oveckat ett tag. Föreställ dig BiP som en annan Pac-Man som biter sig fast i proteinet för att hålla det linjärt, som Hsc70 i den mitokondriella målinriktningsprocessen (se förra veckan).
Här är en video av det:
De första minuterna visar det grundläggande scenariot som beskrivs ovan. Sedan övergår den till ett mer komplext scenario som jag kommer att presentera om en minut. För övrigt visar videon två ”kontroversiella” saker som inte ingår i beskrivningen ovan: (1) signalpeptiden som bryts ned i membranet och (2) ett ”pluggprotein” som stoppar upp kanalen före/efter translation. Alla forskare är ännu inte överens om dessa två saker.
Alla proteiner som vi vet går genom den sekretoriska vägen pekades ut där av människor som gjorde lokaliseringsförsök för att se var i cellen ett protein ligger. Ett märkligt faktum om ER är att man kan sätta cellen i en mixer och efteråt kommer ER bara att börja återknyta till sig själv och bilda små ”mikrosomer” som inte är fästa vid kärnan utan bildar sammanhängande bubblor av ER. Man kan sedan börja leka med proteaser – som bryter ner proteiner – och tvättmedel – som löser upp ER-membranet. Om du antar att ditt protein av intresse är översatt kan du kontrollera om det (1) överlever proteasbehandling men (2) inte överlever proteas + detergentbehandling, då är det ett sekretoriskt vägprotein. Logiken är att i fall (1) var det skyddat inuti ER, men i fall (2) löste du upp ER, så det blev uppätet av proteaset. Allt detta förutsätter att du har en antikropp eller något annat sätt att upptäcka om det intressanta proteinet finns där efter dessa behandlingar.
Människor använde också sådana tekniker för att räkna ut att endast 70 aminosyror av ett nytt protein kan översättas innan det blir för sent för det proteinet att hamna i ER. Kom ihåg att signalpeptiden finns i de första 16-30 aminosyrorna, och translokationen till ER är beroende av att SRP är närvarande. Ribosomer översätter i en förutsägbar takt, så man fick ribosomer att börja översätta något mRNA och väntade sedan med bestämda mängder tid innan man tillsatte SRP, för att se hur mycket översättning som kunde ske innan SRP inte längre kunde göra sitt jobb.
SRP-receptorn och Sec61-proteinerna är ER-membranproteiner – och det finns många andra ER-membran-, Golgimembran- och lysosomemembranproteiner också. Faktum är att även cellmembranets membranproteiner (se klass 02) bearbetas i den sekretoriska vägen. Många av dessa har flera eller tiotals transmembrandomäner (20-25 hydrofoba aminosyror vardera) som måste sättas in i rätt ordning och orientering (till exempel vill man verkligen att jonkanalerna och transportörerna ska peka i rätt riktning, in mot ut ur cellen). Följaktligen finns det en massa fantasifulla biologiska mekanismer för att få in dessa proteiner i membranet på rätt sätt. Detta är vad den senare halvan av ovanstående video skildrar.
Så här är en tautologi: vissa proteiner har en topogen sekvens som bestämmer deras orientering i membranet. Denna sekvens består av två typer av signalsekvenser:
- En stopp-överföringssekvens (förkortad STA av någon anledning) är en 22-25 hydrofob aminosyrasekvens någonstans i mitten av proteinet som bildar en alfahelix. När den träffas skjuts den in i membranet, och sedan fortsätter översättningen av resten av proteinet i cytosolen. Detta ”ångrar” alltså på sätt och vis den translokation till ER som startades av signalpeptiden i början (N-terminus) av proteinet.
- En signalankringssekvens (förkortad SA) är också en 22-25aa hydrofob alfahelix, men med en serie av ~3 positivt laddade aminosyror till vänster eller höger om den. Liksom signalpeptiden känns den igen av SRP, som för ribosomen till ER. Men till skillnad från signalpeptiden kommer denna alfahelixsekvens att införas i ER-membranet. Införandets riktning bestäms av de tre positivt laddade aminosyrorna. De positiva laddningarna måste alltid hamna på den cytosoliska sidan, så om de kommer efter (dvs. C-terminal av) den hydrofoba sekvensen hamnar proteinet med sin C-terminala ände riktad mot cytosolen, men om de kommer före (dvs. N-terminal av) den hydrofoba sekvensen hamnar proteinet med sin N-terminal riktad mot cytosolen.
Med dessa två signaler som byggstenar kan man föreställa sig ett protein med en rad stoppöverförings- och signalankringssekvenser för att skapa en hel serie fram och tillbaka transmembrandomäner som sys in i membranet som av en symaskin. Man har klassificerat membranproteinerna i fem kategorier:
- Typ I har bara en signalpeptid och sedan en stoppöverföring i mitten. Därför hamnar den med sin (hydrofila) N-terminus i lumen, sin (hydrofoba) mitt i membranet och sin (hydrofila) C-terminus i cytosolen.
- Typ II börjar inte med en signalpeptid. Det börjar som vilket annat protein som helst, men i mitten har det en signalankringssekvens där +++ aminosyrorna kommer först och den hydrofoba serien efter. Detta gör att proteinet translokeras halvvägs genom översättningen, där den redan översatta N-terminala delen sticker ut i cytosolen (eftersom +++ måste förbli cytosoliska) och den nu begynnande C-terminala delen som ska översättas översätts direkt till ER. Så den hamnar i transmembran med sin C-terminus i ER och N-terminus i cytosolen – tvärtemot typ I.
- Typ III är som typ II – ingen signalpeptid, bara ett signalankare i mitten, men i det här fallet kommer +++ efter den hydrofoba sekvensen, vilket vänder på orienteringen. Så detta slutar med sin N-terminus i ER och sin C-terminus i cytosolen. Motsatsen till typ II och i slutändan samma som typ I, även om den kom dit på ett annat sätt – den har ingen signalpeptid som klyvs av i ER.
- Typ IV eller ”multipass”-proteiner har en omväxlande serie av signalsekvenser och stoppöverföringssekvenser. Dessa är helt klart mer än en ”typ”, men är ändå inte alls så olika som din kombinatoriska fantasi kan tillåta. Orienteringen av den första signalsekvensen avgör om N-terminus hamnar i cytosolen eller ER, och det totala antalet stoppöverföringssekvenser + signalankringssekvenser avgör var C-terminus hamnar: ett jämnt antal = samma sida som N-terminus, ett udda antal = motsatt sida som N-terminus. STA- och SA-sekvenserna måste vara strikt alternerande, med undantaget att man kan börja med två signalankringssekvenser om den första är orienterad med N-terminus mot cytosolen. Bara för att göra narr av detta kategoriseringsschema har folk definierat några ofullständigt definierade undertyper av typ IV, där typ IVa är N-terminal i cytosol (det börjar alltså som ett typ II-protein) och typ IVb är N-terminal i lumen (det börjar som ett typ III-protein, men har sedan en annan SA-sekvens som placerar det tillbaka i ER). GLUT1 från klass 02 är en typ IVa.
- GPI-förankrade proteiner, som är den femte typen men inte kallas typ V, börjar med en signalpeptid och slutar med en hydrofob C-terminus som förblir inbäddad i membranet. Den hydrofoba ändan klyvs av och ersätts med GPI, som också förblir inbäddad i membranet. PrP är en av dessa – mer om det senare.
Vidare har vi diskuterat hur proteiner kan hamna i ER-lumen eller spänna över ER-membranet. De flesta proteiner lämnar ER inom några minuter och transporteras i vesiklar som är bundna till Golgi och senare till utsöndring, lysosomer eller cellmembranen. Denna framåtriktade färdriktning kallas anterograd; att gå bakåt från Golgi till ER är retrograd transport.
Båda typerna av transport sker i membranbundna vesiklar. Dessa sprider sig från membranet i det land de kommer ifrån och smälter senare samman med membranet i det land de är på väg till – vackert skildrat vid ~2:25 i Life of the Cell-videon ovan. Den kropp från vilken vesiklarna bildas är ”donatoravdelningen”, och den destination som de senare smälter samman med är ”acceptatoravdelningen”.
För att knoppningsprocessen ska fungera krävs att G-proteiner i membranet rekryterar Coat-proteiner. För anterograd transport rekryterar G-proteinet Sar1 (gen: SAR1A) COPII (”cop two”); för retrograd transport rekryterar ett ARF G-protein COPI (uttalas ”cop one”). Dessa G-proteiner aktiveras för att utföra detta arbete när GEF laddar dem med GTP och byter ut GDP.
Så stegen i anterograd transport är till exempel följande:
- Sec12-GEF (Sec står för sekretorisk) laddar Sar1 med GTP. När Sar1 är bunden till GDP flyter den bara runt i donatorkompartmentet, men när den är bunden till GTP genomgår den en konformationsförändring som gör att dess annars begravda N-terminala hydrofoba svans sticker ut, vilket gör att den sticker in i membranet, där COPII-proteiner sedan börjar ackumuleras eftersom de verkligen gillar den svansen.
- COPII-proteinerna börjar polymeriseras och har på grund av sin konformation en inneboende preferens för krökning, så deras ackumulering börjar göra att knoppning sker. Samtidigt rekryteras membranbundna proteiner som måste transporteras – identifierade genom en DXE (dvs. aspartat-anything-glutamat) aminosyrasekvens som bildar en bindningsplats i deras cytosoliska del – till den nybildade vesikeln. Membranbundna proteiner fungerar som receptorer och rekryterar lumenala proteiner som är bundna till Golgi för att hänga i det konkava utrymmet där de kommer att hamna i vesikeln när den väl bildas.
- När tillräckligt många COPII har anlänt, knoppas vesikeln av. Sar1 hydrolyserar då sitt GTP, vilket ger den energi som krävs för att suga in sin hydrofoba svans i sig själv igen, vilket gör att COPII:erna lossnar. Vesikeln är nu frikopplad från donatorkompartmentet.
- Nu, av dåligt förklarade (eller dåligt förstådda?) orsaker, sönderdelas COPIIs päls och blottar receptorer under pälsen som styr vesikelns inriktning. När vesikeln väl anländer till sin destination interagerar Rab-GTP inbäddat i vesikelns membran med en Rab-effektor inbäddad i acceptörkompartmentets membran. En sidoblick utbyts, intresse väcks. Snart kommer vesikeln att smälta samman med membranet.
- SNARE-proteiner som finns på både vesikelns och målets membran (V-SNARE respektive T-SNARE) samverkar för att föra membranen ännu närmare varandra. I det här exemplet betraktar vi VAMP (VAMP_-generna) som V-SNARE och Syntaxin (STX__-generna) och SNAP25 (SNAP25-genen) som T-SNARE. Syntaxin och SNAP25 är båda membranproteiner; Syntaxin har 1 alfahelix och SNAP25 har 2, alla på den cytosoliska sidan. Alfahelikerna driver interaktionen med VAMP. De motsatta sidornas alfahelixer har extremt stark affinitet för varandra, vilket gör att membranen kommer tillräckligt nära varandra för att smälta samman. När detta väl har skett krävs det två proteiner för att få isär V-SNAREs och T-SNAREs igen: NSF (gen: NSF; står för NEM sensitive factor) och alfa-SNAP (gen: NAPA), ett lösligt NSF-anslutningsprotein. NSF är ett ATPas och förbränner ATP för att driva den energimässigt uppåtgående nedmonteringen av komplexet.
Nu kommer den retrograda transporten. Varför finns det retrograd transport överhuvudtaget? Här är en icke uttömmande lista över några orsaker:
- Vissa membranproteiner börjar sitt liv i ER, behöver modifieras i Golgi, men måste sedan återvända till ER. De gör detta med en KKXX-aminosyrasekvens.
- Det finns också en KDEL-aminosyrasekvens vid C-terminus på vissa lumenala proteiner som antas hålla dem kvar i ER, men det är inte perfekt – ibland hamnar de i Golgi, och i så fall riktas de tillbaka till ER via retrograd transport som är beroende av den KDEL-sekvensen för igenkänning. Mekanismen är ganska snygg – de proteiner som känner igen och binder till KDEL gör det bara vid lågt pH, och pH i Golgi är lägre än i ER, så de binder KDEL i Golgi och släpper det sedan när de är tillbaka i ER:s mer neutrala pH.
- Tänk också på att alla proteiner som deltar i anterograd transport – V-SNARES, Rab osv. – måste komma tillbaka till ER så att de kan göra om allt igen, på samma sätt som bussen måste komma tillbaka till bussdepån i slutet av dagen.
- Som vi kommer att se inom kort kommer Golgi i flera steg som är beroende av att enzymer läggs till längre nedströms.
Processen för retrograd transport skiljer sig inte så mycket från den anterograda. Den använder ARF i stället för Sar1, COPI i stället för COPII, men den fungerar på samma sätt: ARF laddad med GTP låter sin hydrofoba svans sticka in i membranet och drar till sig COPIs uppmärksamhet. COPI har två komponenter, COPIalpha och COPIbeta, som båda interagerar med KKXXX-sekvensen för att rekrytera membranbundna proteiner avsedda för retrograd transport. Vissa proteiner har också en RR-sekvens (någonstans i proteinet) som kan flagga dem för retrograd transport.
Golgiapparaten är inte sammanhängande. Den är en staplad uppsättning separata delfack som kallas säckar eller cisterner. Olika fack har olika egenskaper och proteiner besöker dem i en viss ordning. I ordning från ER till cellmembran kallas Golgi-avdelningarna för cis-, medial-, trans- och trans-Golgi-nätverket. Varje fack har olika enzymer som modifierar proteiner, och modifieringarna måste ske i en viss ordning, därav behovet av en staplad uppsättning fack.
Men när proteiner mognar i Golgi är det inte så att de knoppar av i vesiklar från ett fack och flyttar till nästa. Snarare är det så att den avdelning de redan befinner sig i rör sig utåt och ”mognar” när nya enzymer läggs till i den (från längre ner i Golgikedjan) via retrograd transport. Konstigt, eller hur? Det är ungefär som om du i stället för att flytta från en grundskola till en mellanstadieskola till en högstadieskola bara stannade i en och samma skolbyggnad under hela din barn- och ungdomstid, och de bara tog in nya läroböcker och lärare varje år för att hålla det lämpligt för den årskurs som du och dina klasskamrater nu hade nått. Så här ser Golgi ut när de rör sig och utvecklas:
Det finns alltså (lite eller) ingen anterograd transport inom Golgi, men gott om retrograd transport för att föra in varje ny omgång enzymer. När proteinerna äntligen har fullföljt hela K-12-läroplanen i Golgi-nätverket genomgår de en transport för att gå vidare till sin slutliga destination. De kommer att gå till ett av tre ställen:
- Exocytos – fusion med cellmembranet. På så sätt kommer lumenproteinerna att utsöndras extracellulärt, och membranproteinerna kommer att bli cellmembranproteiner.
- Sekretoriska vesiklar – dessa stannar bara kvar som vesiklar i cellen tills de behövs – där ”behövs” betyder att de så småningom genomgår exocytos. I neuroner är det här som neurotransmittorer lagras tills en aktionspotential kräver att de utsöndras i synapsen. I magen förvarar de celler som producerar magenzymer dessa enzymer i sekretoriska vesiklar tills födointag utlöser deras frisättning i magen.
- Lysosomer – dit felaktigt veckade proteiner går för att brytas ned.
Transporten från trans-Golgi-nätverket till dessa destinationer skiljer sig från den övriga transport som diskuterats ovan och involverar ofta clathrin (CLT__-gener). Vesiklar som knoppar av har en tvåskiktsbeläggning, med adapterproteinkomplex (AP) som det inre lagret och clathrin som det yttre lagret. Adapterproteinerna har en målsignal med ett YXXh-motiv (h = Φ = vilken hydrofob aminosyra som helst). Clathrin bildar den så kallade ”clathrin-triskelion”-formationen som visas här:

(Bild tack vare Wikimedia Commons-användaren Phoebus87)
Clathrin är också ansvarigt för endocytos – att vesiklar med extracellulära saker (och cellmembranproteiner) knoppas av för att komma in i cellen. Detta kallas clathrinmedierad endocytos. Receptorer i cellmembranet endocytoseras mycket ofta: hela populationen av hormonreceptorer vänds om ungefär varje timme, särskilt när hormoner tas emot. Att ta upp receptorn i en vesikel är ett sätt för cellen att stänga av den inkommande signalen tills den kan bearbetas.
I anteckningarna om plasmamembranen diskuteras cystisk fibros kortfattat: CFTR är en ABC-transportör som ansvarar för att pumpa ut Cl- ur cellen (den släpper också in Na+). Mutanter med förlust av funktion pumpar inte Cl-, vilket tar bort drivkraften för osmos, vilket gör slemmet tjockare och orsakar andningsproblem. Det finns minst 127 olika CFTR-mutanter med funktionsförlust (åtminstone är det så många som Natera testar för) som (om båda allelerna är inaktiverade) orsakar cystisk fibros. Den vanligaste mutationen är ΔF508, som utgör ~3 % av alla europeiska CFTR-alleler och cirka 70 % av de muterade. Förlusten av denna ena fenylalanin ändrar CFTR:s konformation så att den di-sura utgångskoden (aminosyrorna D565 och D567) som riktar CFTR till exocytotiska vesiklar inte längre exponeras på rätt sätt och proteinet når aldrig fram till cellmembranet .
diskussionsavsnitt
I avsnittet läser vi Hu 2009, som visade att atlastinproteiner är involverade i skapandet av det tubulära ER-nätverket. Bevisen kom nästan helt och hållet från protein-proteininteraktioner. Jag blev förvånad över att den här artikeln var en stor grej, eftersom det har funnits en miljon artiklar som visar protein-proteininteraktioner för huntingtin, och ingen tror egentligen på alla, och det har inte nödvändigtvis fört oss närmare en kunskap om vad huntingtin gör eller vad som går fel vid Huntingtons sjukdom. Men tydligen kunde Hu göra en ganska ren argumentation för att atlastinernas interaktioner med reticuloner innebär att de har en roll i ER-bildningen. Det underlättar att Hu kunde visa på en ”genetisk interaktion” utöver en fysisk (bindande) interaktion. En ”genetisk interaktion” (jag var tvungen att slå upp det) innebär när ”Ibland ger mutationer i två gener upphov till en fenotyp som är överraskande mot bakgrund av varje mutations individuella effekter. Detta fenomen, som definierar genetisk interaktion, kan avslöja funktionella relationer mellan gener och vägar.” .
PrP
Detta är ett decennium gammalt, så vissa saker kan vara föråldrade, men jag tyckte att Harris 2003 (ft):s genomgång av PrP:s cellbiologi var extremt tydlig och användbar. Kim & Hegde 2002 var också till hjälp. PrP är ett sekretoriskt protein. Dess första 22 aminosyror (MANLGCWMLVLFVATWSDLGLC) är en signalpeptid som orsakar cotranslationell translokation till ER. Normalt blir PrP bara GPI-linkat vid sin C-terminus och förankras på den exoplasmatiska sidan av membranet. Men aminosyrorna 111-134 (HMAGAAAAGAVVGGLGGGGYMLGSAM) är en slags svag signalankringssekvens (typ II, där +++ aminosyrorna kommer före signalankret) som ibland men inte alltid blir en transmembrandomän, vilket inverterar C-terminalen in i lumen. Ännu mer förvirrande är att den sekvensen ibland bara kan sluta som en transmembrandomän utan invertering, så att N-terminus ligger i lumen. Det finns alltså tre membrantopologier för PrP: vanlig gammal GPI-förankrad och två transmembranorienteringar, som visas i Harris 2003 Fig 3:
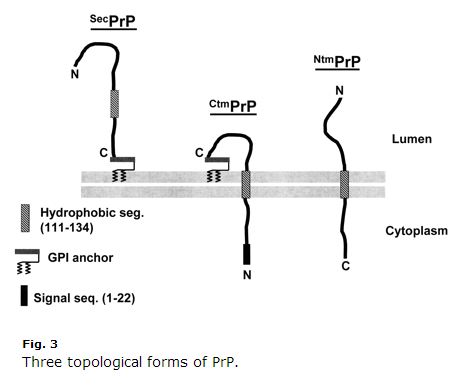
Bemärk hur konstig CtmPrP är. Den är transmembran men också GPI-förankrad, och den N-terminala signalpeptiden klyvs aldrig bort. Normalt utgör transmembranformerna < 10 % av det totala PrP. Under vissa laboratorieförhållanden är andelen högre, och två av de mutationer som orsakar GSS (A117V och P105L) ökar också andelen CtmPrP till 20-30 % av allt PrP. Av dessa tre former finns det en hel del bevis för att CtmPrP är giftigt och att det kan spela en roll i prionbildningen, även om de flesta genetiska prionsjukdomsmutationer (inklusive FFI D178N) inte tycks påverka PrP:s membrantopologi eller fraktionen CtmPrP.
När PrP passerar genom Golgi är det målinriktat mot cellmembranet. Men enligt Harris sitter det inte bara där – det genomgår ofta clathrinmedierad endocytos och cyklar genom cellen var ~60 minut, där vissa molekyler klyvs vid varje cykel. Koppar stimulerar denna endocytos av PrP. De flesta genetiska prionsjukdomsmutationer ändrar lokaliseringen av PrP – vanligtvis när en mutation föreligger finns mindre PrP på cellytan och mer ansamlas i ER.