Vad är ChIP?
Kromatinimmunuttag, eller ChIP, är en antikroppsbaserad teknik som används för att selektivt anrika specifika DNA-bindande proteiner tillsammans med deras DNA-mål. ChIP används för att undersöka en viss protein-DNA-interaktion, flera protein-DNA-interaktioner eller interaktioner över hela genomet eller en undergrupp av gener.
ChIP använder antikroppar som selektivt känner igen och binder proteiner, inklusive histoner, histonmodifieringar, transkriptionsfaktorer och kofaktorer, för att ge information om kromatintillstånd och gentranskription. En kombination av proteomanalys och molekylärbiologiska tekniker som används i ChIP ger möjlighet att förstå genuttryck och reglering i celler eller vävnader av intresse.
När används ChIP?
Typiskt används ChIP för att identifiera den relativa förekomsten av ett specifikt protein eller en specifik proteinmodifiering vid en viss region i genomet. ChIP kan användas för att besvara en mängd vetenskapliga frågor som rör interaktionen mellan proteiner och kromatin. ChIP kan till exempel användas för att jämföra närvaron av vissa proteiner vid olika loci, kartlägga de olika proteinerna över en intressant genomisk region eller kvantifiera proteinbindning till en inducerbar gen som svar på ett stimulus över tid.
Hur fungerar ChIP?
Principen bakom ChIP är relativt enkel och bygger på att en antikropp används för att isolera, eller fällas ut, ett visst protein, histon, transkriptionsfaktor eller kofaktor och dess bundna kromatin från en proteinblandning som extraherats från celler eller vävnader. Därav teknikens namn: Kromatinimmunprecipitering. I ChIP-PCR eller ChIP-seq kan immunberikade DNA-fragment sedan identifieras och kvantifieras med hjälp av allmänt tillgängliga PCR- eller qPCR-reagenser och Next Generation Sequencing-teknik (NGS).
Vad är native ChIP (N-ChIP) jämfört med crosslinked ChIP (X-ChIP)?
Det finns 2 typer av ChIP-tekniker som kan utföras beroende på den experimentella frågeställningen och utgångsmaterialet för experimentet: 1) native ChIP (N-ChIP) och 2) crosslinked ChIP (X-ChIP). Båda typerna av ChIP har för- och nackdelar:
- I N-ChIP används inget fixeringsmedel för att korslänka proteiner till kromatinet. Istället isoleras nativt kromatin från cellkärnor som smälts med ett nukleas. Eftersom antikroppar framställs mot ofixerade antigener har N-ChIP fördelen att antikropparna bättre känner igen och binder till sina målantigener. PCR kanske inte behövs för nedströmsanalys på grund av den höga förekomsten av histonproteiner. Även om dessa fördelar gör N-ChIP till en attraktiv metod kan den endast användas för detektion av histoner. Dessutom kan förlust av proteinbindning under kromatinförstöringen och immunoprecipiteringsstegen snedvrida data eller hindra korrekta analyser.
- I X-ChIP används kemiska fixeringsmedel, t.ex. formaldehyd, för att tvärbinda det intressanta proteinet till DNA:t, och fragmentering av kromatinet åstadkoms med hjälp av sonikation eller nukleasförstöring. Fördelen med X-ChIP är att den kan användas med histon- och icke-histonproteiner och att den i allmänhet kräver mindre cellulärt utgångsmaterial än N-ChIP. X-ChIP minimerar också risken för förlust av kromatinprotein under extraktionen, vilket gör det möjligt att detektera övergående proteininteraktioner. Utfällningssteget är dock mindre effektivt och DNA-amplifiering genom PCR är nödvändig för nedströmsanalyser.
Vad är de olika typerna av ChIP-analyser?
När själva kromatinimmunutfällningen är avslutad kan flera nedströmsanalyser utföras på det renade kromatinet och de associerade proteinerna, histonerna, transkriptionsfaktorerna och kofaktorerna. De vanligaste metoderna för analys av enskilda gener och analys av hela genomet är qPCR respektive ChIP-seq. PCR och ChIP-chip är också alternativ för nedströmsanalys.
5.1 Vilka är fördelarna med ChIP-PCR?
ChIP-PCR utförs för att analysera histonmodifieringar och/eller proteinbindning till en känd undergrupp av mållokier i genomet. Vid ChIP-PCR identifieras och kvantifieras immunberikade DNA-fragment med hjälp av allmänt tillgängliga PCR- eller qPCR-reagenser och tekniker. Snabba och kvantitativa jämförelser av specifika regioner inom genomet i flera prover kan uppnås med hjälp av ChIP-qPCR. Detta är billigare och mer tidseffektivt än metoder för sekvensering av hela genomet.
5.2 Vilka är fördelarna med ChIP-chip?
ChIP-chip-tekniken avser utnyttjandet av ett DNA-mikroarray-chip för att analysera ChIP-immunberikade DNA-fragment. Användning av genomtiling-mikroarrayteknik möjliggör en helgenomanalys av proteiner som är bundna till isolerat DNA och genererar en genomisk karta med hög upplösning över proteinbindning och proteinmodifieringar. ChIP-chip har flera användningsområden inom såväl grundforskning som sjukdomsbaserad forskning. Det kan till exempel användas för att identifiera bindningsställen för transkriptionsfaktorer, förstärkare och repressorer och för att jämföra dessa typer av bundna proteiner i kontroll- och patologiska prover. Eftersom kostnaden för NGS har minskat avsevärt och liknande resultat erhålls med hjälp av ChIP-seq är det dock fler som väljer att utföra ChIP-seq istället för ChIP-chip.
5.3 Vilka är fördelarna med ChIP-seq?
I likhet med ChIP-chip ger ChIP-seq information om proteinbindning över hela genomet. Till skillnad från ChIP-chip använder ChIP-seq dock NGS-teknik för att identifiera DNA-fragment och kartlägga dem mot hela genomet.
Med modernare DNA-amplifieringsteknik är det möjligt att genomföra robusta analyser på några dagar med en liten mängd ingående DNA. När utgångsmaterialet är knappt har dessa tekniska framsteg i metoderna för biblioteksberedning gjort ChIP-seq-experiment möjliga.
Den nya tekniken där DNA-proverna är unikt märkta med korta sekvenser, så kallade streckkoder, gör det nu möjligt att samla enskilda fragment i en enda sekvenseringsfil för multiplexerad analys. Detta har avsevärt ökat effektiviteten och minskat kostnaden för DNA-sekvenseringsexperiment, vilket ytterligare stöder ChIP-seq-tillämpningar.
Tillsammans är fördelen med ChIP-seq, på grund av framstegen inom DNA-sekvenseringstekniken, att ett stort antal ChIP-berikade DNA-prover kan sekvenseras billigt på relativt kort tid med högre känslighet och noggrannhet än ChIP-chip.
Vilka är de olika stegen i ChIP-analysen?
ChIP-analyser följer ett allmänt protokoll:
- Korslänkning av proteiner till DNA endast för X-ChIP
- Celllysning
- Kromatinfragmentering genom digestion (för X-ChIP och N-ChIP) eller sonikation-
- .(endast för X-ChIP)
- Immunoprecipitation med hjälp av specifika antikroppar
- DNA-rening för nedströmsanalyser
- DNA-analys via PCR, qPCR, mikroarray eller NGS
Väsentligt är att positiva och negativa kontroller i varje steg är viktiga för att avgöra om ett ChIP-experiment har varit framgångsrikt.
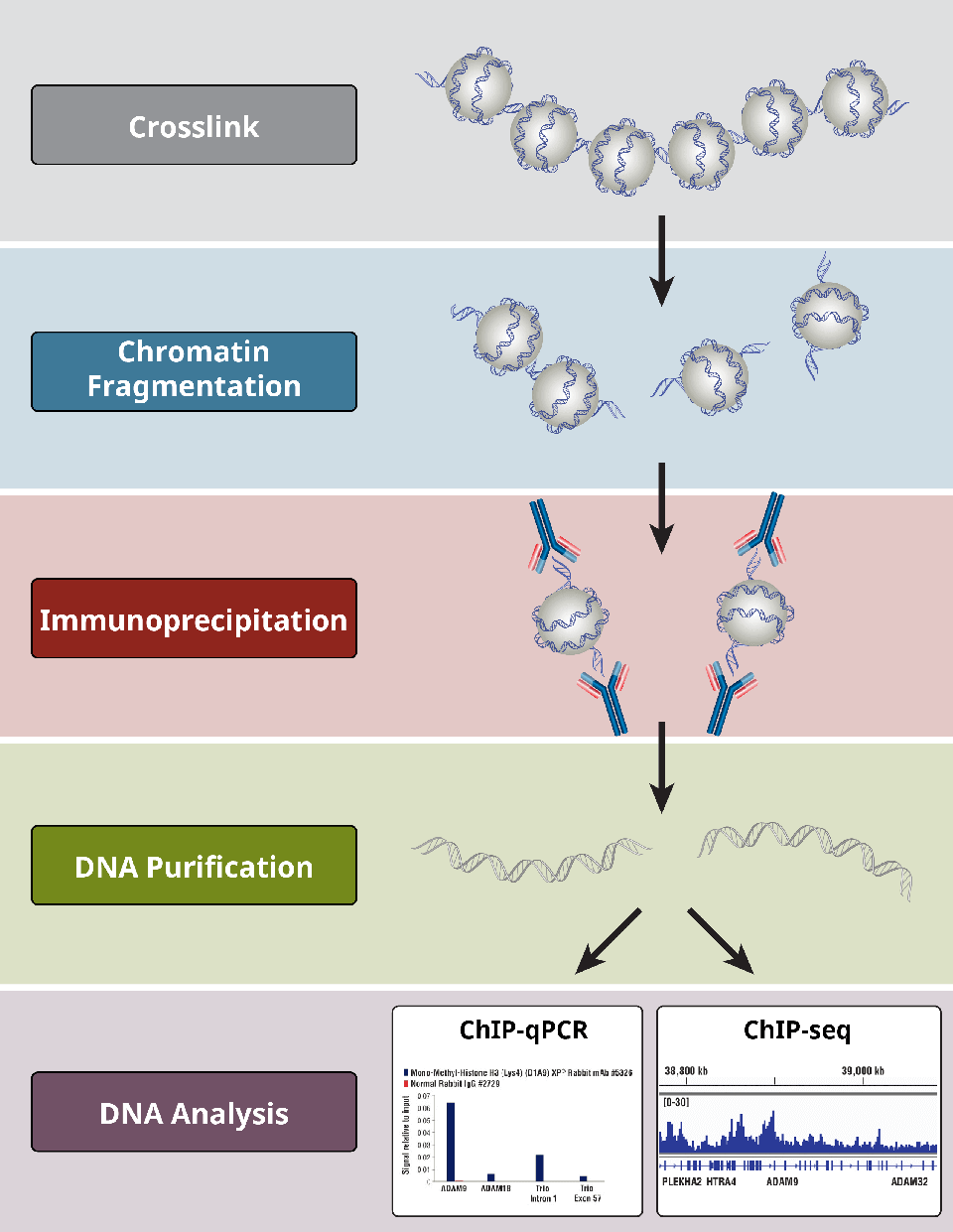
Översikt över de mest kritiska stegen i ett ChIP-protokoll.
Översikt över de mest kritiska stegen i ett ChIP-protokoll.
6.1 Hur korslänkar man celler och vävnader för ChIP?
Korslänkningsreagens används för att ”fixera” proteiner till det DNA de binder. Formaldehydbaserade reagenser används vanligtvis för att uppnå denna fixering. Celler och vävnader fixeras i allmänhet på ett liknande sätt, men vävnader kräver en längre fixeringstid och en snabbare fixeringsleverans för att snabbt tränga igenom målvävnaden innan den börjar degenerera.
Överfixering av kromatin kan minska effektiviteten av fragmentering genom sonikation, förutom att den hämmar antikropparnas bindning till sina proteinmål. Därför bör fixeringstiden bestämmas empiriskt för att möjliggöra maximal antikropp-antigenbindning samtidigt som man uppnår idealisk tvärbindning av proteiner till sitt mål-DNA.
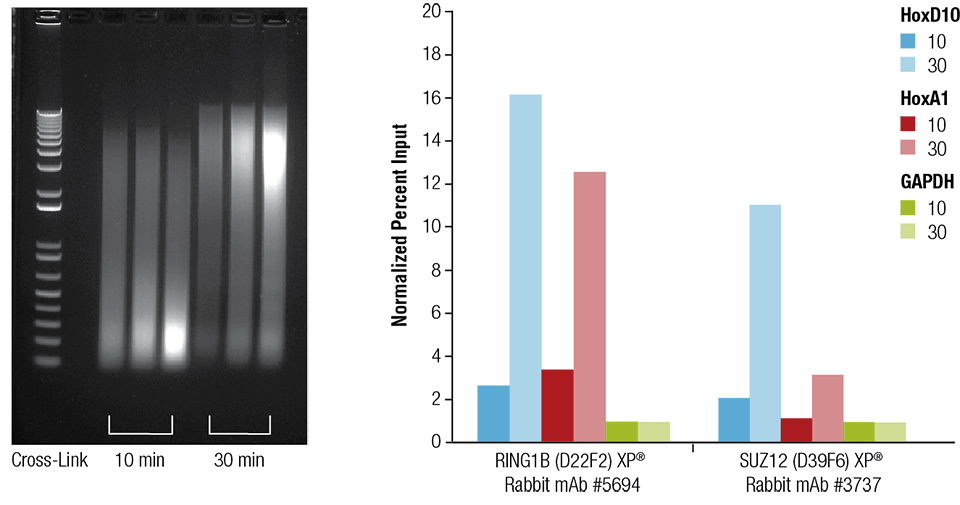
Mushjärta (H), hjärna (B) och lever (L) tvärbindades i 10 eller 30 minuter enligt uppgift (vänster panel). Kromatinet förbereddes och sonades i 4 minuter. ChIP utfördes med kromatin preparerat från hjärtvävnad med de angivna antikropparna med hjälp av SimpleChIP® Plus Sonication Chromatin IP Kit #56383 och det anrikade DNA:t kvantifierades genom realtids-PCR med hjälp av primers till de angivna generna (högra panelen). Mängden immunutfällt DNA i varje prov representeras som normaliserad signal till den negativa loci GAPDH, som är lika med 1.
Mushjärtan (H), hjärnan (B) och levern (L) tvärbindades i 10 eller 30 minuter enligt uppgift (vänster panel). Kromatinet förbereddes och sonades i 4 minuter. ChIP utfördes med kromatin preparerat från hjärtvävnad med de angivna antikropparna med hjälp av SimpleChIP® Plus Sonication Chromatin IP Kit #56383 och det anrikade DNA:t kvantifierades genom realtids-PCR med hjälp av primers till de angivna generna (högra panelen). Mängden immunutfällt DNA i varje prov representeras som normaliserad signal till den negativa loci GAPDH, som är lika med 1.
6.2 Hur fragmenterar man kromatin?
Kromatinfragmentering är avgörande för att ett ChIP-experiment ska lyckas. Fragmentering av kromatin är nödvändig för att kromatinet ska kunna lösas upp och möjliggöra dess samutfällning. Dessutom beror upplösningen av ChIP-analysen på fragmenteringen av kromatinet, eftersom DNA-fragmentens storlek bestämmer upplösningen av ChIP-analysen.
Enzymatisk nedbrytning använder mikrokoksnucleas (MNase), som klyver dubbelsträngat DNA mellan nukleosomerna för att generera kromatinfragment. Medan en fullständig MNase-desmältning genererar DNA-fragment på 150 baspar (mononukleosomer), genererar ofullständig digestion DNA-fragment mellan 150 och 750 baspar (mono-, di- och tri-nukleosomer). Sonikation använder mekanisk kraft för att fragmentera kromatin. Sonikation fragmenterar kromatin mellan och inom nukleosomer, vilket genererar en rad kromatinfragment mellan 150 och 1000 baspar.
För X-ChIP används antingen enzymatisk digestion eller sonikation för att skära kromatinet. Sonikationsförhållandena i sonikations-ChIP-protokollet bör bestämmas empiriskt, eftersom de skiljer sig åt beroende på celltyp och experimentellt tillstånd. Förfarandena för digestion är mer konsekventa mellan olika celltyper och vävnader, men kromatinfragmentens storlek bör ändå analyseras före IP.
För N-ChIP används nukleaser för att fragmentera kromatinet för att bibehålla proteinbindningen i ofixerade prover. Nukleasfragmenteringen bör också bestämmas empiriskt för att minimera överdigestion av kromatinet.
6.3 Varför använda enzymatisk digestion för ChIP?
Nukleasdigestion måste användas för N-ChIP eftersom proteinerna inte är tvärbundna till DNA:t och de hårda förhållanden som är förknippade med sonikationsbaserad fragmentering skulle resultera i dissociation av kromatinproteiner från DNA:t. N-ChIP är idealisk för att analysera histonprotein-DNA-interaktioner, eftersom histon-DNA-bindningen är mycket stark och stabil. N-ChIP fungerar dock inte bra för analys av kromatinbindning av transkriptionsfaktorer och kofaktorer.
Endera enzymatisk digestion eller sonikation kan användas för att fragmentera kromatin i X-ChIP. Fördelarna med enzymatisk digestion är bland annat fragmenteringens konsistens och milda fragmenteringsförhållanden (lägre värme och tvättmedel) som bättre bevarar integriteten hos kromatinet och antikroppsepitopen, vilket resulterar i ökad immunanrikning av transkriptionsfaktor- och kofaktorbundet kromatin.
6.4 Varför använder man sonikation för att fragmentera kromatin för ChIP?
Till skillnad från kromatinfragmenteringen som uppnås genom enzymatisk digestion förlitar sig sonikation på mekaniska krafter för att fragmentera kromatinet i mindre bitar. Den idealiska storleken på kromatinfragmenten för immunanrikning är mellan 200 och 1000 baspar. Sonikation är den traditionella metod som används för att fragmentera kromatin och kan utföras med hjälp av en traditionell sonikapparat eller mer avancerade sonikapparater med vattenbad som ger en mer fokuserad sonikation. Sonikation genererar verkligt randomiserade kromatinfragment, men kräver omfattande optimering i olika cellinjer och vävnader och är svår att reproducera från experiment till experiment. Kravet på höga detergentbuffertar och den värme som genereras under sonikation kan skada integriteten både av kromatinet och antikroppsepitoper på kromatinproteiner.
6.5 Optimering av kromatinsonikation för ChIP
Sonikationsbaserad kromatinfragmentering använder traditionellt höga detergentbuffertar och genererar värme, vilket båda kan skada integriteten av kromatinet och antikroppsepitoper. Därför måste mängden sonikation som används för att fragmentera kromatin fastställas experimentellt för olika cellinjer och vävnader. Man måste identifiera och använda den minimala mängd sonikation som krävs för att generera 150 till 1000 baspars DNA-fragment för att minimera skadorna på kromatinet.
För att påbörja en fullständig ChIP-analys med nedströmsanalys genom qPCR, DNA-chip eller NGS bör gelelektrofores användas för att analysera kromatinprover som sonikatiserats under olika tider. Fragmentstorleken beror på sonikationstiden-fragmentstorleken minskar när sonikationstiden ökar. Data tyder dock på att längre sonikationstider inte leder till bättre resultat. Att köra renat immunoprecipiterat DNA på en gel och bestämma den ideala fragmentstorleken är därför en enkel metod för att bestämma den minimala mängd sonikation som krävs för önskad DNA-storlek och för att undvika onödig skada på kromatin.

Enzymdigerat kromatin kördes på en agarosgel. Lane 1 visar kromatin som är underdigestrerat. Lane 2 visar korrekt digererat kromatin och Lane 3 visar kromatin som är överdigererat.
Enzymdigererat kromatin kördes på en agarosgel. Lane 1 visar kromatin som är underdigererat. Lane 2 visar korrekt digererat kromatin och Lane 3 visar kromatin som är överdigererat.
6.6 Hur väljer man en antikropp för ChIP?
Det är avgörande för att ett ChIP-experiment ska lyckas att välja en lämplig antikropp. Antikroppar som används i ett ChIP-experiment bör vara specifika för det aktuella proteinet och ha hög affinitet för antigenet. Det bästa valet av antikropp för ett ChIP- eller ChIP-seq-experiment är en ChIP- eller ChIP-seq-validerad antikropp. Om det inte finns någon ChIP-validerad antikropp tillgänglig för den aktuella genen, är det näst bästa valet en antikropp som har validerats i IP. Det är viktigt att notera att inte alla IP-validerade antikroppar fungerar för ChIP och inte alla ChIP-validerade antikroppar fungerar för ChIP-seq. Ju mer en antikropp är validerad i andra tillämpningar, t.ex. western, IP, IF, flow och IHC, desto större är förtroendet för antikroppens prestanda och specificitet. Valideringen av en antikropp måste följas av en empirisk bestämning av den optimala koncentrationen av antikroppen, tillsammans med IP-tvättförhållanden.
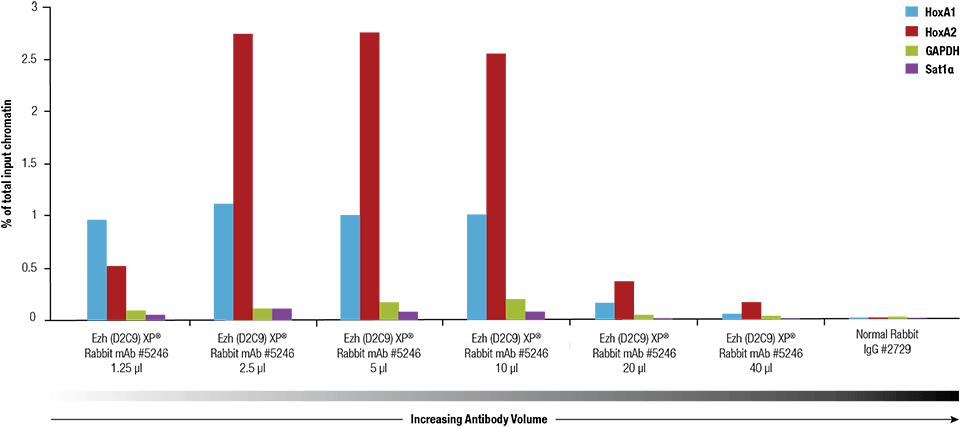
Ezh2 (D2C9) XP® Rabbit mAb #5246 titrerades med hjälp av SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 på tvärbundet kromatin som framställts från 4 x 106 NCCIT-celler.
Ezh2 (D2C9) XP® Rabbit mAb #5246 titrerades med hjälp av SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 på tvärbundet kromatin framställt från 4 x 106 NCCIT-celler.
6.7 Hur man utför immunutfällningen
Antikroppar används för att fånga upp ett protein av intresse och dess bundna DNA. Antikroppskoncentrationen bör bestämmas empiriskt, med en allmän utgångspunkt på 0,5-2,0 μg antikropp som används per 10 μg kromatin-DNA (motsvarar ungefär 4 x 106 celler). Strängheten i buffertar och tvätttider bör också bestämmas empiriskt eftersom de beror på antikroppens affinitet till målantigenet. Vanligtvis utförs antikroppskromatininkubationer i 2 timmar till över natten.
Antikropp-antigenkomplex (+DNA) fångas med affinitet på ett antikroppsbindande harts. I ChIP-experiment består detta harts vanligtvis av magnetiska, sefaros- eller agarospärlor av ChIP-kvalitet som är konjugerade till protein A och/eller protein B. Antikroppar binder protein A- och/eller protein G-pärlor med olika affinitet beroende på den art i vilken de utvecklades och IgG-subtypen i deras tunga kedja. Pärlorna inkuberas vanligen med antikropp:kromatin i 2-4 timmar.
Vasksteg behövs för att avlägsna kromatin som inte är knutet till antikroppar, följt av omvändning av tvärbindningar (för X-ChIP) och rening av DNA. Dessutom måste en IgG-kontroll-IP utföras för att bestämma bakgrunden (signal/brus). Positiva kontrollantikroppar (t.ex. totalt histon H3) och/eller positiva qPCR-kontrollprimer (för kända positiva och negativa målproteinbindande loci) måste också inkluderas för att fastställa ospecifik bindning. För optimala resultat bör QC av kromatin-IP genom qPCR utföras före NGS-analys nedströms.
6.8 Hur man eluerar kromatinet från protein A/G-pärlorna
Kromatinet elueras från protein A/G-pärlorna med hjälp av detergent och värme. ”Vortexing” eller blandning med låg hastighet krävs för att hålla pärlorna i suspension och öka elueringen av kromatinet.
6.9 Hur man reverserar kromatinets tvärbindningar
Trödbindningarna reverseras med hjälp av hög värme och hög salthalt (båda dessa är vitala komponenter). Proteinas K tillsätts också för att smälta de associerade kromatinproteinerna och tillsätts antikroppar, vilket möjliggör effektivare DNA-rening nedströms.
6.10 Hur man renar DNA
När kromatinets tvärbindningar avlägsnats renas DNA antingen med hjälp av klassiska metoder med fenolklorform följt av etanolutfällning eller med hjälp av kolonnbaserade DNA-reningsutrustningar.
Hur man analyserar anrikat DNA
När DNA har renats kan flera nedströmsanalyser utföras, inklusive ChIP-PCR, ChIP-qPCR, ChIP-chip och ChIP-seq.
7.1 ChIP-PCR- och ChIP-qPCR-analys
ChIP-PCR- och ChIP-qPCR-analyser är bäst för analys av enstaka gener och kan användas för att amplifiera och kvantifiera specifika DNA-fragment på ett snabbt och kostnadseffektivt sätt.
7.2 ChIP-chip-analys
ChIP-chip-analys använder tiling DNA-mikroarray-chips för att skapa en genomomfattande, högupplöst karta över proteinbindning och proteinmodifiering.
7.3 ChIP-seq-analys
ChIP-seq-analys använder standard NGS-teknik för att anpassa renat DNA till tidigare annoterade helgenom för att identifiera proteinbindningsprofiler som omfattar hela genomet.