Wat is ChIP?
Chromatine-immunoprecipitatie, of ChIP, is een op antilichamen gebaseerde technologie die wordt gebruikt om selectief specifieke DNA-bindende eiwitten te verrijken samen met hun DNA-doelen. ChIP wordt gebruikt om een bepaalde eiwit-DNA interactie, verschillende eiwit-DNA interacties, of interacties in het hele genoom of een subset van genen te onderzoeken.
ChIP maakt gebruik van antilichamen die selectief eiwitten herkennen en binden, met inbegrip van histonen, histonmodificaties, transcriptiefactoren en cofactoren, om informatie te verstrekken over chromatine toestanden en gentranscriptie. Een combinatie van proteoomanalyse en moleculair-biologische technieken die bij ChIP worden gebruikt, maakt het mogelijk de genexpressie en -regulatie in interessante cellen of weefsels te begrijpen.
Wanneer wordt ChIP gebruikt?
Typisch wordt ChIP gebruikt om de relatieve overvloed van een specifiek eiwit of een specifieke eiwitmodificatie op een bepaalde plaats in het genoom te identificeren. ChIP kan worden gebruikt voor het beantwoorden van een groot aantal wetenschappelijke vragen over de interactie tussen eiwitten en chromatine. ChIP kan bijvoorbeeld worden gebruikt om de aanwezigheid van bepaalde eiwitten op verschillende loci te vergelijken, de verschillende eiwitten over een genomische regio van belang in kaart te brengen, of de eiwitbinding aan een induceerbaar gen in reactie op een stimulus in de tijd te kwantificeren.
Hoe werkt ChIP?
Het principe achter ChIP is betrekkelijk eenvoudig en berust op het gebruik van een antilichaam om een bepaald eiwit, histon, transcriptiefactor of cofactor en het daaraan gebonden chromatine te isoleren, of neer te slaan, uit een eiwitmengsel dat uit cellen of weefsels werd geëxtraheerd. Vandaar de naam van de techniek: Chromatine-immunoprecipitatie. In ChIP-PCR of ChIP-seq, kunnen immuun-verrijkte DNA fragmenten vervolgens worden geïdentificeerd en gekwantificeerd met behulp van algemeen beschikbare PCR of qPCR reagentia en Next Generation Sequencing (NGS) technologieën.
Wat is natieve ChIP (N-ChIP) vs crosslinked ChIP (X-ChIP)?
Er zijn 2 soorten ChIP technieken die kunnen worden uitgevoerd, afhankelijk van de experimentele vraag en het uitgangsmateriaal voor het experiment: 1) natieve ChIP (N-ChIP) en 2) crosslinked ChIP (X-ChIP). Beide soorten ChIP hebben voor- en nadelen:
- In N-ChIP wordt geen fixeermiddel gebruikt om eiwitten aan het chromatine te crosslinken. In plaats daarvan wordt natief chromatine geïsoleerd uit celkernen die zijn verteerd met een nuclease. Omdat antilichamen worden opgewekt tegen niet-gefixeerde antigenen, biedt N-ChIP het voordeel van betere herkenning en binding van antilichamen aan hun doelantigenen. PCR is wellicht niet nodig voor downstream-analyse vanwege de hoge abundantie van histon-eiwitten. Hoewel deze voordelen N-ChIP tot een aantrekkelijke methode maken, kan deze alleen worden gebruikt voor de detectie van histonen. Bovendien kan het verlies van eiwitbinding tijdens de chromatinedigestie en immunoprecipitatiestappen de gegevens vertekenen of een goede analyse in de weg staan.
- In X-ChIP worden chemische fixeermiddelen zoals formaldehyde gebruikt om het belanghebbende eiwit aan het DNA te crosslinken en wordt fragmentatie van chromatine bereikt door sonicatie of nuclease-digestie. Het voordeel van X-ChIP is dat het kan worden gebruikt met histon- en niet-histon-eiwitten en dat er over het algemeen minder cellulair uitgangsmateriaal voor nodig is dan voor N-ChIP. X-ChIP minimaliseert ook de kans op verlies van chromatine-eiwitten tijdens de extractie, waardoor de detectie van voorbijgaande eiwitinteracties mogelijk wordt. De precipitatiestap is echter minder efficiënt en DNA-amplificatie door PCR is noodzakelijk voor downstream analyses.
Wat zijn de verschillende soorten ChIP-assays?
Zodra de chromatine-immunoprecipitatie zelf is voltooid, kunnen verschillende downstream analyses worden uitgevoerd op het gezuiverde chromatine en de bijbehorende eiwitten, histonen, transcriptiefactoren, en cofactoren. De meest gebruikelijke methoden voor analyse van één gen en analyse van het gehele genoom zijn respectievelijk qPCR en ChIP-seq. PCR en ChIP-chip zijn ook opties voor downstream analyse.
5.1 Wat zijn de voordelen van ChIP-PCR?
ChIP-PCR wordt uitgevoerd om histon-modificaties en/of eiwitbinding aan een bekende subset van doelloci in het genoom te analyseren. Bij ChIP-PCR worden met immuun verrijkte DNA-fragmenten geïdentificeerd en gekwantificeerd met behulp van algemeen beschikbare PCR- of qPCR-reagentia en -technologieën. Met ChIP-qPCR kunnen snelle en kwantitatieve vergelijkingen van specifieke regio’s binnen het genoom over meerdere monsters worden gemaakt. Dit is goedkoper en tijdsefficiënter dan whole genome sequencing methoden.
5.2 Wat zijn de voordelen van ChIP-chip?
ChIP-chip technologie verwijst naar het gebruik van een DNA microarray chip voor het analyseren van ChIP-immuun verrijkte DNA fragmenten. Het gebruik van genoom-tiling microarray-technologie maakt een genoomanalyse mogelijk van eiwitten die aan geïsoleerd DNA zijn gebonden en genereert een genomische kaart met hoge resolutie van eiwitbinding en eiwitmodificaties. ChIP-chip heeft talrijke toepassingen, zowel in fundamenteel onderzoek als in onderzoek naar ziekten. Het kan bijvoorbeeld worden gebruikt om de bindingsplaatsen van transcriptiefactoren, enhancers en repressors te identificeren en om deze soorten gebonden eiwitten in controlemonsters en pathologische monsters te vergelijken. Echter, sinds de kosten van NGS aanzienlijk zijn gedaald en vergelijkbare resultaten worden verkregen met behulp van ChIP-seq, kiezen meer mensen ervoor om ChIP-seq uit te voeren in plaats van ChIP-chip.
5.3 Wat zijn de voordelen van ChIP-seq?
Gelijk aan ChIP-chip, geeft ChIP-seq informatie over genoom-brede eiwitbinding. Echter, in tegenstelling tot ChIP-chip, ChIP-seq maakt gebruik van NGS-technologie om DNA-fragmenten te identificeren en in kaart te brengen tegen het volledige genoom.
Meer hedendaagse DNA amplificatie technologie maakt het mogelijk robuuste analyse worden uitgevoerd in een kwestie van dagen met een lage hoeveelheid input DNA. Wanneer uitgangsmateriaal schaars is, hebben deze technologische verbeteringen in bibliotheekvoorbereidingsmethoden ChIP-seq-experimenten mogelijk gemaakt.
Bovendien maakt nieuwe technologie waarbij DNA-monsters uniek worden gelabeld met korte sequenties, bekend als streepjescodes, het nu mogelijk individuele fragmenten te bundelen in een enkele sequencingstraat voor multiplexed analyse. Dit heeft de efficiëntie aanzienlijk verhoogd en de kosten van DNA-sequencing-experimenten verlaagd, wat ChIP-seq-toepassingen verder ondersteunt.
Al met al is het voordeel van ChIP-seq, dankzij de vooruitgang in DNA-sequencingtechnologie, dat een groot aantal met ChIP verrijkte DNA-monsters goedkoop kunnen worden gesequeneerd in een relatief korte tijd met een hogere gevoeligheid en nauwkeurigheid dan ChIP-chip.
Wat zijn de verschillende stappen in de ChIP-assay?
ChIP-assays volgen een algemeen protocol:
- Crosslinking van eiwitten aan DNA, alleen voor X-ChIP
- Cellolyse
- Chromatinefragmentatie door digestie (voor X-ChIP en N-ChIP) of door afschuiving met sonicatiegebaseerd afschuiven (alleen voor X-ChIP)
- Immunoprecipitatie met behulp van specifieke antilichamen
- DNA-opschoning voor downstream-analyses
- DNA-analyse via PCR, qPCR, microarray, of NGS
Belangrijk is dat positieve en negatieve controles bij elke stap integraal zijn om te bepalen of een ChIP-experiment succesvol is geweest.
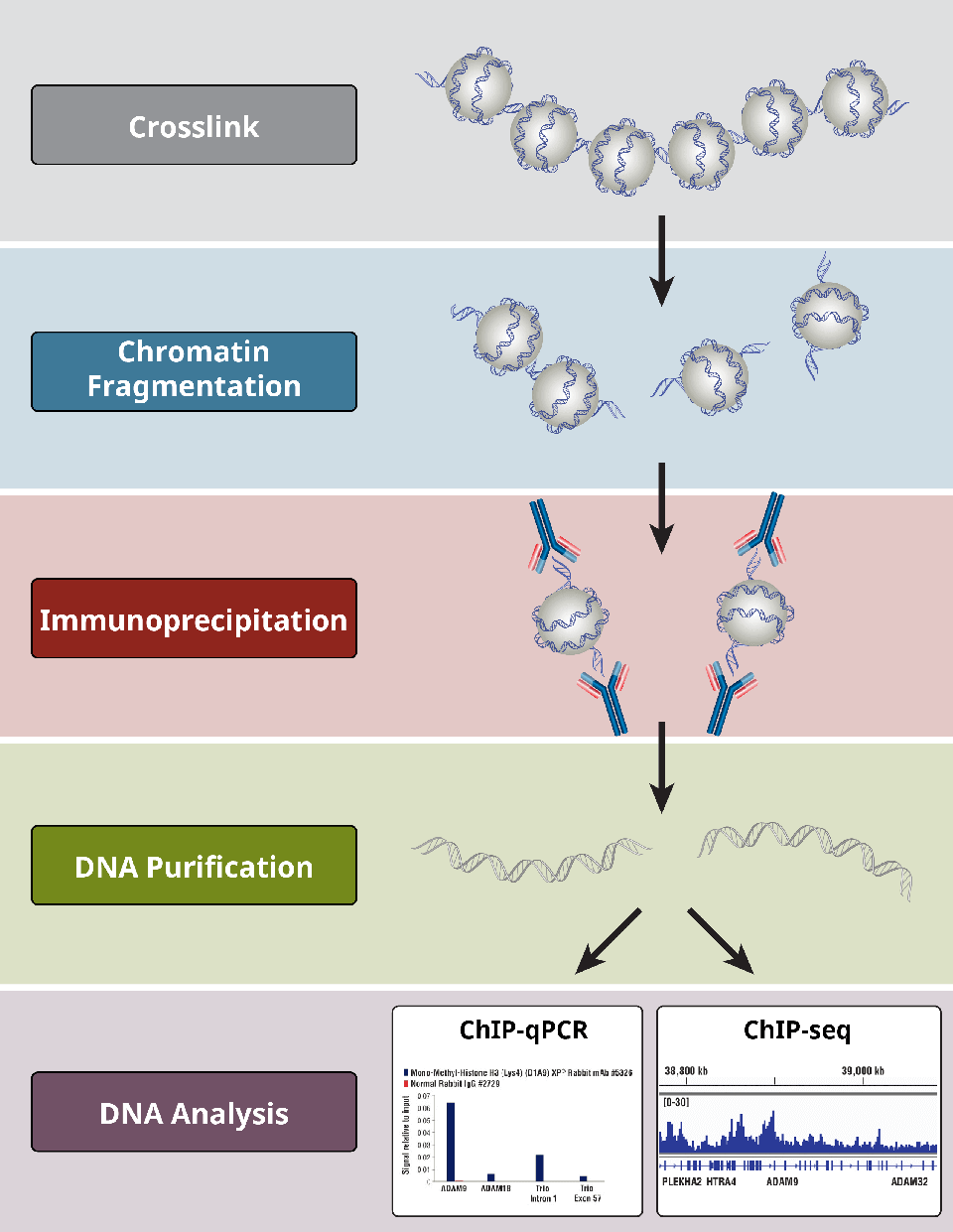
Overzicht van de meest kritische stappen van een ChIP-protocol.
Overzicht van de meest kritische stappen van een ChIP-protocol.
6.1 Hoe crosslink je cellen en weefsels voor ChIP?
Crosslinking-reagentia worden gebruikt om eiwitten te “fixeren” aan het DNA dat ze binden. Meestal worden reagentia op basis van formaldehyde gebruikt om deze fixatie te bereiken. Cellen en weefsels worden over het algemeen op soortgelijke wijze gefixeerd, maar weefsel vereist een langere fixatietijd en een snellere fixatietoediening om snel in het doelweefsel door te dringen voordat het begint te degenereren.
Overfixatie van chromatine kan de efficiëntie van fragmentatie door sonicatie verminderen, naast het remmen van de binding van antilichamen aan hun eiwitdoelen. Daarom moet fixatie tijd empirisch worden bepaald om voor maximale antilichaam-antigeen binding mogelijk te maken, terwijl het bereiken van ideale verknoping van eiwitten aan hun doel-DNA.
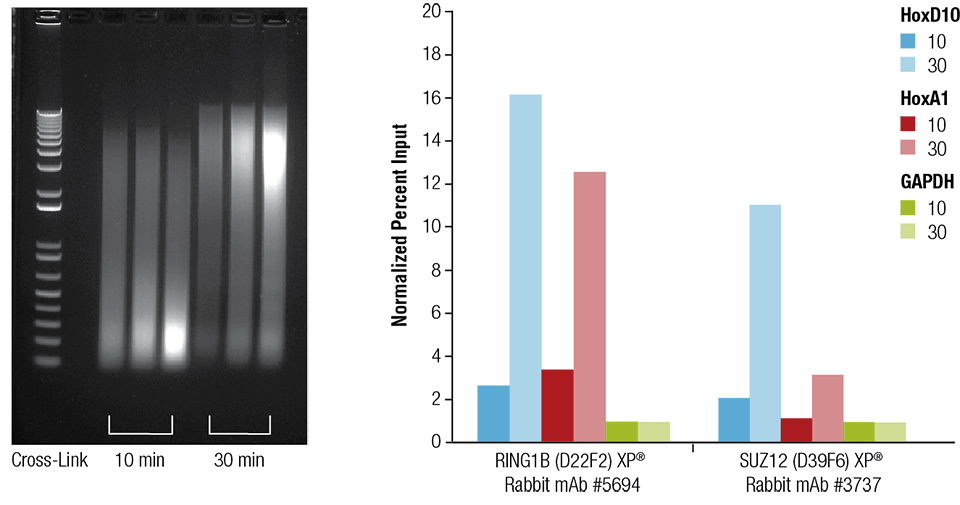
Muis hart (H), hersenen (B), en lever (L) werden verknoopt gedurende 10 of 30 minuten zoals aangegeven (linker paneel). Het chromatine werd geprepareerd en gedurende 4 minuten gesonificeerd. ChIP werd uitgevoerd met chromatine geprepareerd uit hartweefsel met de aangegeven antilichamen met behulp van de SimpleChIP® Plus Sonication Chromatin IP Kit #56383 en het verrijkte DNA werd gekwantificeerd door real-time PCR met primers voor de aangegeven genen (rechter paneel). De hoeveelheid geïmmunoprecipiteerd DNA in elk monster wordt weergegeven als genormaliseerd signaal ten opzichte van de negatieve loci GAPDH, die gelijk is aan 1.
Muis hart (H), hersenen (B), en lever (L) werden crosslinked gedurende 10 of 30 minuten, zoals aangegeven (linker paneel). Het chromatine werd geprepareerd en gedurende 4 minuten gesonificeerd. ChIP werd uitgevoerd met chromatine geprepareerd uit hartweefsel met de aangegeven antilichamen met behulp van de SimpleChIP® Plus Sonication Chromatin IP Kit #56383 en het verrijkte DNA werd gekwantificeerd door real-time PCR met primers voor de aangegeven genen (rechter paneel). De hoeveelheid geïmmunoprecipiteerd DNA in elk monster wordt weergegeven als genormaliseerd signaal ten opzichte van de negatieve loci GAPDH, dat gelijk is aan 1.
6.2 Hoe fragmenteer je chromatine?
Chromatinefragmentatie is essentieel voor het succes van een ChIP-experiment. Fragmentatie van chromatine is noodzakelijk om het chromatine te solubiliseren en maakt coprecipitatie ervan mogelijk. Bovendien is de resolutie van de ChIP-assay afhankelijk van de fragmentatie van het chromatine, aangezien de grootte van het DNA-fragment de resolutie van de ChIP-assay bepaalt.
Enzymatische digestie maakt gebruik van micrococcal nuclease (MNase), dat dubbelstrengs DNA tussen nucleosomen klieft om chromatinefragmenten te genereren. Terwijl een volledige MNase-ontsluiting DNA-fragmenten van 150 basenparen (mono-nucleosomen) genereert, genereert een onvolledige ontsluiting DNA-fragmenten tussen 150 en 750 basenparen (mono-, di-, en tri-nucleosomen). Sonificatie gebruikt mechanische kracht om chromatine te fragmenteren. Sonificatie fragmenteert chromatine tussen en binnen nucleosomen, waarbij een reeks chromatinefragmenten tussen 150 en 1000 basenparen wordt gegenereerd.
Voor X-ChIP wordt hetzij enzymatische digestie hetzij sonicatie gebruikt om het chromatine af te breken. Sonificatie voorwaarden in de sonicatie ChIP-protocol moet empirisch worden bepaald, omdat ze verschillen per celtype en experimentele conditie. Digestie voorwaarden zijn meer consistent over verschillende celtypes en weefsels, maar chromatine fragmentgrootte moet nog worden geanalyseerd voorafgaand aan IP.
Voor N-ChIP, worden nucleasen gebruikt om het chromatine fragmenteren om eiwitbinding te handhaven in niet-vaste monsters. Nuclease fragmentatie moet ook empirisch worden bepaald om overdigestie van het chromatine te minimaliseren.
6.3 Waarom enzymatische digestie gebruiken voor ChIP?
Nuclease digestie moet worden gebruikt voor N-ChIP omdat eiwitten niet crosslinked aan het DNA en de barre omstandigheden geassocieerd met sonicatie-gebaseerde fragmentatie zou resulteren in dissociatie van chromatine-eiwitten van het DNA. N-ChIP is ideaal voor het analyseren van histon-eiwit-DNA-interacties, omdat de histon-DNA-binding zeer sterk en stabiel is. N-ChIP werkt echter niet goed voor de analyse van chromatinebinding door transcriptiefactoren en cofactoren.
Ofwel enzymatische digestie, ofwel sonicatie kan worden gebruikt om chromatine te fragmenteren in X-ChIP. De voordelen van enzymatische digestie zijn de consistentie van fragmentatie en milde fragmentatie omstandigheden (minder warmte en detergent) die beter behouden van de integriteit van het chromatine en antilichaam epitopen, wat resulteert in een verhoogde immuun-verrijking van transcriptiefactor en cofactor gebonden chromatine.
6.4 Waarom sonicatie gebruiken om chromatine te fragmenteren voor ChIP?
In tegenstelling tot de chromatine fragmentatie bereikt door enzymatische digestie, sonicatie vertrouwt op mechanische krachten om chromatine fragmenteren in kleinere stukjes. De ideale grootte van chromatinefragmenten voor immuunverrijking ligt tussen 200 en 1000 basenparen. Sonificatie is de traditionele methode die wordt gebruikt om chromatine te fragmenteren en kan worden uitgevoerd met een traditionele sonde-sonicator of met meer geavanceerde waterbad-sonicators die meer gerichte sonicatie bieden. Sonificatie genereert echt willekeurig fragmenten van chromatine, maar het vereist uitgebreide optimalisatie in verschillende cellijnen en weefsels en is moeilijk te reproduceren van experiment tot experiment. De eis voor hoge detergenten buffers en de warmte die wordt opgewekt tijdens sonicatie kan schade toebrengen aan de integriteit van zowel de chromatine en antilichaam epitopen op chromatine proteins.
6.5 Het optimaliseren van chromatine sonicatie voor ChIP
Sonication-based chromatine fragmentaton traditioneel gebruik maakt van hoge detergenten buffers en genereert warmte, die beide kan schade toebrengen aan de integriteit van het chromatine en antilichaam epitopen. Daarom moet de hoeveelheid sonicatie die wordt gebruikt om chromatine te fragmenteren experimenteel worden bepaald voor verschillende cellijnen en weefsels. Men moet identificeren en gebruiken de minimale hoeveelheid sonicatie nodig is om 150 tot 1000 basepaar DNA-fragmenten genereren om schade aan het chromatine minimaliseren.
Voordat men begint aan een volledige ChIP assay met downstream-analyse door qPCR, DNA-chip, of NGS, moet gelelektroforese worden gebruikt om chromatine monsters gesoniceerd voor verschillende tijden te analyseren. Fragmentgrootte is afhankelijk van de sonicatietijd-fragmentgrootte neemt af naarmate de sonicatietijd toeneemt. Uit de gegevens blijkt echter dat langere sonicatietijden niet tot betere resultaten leiden. Daarom is het uitvoeren van gezuiverd immuungeprecipiteerd DNA op een gel en het bepalen van de ideale fragmentgrootte een eenvoudige methode om de minimale hoeveelheid sonicatie te bepalen die nodig is voor de gewenste DNA-grootte en om onnodige schade aan chromatine te voorkomen.

Enzymatisch gedigesteerd chromatine werd uitgevoerd op een agarosegel. Rij 1 toont chromatine dat onvoldoende is verteerd. Rij 2 toont chromatine dat goed is verteerd, en rij 3 toont chromatine dat te ver is verteerd.
Enzymatisch verteerd chromatine is op een agarosegel gerund. Rij 1 toont chromatine dat niet volledig is verteerd. Rij 2 toont chromatine dat goed is verteerd, en rij 3 toont chromatine dat te ver is verteerd.
6.6 Hoe kies je een antilichaam voor ChIP?
Het kiezen van een geschikt antilichaam voor een ChIP-experiment is essentieel voor het succes ervan. Antilichamen die bij een ChIP-experiment worden gebruikt, moeten specifiek zijn voor het belangwekkende eiwit en een hoge affiniteit hebben voor het antigeen. De beste keuze van antilichaam voor een ChIP- of ChIP-seq-experiment is een ChIP- of ChIP-seq-gevalideerd antilichaam. Als er geen voor ChIP gevalideerd antilichaam beschikbaar is voor het gen van interesse, is de volgende beste keuze een antilichaam dat in IP gevalideerd is. Het is belangrijk op te merken dat niet alle IP-gevalideerde antilichamen werken bij ChIP en dat niet alle ChIP-gevalideerde antilichamen werken bij ChIP-seq. Bovendien geldt dat hoe meer een antilichaam gevalideerd is voor andere toepassingen, zoals western, IP, IF, flow en IHC, hoe meer vertrouwen men kan hebben in de prestaties en specificiteit van het antilichaam. De validatie van een antilichaam moet worden gevolgd door empirische bepaling van de optimale concentratie van het antilichaam, samen met IP wascondities.
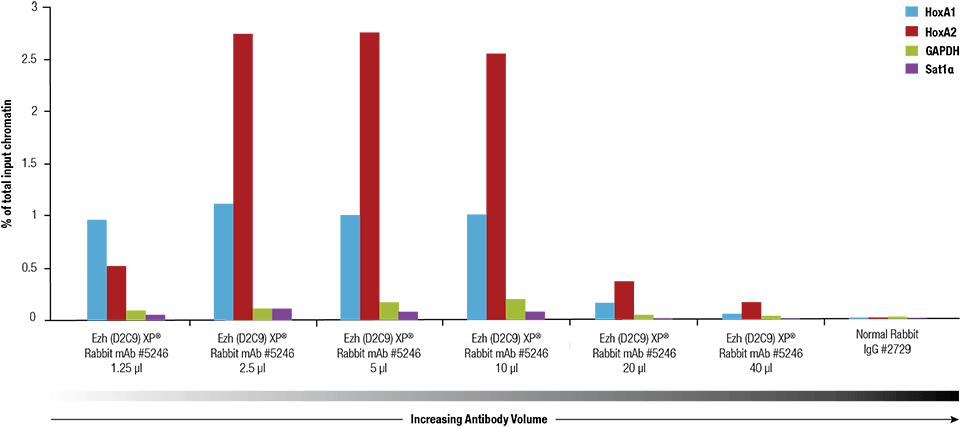
Ezh2 (D2C9) XP® Rabbit mAb #5246 werd getitreerd met behulp van de SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 op crosslinked chromatine bereid uit 4 x 106 NCCIT-cellen.
Ezh2 (D2C9) XP® Rabbit mAb #5246 werd getitreerd met behulp van de SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 op crosslinked chromatine bereid uit 4 x 106 NCCIT-cellen.
6.7 Hoe de immunoprecipitatie uit te voeren
Antilichamen worden gebruikt om een interessant eiwit en het daaraan gebonden DNA te vangen. De antilichaamconcentratie moet empirisch worden bepaald, met een algemeen uitgangspunt van 0,5-2,0 ug antilichaam gebruikt per 10 ug chromatine DNA (komt overeen met ongeveer 4 x 106 cellen). De striktheid van buffers en wastijden moeten ook empirisch worden bepaald omdat zij afhankelijk zijn van de affiniteit van het antilichaam op zijn doelantigeen. Gewoonlijk worden antilichaam-chromatine-incubaties gedurende 2 uur tot een nacht uitgevoerd.
Antilichaam-antigeen(+DNA)-complexen worden affiniteit-gevangen op een antilichaam-bindende hars. Bij ChIP-experimenten bestaat deze hars gewoonlijk uit magnetische, sepharose- of agarosekorrels van ChIP-kwaliteit, die zijn geconjugeerd met eiwit A en/of eiwit B. Antilichamen binden eiwit A en/of eiwit G-korrels met uiteenlopende affiniteit, afhankelijk van de diersoort waarin zij zijn ontwikkeld en het IgG-subtype van hun zware keten. De korrels worden gewoonlijk gedurende 2 tot 4 uur geïncubeerd met antilichaam:chromatine.
Wasstappen zijn nodig om niet-antilichaamgebonden chromatine te verwijderen, gevolgd door omkering van crosslinks (voor X-ChIP) en zuivering van DNA. Bovendien moet een IgG-controle-IP worden uitgevoerd om de achtergrond (signaal:ruis) te bepalen. Positieve controleantilichamen (d.w.z. totaal histon H3) en/of positieve controle qPCR-primers (voor bekende positieve en negatieve doeleiwitbindingsloci) moeten ook worden opgenomen om niet-specifieke binding te bepalen. Voor optimale resultaten moet QC van de chromatine-IP door qPCR voorafgaand aan downstream NGS-analyse worden uitgevoerd.
6.8 Hoe de chromatine van de protein A/G-korrels te elueren
Chromatine wordt van de protein A/G-korrels geëlueerd met behulp van detergenten en warmte. Met lage snelheid “vortexen” of mengen is nodig om de korrels in suspensie te houden en de elutie van het chromatine te bevorderen.
6.9 Hoe de chromatine-crosslinks worden omgekeerd
Crosslinks worden omgekeerd door hoge hitte en hoog zout (dit zijn beide vitale componenten). Proteinase K wordt ook toegevoegd om de geassocieerde chromatine-eiwitten en toegevoegde antilichamen te verteren, waardoor een efficiëntere downstream DNA-zuivering mogelijk wordt.
6.10 Hoe DNA te zuiveren
Nadat de chromatine crosslinks zijn verwijderd, wordt DNA gezuiverd met behulp van klassieke fenol-chloroform gevolgd door ethanol-precipitatiemethoden of met behulp van kolom-gebaseerde DNA-zuiveringskits.
Hoe verrijkt DNA te analyseren
Zodra DNA gezuiverd is, kunnen verschillende downstream analyses worden uitgevoerd, waaronder ChIP-PCR, ChIP-qPCR, ChIP-chip, en ChIP-seq.
7.1 ChIP-PCR en ChIP-qPCR analyse
ChIP-PCR en ChIP-qPCR analyses zijn het beste voor single-gen analyse en kunnen worden gebruikt om specifieke fragmenten van DNA te amplificeren en te kwantificeren op een snelle en kosteneffectieve manier.
7.2 ChIP-chip analyse
ChIP-chip analyse maakt gebruik van tiling DNA microarray chips om een genoom-brede, hoge resolutie kaart van eiwitbinding en eiwitmodificatie te creëren.
7.3 ChIP-seq analyse
ChIP-seq analyse maakt gebruik van standaard NGS-technologie om gezuiverd DNA uit te lijnen met eerder geannoteerde volledige genomen om genoom-brede eiwitbindingsprofielen te identificeren.