ChIPとは
クロマチン免疫沈降法(ChIP)は、特定のDNA結合タンパク質とそのDNAターゲットを選択的に濃縮するために用いられる抗体ベースの技術であります。
ChIPは、ヒストン、ヒストン修飾、転写因子、補因子などのタンパク質を選択的に認識・結合する抗体を利用し、クロマチン状態や遺伝子転写に関する情報を提供するために使用されます。
ChIPはどのような場合に使用されますか?
一般的に、ChIPはゲノムの特定の領域における特定のタンパク質または特定のタンパク質修飾の相対量を特定するために使用されます。 ChIPは、タンパク質とクロマチンの相互作用に関する多くの科学的な疑問に答えるために使用することができます。 たとえば、さまざまな遺伝子座における特定のタンパク質の存在を比較したり、関心のあるゲノム領域全体にさまざまなタンパク質をマッピングしたり、刺激に応答する誘導性遺伝子へのタンパク質結合を経時的に定量したりするために、ChIPを使用することが可能です。
ChIPの原理は比較的単純で、細胞や組織から抽出したタンパク質混合物から、特定のタンパク質、ヒストン、転写因子、補因子とその結合したクロマチンを分離、または沈殿させるために抗体を使用することに依存します。 そのため、この手法の名前がついた。 クロマチン免疫沈降法(Chromatin Immunoprecipitation)。
Native ChIP (N-ChIP) と Crosslinked ChIP (X-ChIP) とは?
ChIP技術には、実験課題と実験の出発材料に応じて、2種類の手法が存在します。 1) native ChIP (N-ChIP) と2) crosslinked ChIP (X-ChIP)です。 どちらのタイプのChIPにも利点と欠点があります。
- N-ChIPでは、タンパク質とクロマチンを架橋するための固定剤を使用しません。 その代わりに、ヌクレアーゼで消化された細胞核からネイティブなクロマチンが単離されます。 固定化されていない抗原に対して抗体を作成するため、抗体の標的抗原に対する認識・結合が良好であるという利点がある。 また、ヒストン蛋白質が豊富に含まれているため、下流の解析にPCRを必要としない場合もある。 これらの利点からN-ChIPは魅力的な方法であるが、ヒストンの検出にしか使用できない。 また、クロマチン消化や免疫沈降の過程でタンパク質の結合が失われると、データに偏りが生じたり、適切な解析ができなくなったりする。
- X-ChIPでは、ホルムアルデヒドなどの化学固定剤を用いて目的のタンパク質をDNAに架橋し、超音波処理またはヌクレアーゼ消化によってクロマチンを断片化させる。 X-ChIPの利点は、ヒストンおよび非ヒストンタンパク質に使用できること、一般にN-ChIPよりも細胞由来の出発物質が少なくてすむことである。 また、X-ChIPは抽出中にクロマチンタンパク質が失われる可能性を最小限に抑え、一過性のタンパク質相互作用の検出を可能にする。
ChIPアッセイの種類は?
クロマチン免疫沈降が完了したら、精製したクロマチンや関連タンパク質、ヒストン、転写因子、コファクターについていくつかのダウンストリーム解析を行うことができる。 単一遺伝子解析ではqPCR、全ゲノム解析ではChIP-seqが最も一般的な方法である。 また、PCRやChIP-chipも下流の解析の選択肢となります。
5.1 ChIP-PCRの利点は?
ChIP-PCRは、ゲノム中のターゲット遺伝子座の既知のサブセットに対するヒストン修飾および/またはタンパク質結合を解析するために実施されます。 ChIP-PCRでは、免疫強化されたDNA断片が、広く利用されているPCRまたはqPCR試薬および技術を使用して同定および定量化されます。 ChIP-qPCRを用いることで、複数のサンプルにまたがるゲノム内の特定領域の迅速かつ定量的な比較が可能となります。
5.2 ChIP-chipの利点は?
ChIP-chip技術とは、ChIP-免疫強化DNA断片を分析するためにDNAマイクロアレイチップを利用することを指します。 ゲノムタイリングマイクロアレイ技術を用いることで、単離されたDNAに結合するタンパク質の全ゲノム解析が可能となり、タンパク質結合やタンパク質修飾の高解像度ゲノムマップを作成することができます。 ChIP-chipは、基礎研究だけでなく、疾患を対象とした研究にも多面的に利用されています。 例えば、転写因子、エンハンサー、リプレッサーの結合部位の同定や、対照試料と病的試料におけるこの種の結合タンパク質の比較に用いることができる。 しかし、NGSのコストが大幅に低下し、ChIP-seqを使用して同様の結果が得られるため、ChIP-chipの代わりにChIP-seqを行うことを選択する人が増えています。
5.3 ChIP-seqの利点は何か
ChIP-chipと同様に、ChIP-seqではゲノム全体のタンパク質結合について情報を得ることができます。 しかし、ChIP-chipとは異なり、ChIP-seqはNGS技術を使用してDNA断片を特定し、ゲノム全体に対してマッピングします。
より現代的なDNA増幅技術により、少量の入力DNAで数日のうちに堅牢な分析を実施することが可能です。 出発材料が乏しい場合、ライブラリー調製法におけるこれらの技術の進歩により、ChIP-seq実験が可能になりました。
さらに、DNAサンプルにバーコードとして知られる短い配列で一意にタグ付けする新技術により、個々のフラグメントを単一のシーケンスレーンにプールして多重化分析することができるようになりました。
DNAシーケンサー技術の進歩により、ChIP-seqの利点は、大量のChIP富化DNAサンプルを比較的短時間で安価に、ChIP-chipよりも高い感度と精度でシーケンスできることです。
ChIPアッセイは一般的なプロトコルに沿って行われます。
- X-ChIPのみ、タンパク質をDNAに架橋する
- 細胞溶解
- 消化(X-ChIPおよびN-ChIP)または超音波処理によるクロマチン断片化
- 細胞溶解
- 細胞溶解
- クロマチン断片化(X-ChIPおよびN-ChIP)
- 特異的抗体を用いた免疫沈降
- DNA clean-up for downstream analyses
- DNA analysis via PCR, qPCR、マイクロアレイ、またはNGS
重要なのは、各ステップでの陽性および陰性コントロールが、ChIP実験が成功したかどうかを判断するために不可欠であるということです。
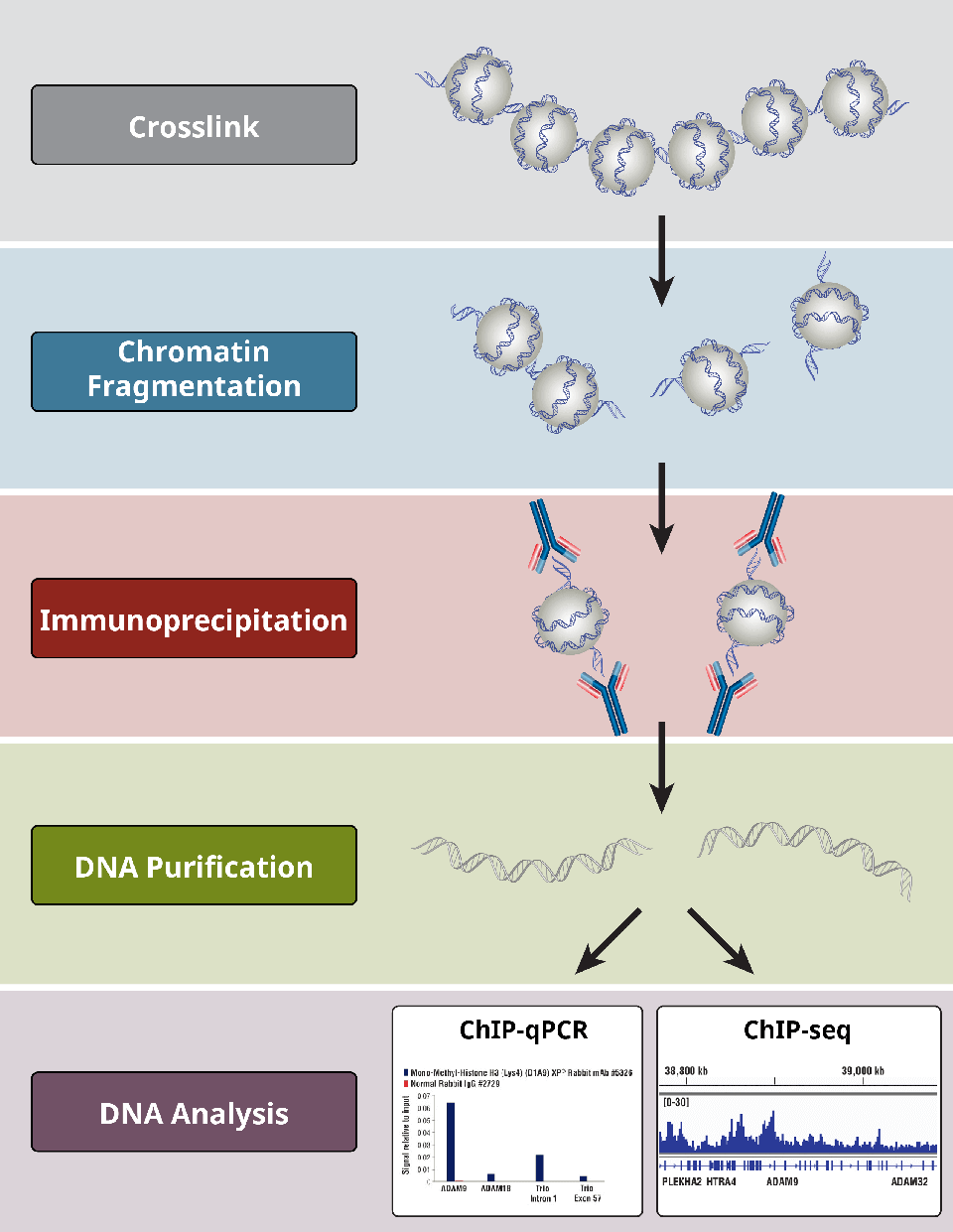
ChIPプロトコルの最も重要なステップの概要
ChIPプロトコルの最も重要なステップの概要
6.1 ChIP用に細胞や組織をクロスリンクするには?
クロスリンク試薬はタンパク質とそれらが結合するDNAを「固定」するのに使用されます。 この固定を実現するために、一般的にホルムアルデヒドベースの試薬が使用されます。 細胞や組織も一般的に同様の方法で固定されますが、組織は変性し始める前に標的組織に素早く浸透させるため、より長い固定時間とより迅速な固定送達が必要です。
クロマチンの過剰固定は、抗体のタンパク質標的への結合を阻害することに加え、超音波処理による断片化の効率も低下させることがあります。 そのため、タンパク質とその標的DNAの理想的な架橋を達成しながら、抗体と抗原の結合を最大化するために、固定時間を経験的に決定する必要がある。
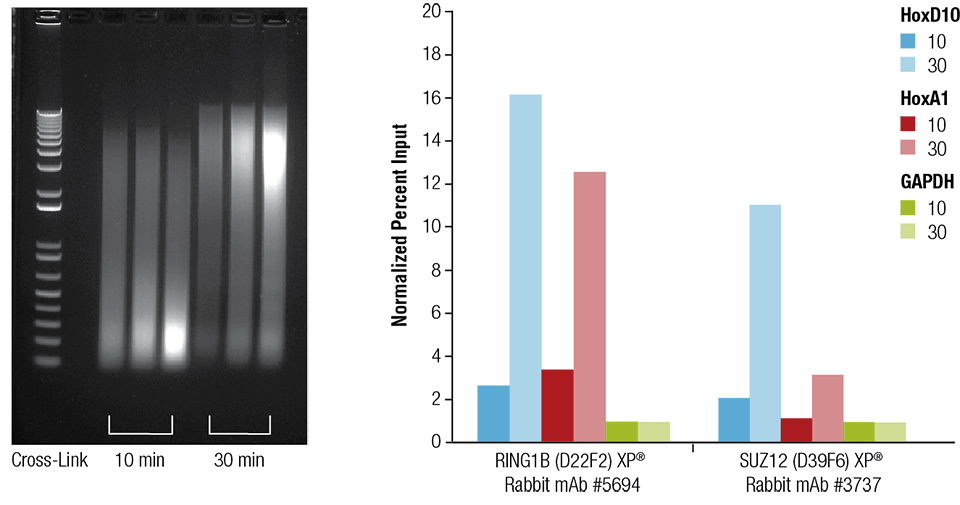
マウス心臓(H)、脳(B)、肝臓(L)を、指示通りに10分または30分架橋した(左側パネル)。 クロマチンを調製し、4分間超音波処理した。 SimpleChIP® Plus Sonication Chromatin IP Kit #56383を用いて、心臓組織から調製したクロマチンと指示した抗体を用いてChIPを行い、濃縮したDNAを指示した遺伝子に対するプライマーを用いたリアルタイムPCRにより定量した(右図)。 各サンプルの免疫沈降DNA量は、1に等しい陰性遺伝子座GAPDHに対する正規化シグナルとして表した。
マウス心臓(H)、脳(B)、肝臓(L)を指示通りに10分または30分架橋した(左図)。 クロマチンを調製し、4分間超音波処理した。 SimpleChIP® Plus Sonication Chromatin IP Kit #56383を用いて、心臓組織から調製したクロマチンと指示した抗体を用いてChIPを行い、濃縮したDNAを指示した遺伝子に対するプライマーを用いたリアルタイムPCRにより定量した(右図)。 各サンプルの免疫沈降DNA量は、陰性遺伝子座GAPDHに対する正規化シグナルとして表し、1.に等しい
6.2 クロマチンの断片化はどのように行うのか
ChIP 実験の成功には、クロマチン断片化が不可欠である。 クロマチンの断片化はクロマチンを可溶化するために必要であり、その共沈を可能にする。 また、DNA断片のサイズがChIPアッセイの分解能を決定するため、クロマチンの断片化に依存する。
酵素消化では、ミクロコッカールヌクレアーゼ(MNase)を用い、ヌクレオソーム間の二本鎖DNAを切断してクロマチン断片を生成させる。 MNaseによる完全消化では150塩基対のDNA断片(モノヌクレオソーム)が、不完全消化では150から750塩基対のDNA断片(モノ、ジ、トリヌクレオソーム)が生成されます。 ソニケーションは、機械的な力を利用してクロマチンを断片化する。 X-ChIPでは、酵素消化または超音波処理のいずれかを用いてクロマチンをせん断する。 超音波処理ChIPプロトコルの超音波処理条件は、細胞の種類や実験条件によって異なるため、経験的に決定する必要がある。 消化条件は細胞種や組織によって異なりますが、クロマチン断片のサイズはIPの前に分析する必要があります。
N-ChIPでは、未固定のサンプルでタンパク質結合を維持するために、ヌクレアーゼを用いてクロマチンを断片化します。
6.3 ChIPに酵素消化を使用する理由
N-ChIPでは、タンパク質がDNAに架橋されておらず、超音波による断片化に伴う過酷な条件では、DNAからクロマチンタンパク質が解離するため、核酸分解を用いる必要がある。 ヒストンとDNAの結合は非常に強固で安定しているため、N-ChIPはヒストン蛋白質とDNAの相互作用の解析に理想的である。 しかし、N-ChIPは転写因子や補酵素のクロマチン結合の解析には適していない。
X-ChIPでは、酵素分解または超音波処理のどちらかを用いてクロマチンを断片化することが可能である。 酵素消化の利点は、断片化の一貫性と穏やかな断片化条件(低熱と洗剤)により、クロマチンと抗体エピトープの完全性をよりよく維持できることで、転写因子や補酵素結合クロマチンの免疫エンリッチメントが向上する。
6.4 なぜChIPに超音波処理を用いるのか
酵素消化によるクロマチン断片化とは異なり、超音波処理では機械力を頼りにクロマチンを小さな断片に断片化している。 免疫強化のためのクロマチン断片の理想的なサイズは、200から1000塩基対の間である。 ソニケーションはクロマチンを断片化するために用いられる伝統的な方法であり、従来のプローブソニケーターや、より集中的なソニケーションが可能なウォーターバスソニケーターを用いて実行することができる。 ソニケーションは、クロマチンの真にランダムな断片を生成する。しかし、異なる細胞株や組織で広範囲に最適化する必要があり、実験ごとに再現することは困難である。
6.5 ChIPにおけるクロマチン超音波処理の最適化
ソニケーションによるクロマチン断片化は、従来から高い洗剤バッファーを使用し、熱を発生するため、クロマチンや抗体エピトープの完全性が損なわれる可能性がありました。 したがって、クロマチンの断片化に使用する超音波処理の量は、異なる細胞株や組織に対して実験的に決定する必要がある。 qPCR、DNAチップ、またはNGSによるダウンストリーム解析を伴う完全なChIPアッセイに着手する前に、ゲル電気泳動を使用して、さまざまな時間超音波処理したクロマチン試料を解析する必要があります。 フラグメントサイズは超音波処理時間に依存する-超音波処理時間が長くなると、フラグメントサイズは減少する。 しかし、超音波処理時間を長くしても、良い結果が得られないことがデータから示唆されている。 したがって、精製した免疫沈降DNAをゲル上で処理し、理想的なフラグメントサイズを決定することは、目的のDNAサイズに必要な最小限の超音波処理量を決定し、クロマチンに不必要な損傷を与えないための簡単な方法である。 レーン1は消化不足のクロマチンを示している。 レーン2は適切に消化されたクロマチン、レーン3は過剰消化されたクロマチンを示す。
Enzyme-digested chromatin on an agarose gelを実行した。 Lane 1は消化不良のクロマチンを示す。 レーン2は適切に消化されたクロマチン、レーン3は消化されすぎたクロマチンです。
6.6 ChIPのための抗体の選び方
ChIP実験の成功には、適切な抗体を選ぶことが欠かせません。 ChIP実験に使用する抗体は、目的のタンパク質に特異的で、抗原に対して高い親和性を持っている必要があります。 ChIPまたはChIP-seq実験に最適な抗体は、ChIP-validatedまたはChIP-seq-validated抗体です。 目的の遺伝子に対して利用可能なChIP-validated抗体がない場合、次善の策としてIPでバリデートされた抗体を選択することができます。 IPバリデートされた抗体がすべてChIPで機能するわけではなく、ChIPバリデートされた抗体がすべてChIP-seqで機能するわけでもないことに留意することが重要です。 さらに、ウエスタン、IP、IF、フロー、IHCなど、他のアプリケーションでバリデートされた抗体ほど、抗体の性能と特異性に対する信頼性が高くなります。
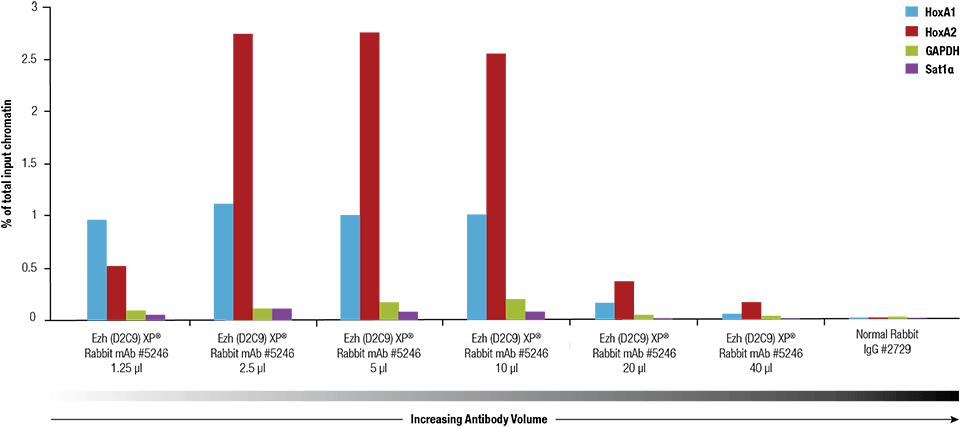
Ezh2 (D2C9) XP® Rabbit mAb #5246 は SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 を用いて 4 x 106 NCCIT 細胞から調製したクロスリンクしたクロマチンで滴定されました。
Ezh2 (D2C9) XP® Rabbit mAb #5246 は SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 を用いて 4 x 106 NCCIT 細胞から調製したクロスリンクロマチンに滴定しました。
6.7 免疫沈降法
抗体は目的のタンパク質とその結合した DNA を捉えるのに使用されています。 抗体濃度は経験的に決定する必要があり、一般的にはクロマチンDNA10μg(約4×106細胞に相当)あたり0.5〜2.0μgの抗体を使用することを出発点として決定する。 バッファーのストリンジェンシーや洗浄時間は、抗体の標的抗原への親和性に依存するため、経験的に決定する必要があります。 通常、抗体とクロマチンのインキュベーションは2時間から一晩行います。
抗体-抗原(+DNA)複合体は、抗体結合樹脂にアフィニティ捕捉されます。 プロテインAやプロテインBを結合させたChIPグレードの磁性ビーズ、セファロースビーズ、アガロースビーズで構成されています。 ビーズは通常、抗体:クロマチンとともに2~4時間インキュベートされます。
非抗体結合クロマチンを除去するための洗浄工程、架橋の反転(X-ChIP)、DNAの精製が必要となります。 また、バックグラウンド(シグナル:ノイズ)を測定するためにIgGコントロールのIPを行う必要があります。 非特異的結合を決定するために、陽性対照抗体(例えば、総ヒストンH3)および/または陽性対照qPCRプライマー(既知の陽性および陰性標的タンパク質結合遺伝子座用)も含まれなければならない。 最適な結果を得るためには、qPCRによるクロマチンIPのQCは、下流のNGS解析の前に実施されるべきである。
6.9 クロマチン架橋の解除方法
架橋は高熱と高塩(いずれも重要な成分)で解除される。
6.10 DNAの精製方法
クロマチン架橋を除去した後、DNAは古典的なフェノール-クロロホルムに続いてエタノール沈殿法を用いて精製するか、カラムベースのDNA精製キットを用いて精製されます。
濃縮DNAの解析方法
DNAが精製されたら、ChIP-PCR、ChIP-qPCR、ChIP-chip、ChIP-seqなどいくつかのダウンストリーム解析を行うことができる
7.1 ChIP-PCRおよびChIP-qPCR解析
ChIP-PCRおよびChIP-qPCR解析は単一遺伝子解析に最適で、迅速かつコスト効率の高い方法でDNAの特定の断片を増幅および定量するために使用できる
7.。2 ChIP-chip analysis
ChIP-chip analysis using tiling DNA microarray chips to create a genome-wide, high resolution map of protein binding and protein modification.
7.3 ChIP-seq analysis
ChIP-seq analysis uses standard NGS technology to align purified DNA with previously annotated whole genome to identify genome-wide protein binding profiles.これは、精製されたDNAと以前に注釈された全ゲノムとのアラインメントをとるための標準NGS技術を用いた分析で、ゲノム全体のタンパク質結合プロファイルを特定します。