Che cos’è la ChIP?
L’immunoprecipitazione della cromatina, o ChIP, è una tecnologia basata su anticorpi usata per arricchire selettivamente proteine specifiche legate al DNA insieme ai loro obiettivi del DNA. ChIP è usata per studiare una particolare interazione proteina-DNA, diverse interazioni proteina-DNA, o interazioni attraverso l’intero genoma o un sottoinsieme di geni.
ChIP utilizza anticorpi che riconoscono e legano selettivamente le proteine, compresi gli istoni, le modifiche degli istoni, i fattori di trascrizione e i cofattori, per fornire informazioni sugli stati della cromatina e sulla trascrizione genica. Una combinazione di analisi proteomica e tecniche di biologia molecolare utilizzate nella ChIP permettono di comprendere l’espressione e la regolazione genica in cellule o tessuti di interesse.
Quando viene utilizzata la ChIP?
Tipicamente, la ChIP viene utilizzata per identificare l’abbondanza relativa di una specifica proteina o una specifica modifica proteica in una certa regione del genoma. La ChIP può essere usata per rispondere a una moltitudine di domande scientifiche che coinvolgono l’interazione delle proteine e della cromatina. Per esempio, la ChIP può essere usata per confrontare la presenza di certe proteine in vari loci, mappare le varie proteine in una regione genomica di interesse, o quantificare il legame delle proteine a un gene inducibile in risposta a uno stimolo nel tempo.
Come funziona la ChIP?
Il principio dietro la ChIP è relativamente semplice e si basa sull’uso di un anticorpo per isolare, o precipitare, una certa proteina, istone, fattore di trascrizione o cofattore e la sua cromatina legata da una miscela proteica che è stata estratta da cellule o tessuti. Da qui, il nome della tecnica: Immunoprecipitazione della cromatina. In ChIP-PCR o ChIP-seq, i frammenti di DNA immunizzati sono poi in grado di essere identificati e quantificati utilizzando reagenti PCR o qPCR ampiamente disponibili e tecnologie di Next Generation Sequencing (NGS).
Cos’è la ChIP nativa (N-ChIP) rispetto alla ChIP reticolata (X-ChIP)?
Ci sono 2 tipi di tecniche ChIP che possono essere eseguite a seconda della questione sperimentale e del materiale di partenza dell’esperimento: 1) ChIP nativa (N-ChIP) e 2) ChIP reticolata (X-ChIP). Entrambi i tipi di ChIP hanno vantaggi e svantaggi:
- Nella N-ChIP, non viene usato alcun agente di fissaggio per reticolare le proteine alla cromatina. Invece, la cromatina nativa viene isolata da nuclei di cellule che vengono digeriti con una nucleasi. Poiché gli anticorpi sono sollevati contro gli antigeni non fissati, N-ChIP offre il vantaggio di un migliore riconoscimento e legame degli anticorpi ai loro antigeni bersaglio. La PCR può non essere necessaria per l’analisi a valle a causa dell’elevata abbondanza di proteine istone. Mentre questi vantaggi rendono N-ChIP un metodo attraente, esso può essere utilizzato solo per il rilevamento degli istoni. Inoltre, la perdita di legame della proteina durante la digestione della cromatina e le fasi di immunoprecipitazione possono distorcere i dati o impedire le analisi corrette.
- In X-ChIP, i fissatori chimici come la formaldeide sono usati per reticolare la proteina di interesse al DNA e la frammentazione della cromatina è ottenuta attraverso la sonicazione o la digestione nucleasi. Il vantaggio di X-ChIP è che può essere usato con proteine istone e non istone e generalmente richiede meno materiale cellulare di partenza rispetto a N-ChIP. X-ChIP minimizza anche le possibilità di perdita di proteine della cromatina durante l’estrazione, permettendo il rilevamento di interazioni proteiche transitorie. Tuttavia, la fase di precipitazione è meno efficiente e l’amplificazione del DNA tramite PCR è necessaria per le analisi a valle.
Quali sono i diversi tipi di test ChIP?
Una volta che l’immunoprecipitazione della cromatina è completa, diverse analisi a valle possono essere condotte sulla cromatina purificata e le proteine associate, istoni, fattori di trascrizione e cofattori. I metodi più comuni per l’analisi del singolo gene e l’analisi del genoma intero sono qPCR e ChIP-seq, rispettivamente. PCR e ChIP-chip sono anche opzioni per l’analisi a valle.
5.1 Quali sono i vantaggi della ChIP-PCR?
ChIP-PCR viene eseguita per analizzare le modifiche degli istoni e/o il legame delle proteine ad un sottoinsieme noto di loci target nel genoma. In ChIP-PCR, i frammenti di DNA immunizzati sono identificati e quantificati utilizzando reagenti e tecnologie PCR o qPCR ampiamente disponibili. Confronti rapidi e quantitativi di regioni specifiche all’interno del genoma su più campioni possono essere ottenuti usando ChIP-qPCR. Questo è più economico e più efficiente in termini di tempo rispetto ai metodi di sequenziamento dell’intero genoma.
5.2 Quali sono i vantaggi di ChIP-chip?
La tecnologia ChIP-chip si riferisce all’utilizzo di un chip microarray di DNA per analizzare i frammenti di DNA arricchiti di ChIP-immunità. L’utilizzo della tecnologia microarray di tiling del genoma permette un’analisi dell’intero genoma delle proteine che sono legate al DNA isolato e genera una mappa genomica ad alta risoluzione del legame delle proteine e delle modifiche delle proteine. ChIP-chip ha molteplici usi nella ricerca di base così come nella ricerca sulle malattie. Per esempio, può essere usato per identificare i siti di legame di fattori di trascrizione, enhancer e repressori e per confrontare questi tipi di proteine legate in campioni di controllo e patologici. Tuttavia, poiché il costo di NGS è diminuito sostanzialmente e risultati simili si ottengono utilizzando ChIP-seq, sempre più persone scelgono di eseguire ChIP-seq invece di ChIP-chip.
5.3 Quali sono i vantaggi di ChIP-seq?
Simile a ChIP-chip, ChIP-seq fornisce informazioni sul legame delle proteine a livello di genoma. Tuttavia, a differenza di ChIP-chip, ChIP-seq utilizza la tecnologia NGS per identificare i frammenti di DNA e mapparli rispetto all’intero genoma.
La tecnologia di amplificazione del DNA più contemporanea permette di condurre un’analisi robusta in pochi giorni con una bassa quantità di DNA in ingresso. Quando il materiale di partenza è scarso, questi progressi tecnologici nei metodi di preparazione delle librerie hanno reso possibili gli esperimenti ChIP-seq.
Inoltre, la nuova tecnologia in cui i campioni di DNA sono etichettati in modo univoco con brevi sequenze, note come codici a barre, ora permette ai singoli frammenti di essere raggruppati in una singola corsia di sequenziamento per analisi multiplex. Questo ha sostanzialmente aumentato l’efficienza e diminuito il costo degli esperimenti di sequenziamento del DNA, sostenendo ulteriormente le applicazioni ChIP-seq.
Insieme, a causa dei progressi nella tecnologia di sequenziamento del DNA, il vantaggio del ChIP-seq è che un gran numero di campioni di DNA arricchiti con ChIP può essere sequenziato a basso costo in un periodo di tempo relativamente breve con una maggiore sensibilità e precisione rispetto al ChIP-chip.
Quali sono le diverse fasi del test ChIP?
I test ChIP seguono un protocollo generale:
- Crosslinking delle proteine al DNA solo per X-ChIP
- Lisi delle cellule
- Frammentazione della cromatina per digestione (per X-ChIP e N-ChIP) o per sonicazione-(solo per X-ChIP)
- Immunoprecipitazione con anticorpi specifici
- Pulizia del DNA per analisi a valle
- Analisi del DNA tramite PCR, qPCR, microarray o NGS
Importante, i controlli positivi e negativi in ogni fase sono fondamentali per determinare se un esperimento ChIP ha avuto successo.
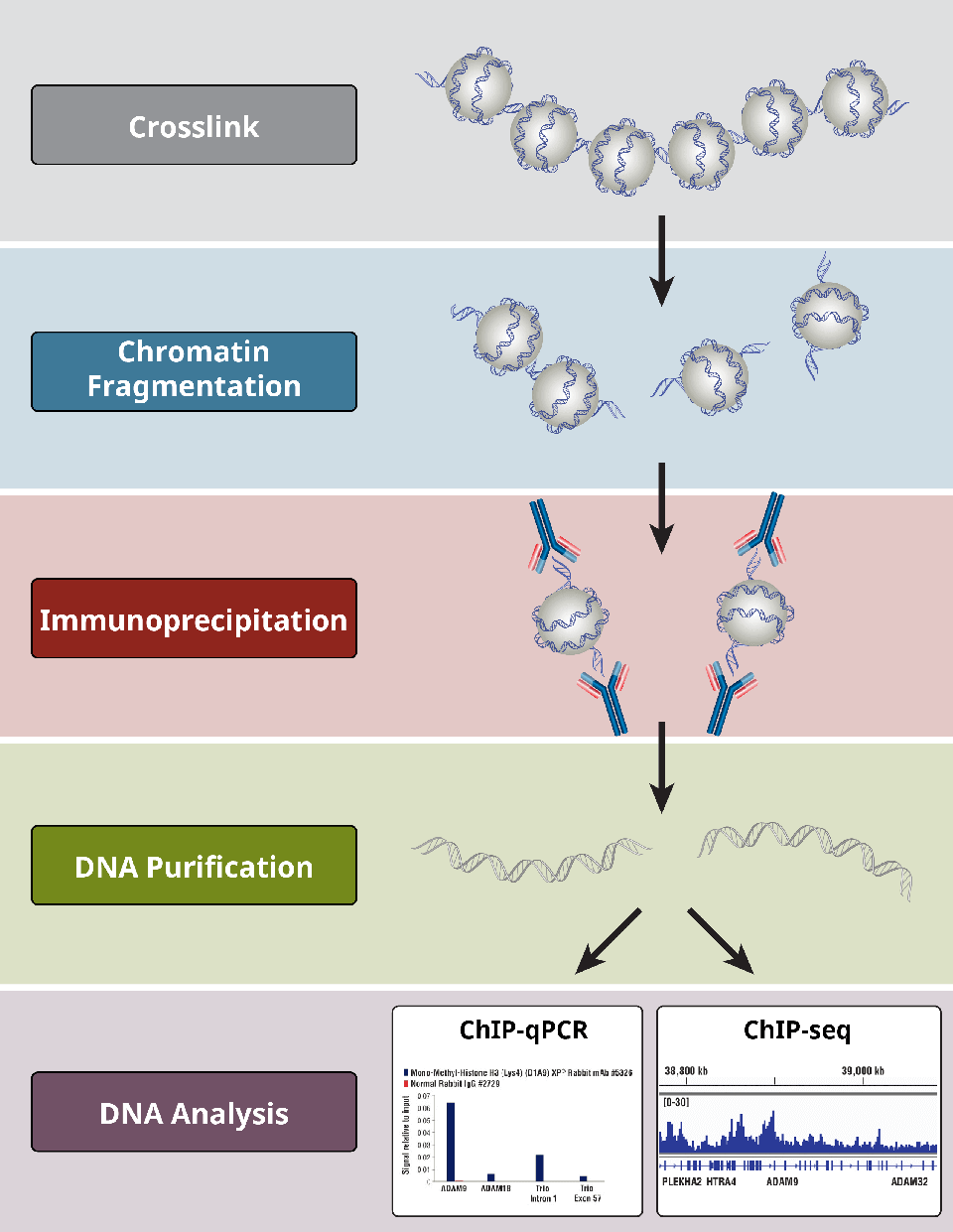
Panoramica dei passi più critici di un protocollo ChIP.
Panoramica dei passi più critici di un protocollo ChIP.
6.1 Come si reticolano cellule e tessuti per ChIP?
I reagenti di reticolazione sono usati per “fissare” le proteine al DNA che legano. I reagenti a base di formaldeide sono tipicamente usati per ottenere questa fissazione. Cellule e tessuti sono generalmente fissati in modo simile, ma il tessuto richiede un tempo di fissazione più lungo e una consegna di fissazione più rapida al fine di permeare rapidamente il tessuto bersaglio prima che inizi a degenerare.
La sovra-fissazione della cromatina può ridurre l’efficienza della frammentazione mediante sonicazione, oltre a inibire il legame degli anticorpi ai loro obiettivi proteici. Pertanto, il tempo di fissazione dovrebbe essere determinato empiricamente al fine di consentire il massimo anticorpo-antigene vincolante pur raggiungendo ideale reticolazione delle proteine al loro DNA bersaglio.
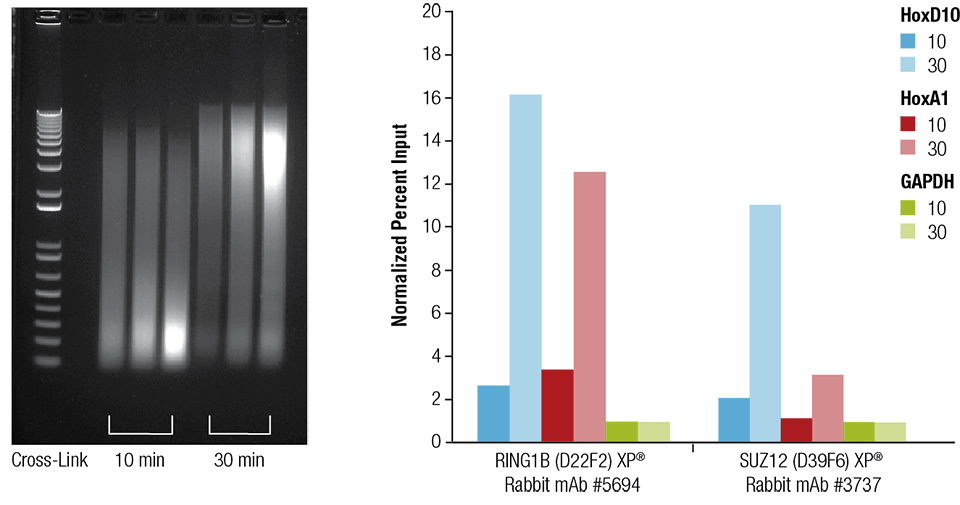
Cuore di topo (H), cervello (B) e fegato (L) sono stati reticolati per 10 o 30 minuti come indicato (pannello sinistro). La cromatina è stata preparata e sonicata per 4 minuti. ChIP è stata eseguita utilizzando la cromatina preparata dal tessuto cardiaco con gli anticorpi indicati utilizzando il SimpleChIP® Plus Sonication Chromatin IP Kit #56383 e il DNA arricchito è stato quantificato mediante PCR in tempo reale utilizzando primer per i geni indicati (pannello destro). La quantità di DNA immunoprecipitato in ogni campione è rappresentata come segnale normalizzato al loci negativo GAPDH, che è uguale a 1.
Cuore di topo (H), cervello (B), e fegato (L) sono stati reticolati per 10 o 30 minuti come indicato (pannello sinistro). La cromatina è stata preparata e sonicata per 4 minuti. ChIP è stata eseguita utilizzando la cromatina preparata dal tessuto cardiaco con gli anticorpi indicati utilizzando il SimpleChIP® Plus Sonication Chromatin IP Kit #56383 e il DNA arricchito è stato quantificato mediante PCR in tempo reale utilizzando primer per i geni indicati (pannello destro). La quantità di DNA immunoprecipitato in ogni campione è rappresentata come segnale normalizzato al loci negativo GAPDH, che è uguale a 1.
6.2 Come si frammenta la cromatina?
La frammentazione della cromatina è essenziale per il successo di un esperimento ChIP. La frammentazione della cromatina è necessaria per solubilizzare la cromatina e permette la sua coprecipitazione. Inoltre, la risoluzione del saggio ChIP dipende dalla frammentazione della cromatina, poiché la dimensione del frammento di DNA determina la risoluzione del saggio ChIP.
La digestione enzimatica utilizza la nucleasi micrococcica (MNase), che taglia il DNA a doppio filamento tra i nucleosomi per generare frammenti di cromatina. Mentre una digestione completa della MNasi genera frammenti di DNA di 150 coppie di basi (mono-nucleosomi), una digestione incompleta genera frammenti di DNA tra 150 e 750 coppie di basi (mono-, di- e tri-nucleosomi). La sonicazione usa la forza meccanica per frammentare la cromatina. La sonicazione frammenta la cromatina tra e all’interno dei nucleosomi, generando una gamma di frammenti di cromatina tra 150 e 1000 paia di basi.
Per X-ChIP, sia la digestione enzimatica che la sonicazione sono usate per tagliare la cromatina. Le condizioni di sonicazione nel protocollo di sonicazione ChIP devono essere determinate empiricamente, in quanto differiscono per tipo di cellula e condizione sperimentale. Condizioni di digestione sono più coerenti tra i diversi tipi di cellule e tessuti, ma la dimensione del frammento di cromatina dovrebbe ancora essere analizzato prima di IP.
Per N-ChIP, nucleasi vengono utilizzati per frammentare la cromatina al fine di mantenere il legame proteico in campioni non fissati. La frammentazione nucleasica dovrebbe anche essere determinata empiricamente per ridurre al minimo la sovradigestione della cromatina.
6.3 Perché usare la digestione enzimatica per ChIP?
La digestione nucleasica deve essere usata per N-ChIP poiché le proteine non sono reticolate al DNA e le condizioni difficili associate alla frammentazione basata sulla sonicazione porterebbero alla dissociazione delle proteine della cromatina dal DNA. N-ChIP è ideale per analizzare le interazioni istone-proteina-DNA, perché il legame istone-DNA è molto forte e stabile. Tuttavia, N-ChIP non funziona bene per l’analisi del fattore di trascrizione e del legame alla cromatina dei cofattori.
Per frammentare la cromatina in X-ChIP si può usare sia la digestione enzimatica che la sonicazione. I vantaggi della digestione enzimatica includono la consistenza della frammentazione e le condizioni di frammentazione mite (meno calore e detergente) che preservano meglio l’integrità della cromatina e gli epitopi degli anticorpi, con conseguente aumento dell’arricchimento immunitario del fattore di trascrizione e della cromatina legata al cofattore.
6.4 Perché usare la sonicazione per frammentare la cromatina per ChIP?
A differenza della frammentazione della cromatina ottenuta tramite digestione enzimatica, la sonicazione si basa su forze meccaniche per frammentare la cromatina in pezzi più piccoli. La dimensione ideale dei frammenti di cromatina per l’arricchimento immunitario è compresa tra 200 e 1000 coppie di basi. La sonicazione è il metodo tradizionale utilizzato per frammentare la cromatina e può essere eseguita utilizzando un sonicatore tradizionale o sonicatori più avanzati a bagno d’acqua che forniscono una sonicazione più mirata. La sonicazione genera frammenti veramente randomizzati di cromatina; tuttavia, richiede una vasta ottimizzazione attraverso diverse linee cellulari e tessuti ed è difficile da riprodurre da un esperimento all’altro. Il requisito di alti tamponi detergenti e il calore generato durante la sonicazione può danneggiare l’integrità sia della cromatina che degli epitopi degli anticorpi sulle proteine della cromatina.
6.5 Ottimizzazione della sonicazione della cromatina per ChIP
La sonicazione basata sulla frammentazione della cromatina utilizza tradizionalmente alti tamponi detergenti e genera calore, entrambi i quali possono danneggiare l’integrità della cromatina e degli epitopi degli anticorpi. Pertanto, la quantità di sonicazione utilizzata per frammentare la cromatina deve essere determinata sperimentalmente per diverse linee cellulari e tessuti. Si deve identificare e utilizzare la quantità minima di sonicazione necessaria per generare frammenti di DNA da 150 a 1000 paia di basi per ridurre al minimo i danni alla cromatina.
Prima di intraprendere un test ChIP completo con analisi a valle di qPCR, DNA chip, o NGS, elettroforesi gel dovrebbe essere utilizzato per analizzare campioni di cromatina sonicata per vari tempi. La dimensione del frammento dipende dal tempo di sonicazione: la dimensione del frammento diminuisce all’aumentare del tempo di sonicazione. Tuttavia, i dati suggeriscono che tempi di sonicazione più lunghi non portano a risultati migliori. Pertanto, l’esecuzione di DNA purificato immunoprecipitato su un gel e la determinazione della dimensione ideale del frammento è un metodo semplice per determinare la quantità minima di sonicazione necessaria per la dimensione desiderata del DNA e per evitare danni inutili alla cromatina.

La cromatina digerita dall’enzima è stata eseguita su un gel di agarosio. La corsia 1 mostra la cromatina sottodigerita. La corsia 2 mostra la cromatina correttamente digerita e la corsia 3 mostra la cromatina sovradigerita.
La cromatina digerita dall’enzima è stata eseguita su un gel di agarosio. La corsia 1 mostra una cromatina sottodigerita. La corsia 2 mostra la cromatina correttamente digerita, e la corsia 3 mostra la cromatina sovradigerita.
6.6 Come si sceglie un anticorpo per ChIP?
Scegliere un anticorpo appropriato per un esperimento ChIP è fondamentale per il suo successo. Gli anticorpi usati in un esperimento ChIP dovrebbero essere specifici per la proteina di interesse e avere un’alta affinità per l’antigene. La scelta migliore di anticorpo per un esperimento ChIP o ChIP-seq è un anticorpo ChIP- o ChIP-seq-validato. Se non c’è nessun anticorpo convalidato per ChIP disponibile per il gene di interesse, la prossima scelta migliore è un anticorpo che è stato convalidato in IP. È importante notare che non tutti gli anticorpi convalidati IP funzionano in ChIP e non tutti gli anticorpi convalidati ChIP funzionano in ChIP-seq. Inoltre, più un anticorpo è convalidato in altre applicazioni, come western, IP, IF, flusso e IHC, più fiducia si può avere nelle prestazioni e specificità dell’anticorpo. La convalida di un anticorpo deve essere seguita dalla determinazione empirica della concentrazione ottimale dell’anticorpo, insieme alle condizioni di lavaggio IP.
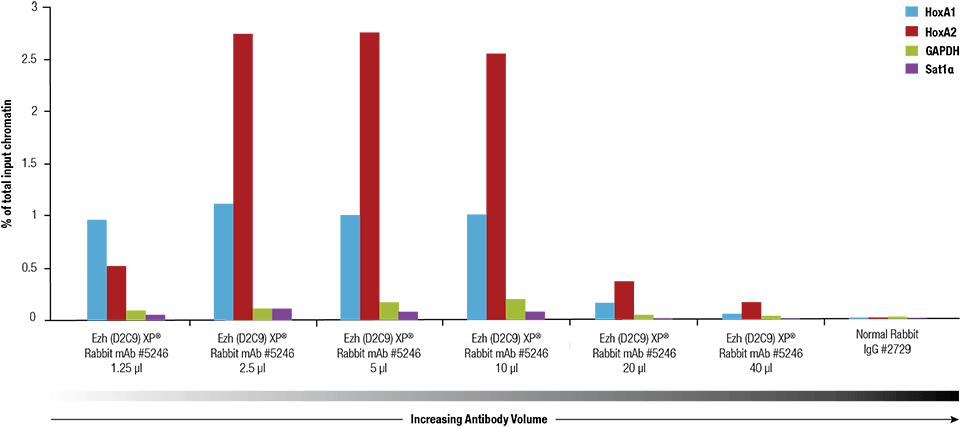
Ezh2 (D2C9) XP® Rabbit mAb #5246 è stato titolato utilizzando il SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 su cromatina reticolata preparata da 4 x 106 cellule NCCIT.
Ezh2 (D2C9) XP® Rabbit mAb #5246 è stato titolato usando il SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 su cromatina reticolata preparata da 4 x 106 cellule NCCIT.
6.7 Come eseguire l’immunoprecipitazione
Gli anticorpi sono usati per catturare una proteina di interesse e il suo DNA legato. La concentrazione degli anticorpi dovrebbe essere determinata empiricamente, con un punto di partenza generale di 0,5-2,0 μg di anticorpo utilizzato per 10 μg di DNA della cromatina (corrisponde a circa 4 x 106 cellule). La rigorosità dei buffer e i tempi di lavaggio dovrebbero anche essere determinati empiricamente in quanto dipendono dall’affinità dell’anticorpo al suo antigene bersaglio. Tipicamente, le incubazioni anticorpo-cromatina sono fatte da 2 ore a una notte.
I complessi anticorpo-antigene (+DNA) sono catturati per affinità su una resina che lega l’anticorpo. Negli esperimenti ChIP questa resina è tipicamente composta da perline magnetiche, sefarosio o agarosio di grado ChIP coniugate con la proteina A e/o la proteina B. Gli anticorpi legano le perline di proteina A e/o proteina G con varie affinità a seconda della specie in cui sono state sviluppate e il sottotipo IgG della loro catena pesante. Le microsfere sono tipicamente incubate con anticorpo:cromatina per 2-4 ore.
Le fasi di lavaggio sono necessarie per rimuovere la cromatina non legata all’anticorpo, seguita dall’inversione dei crosslink (per X-ChIP) e dalla purificazione del DNA. Inoltre, un IP di controllo IgG deve essere eseguito per determinare il background (segnale:rumore). Anticorpi di controllo positivi (es. istone totale H3) e/o primer qPCR di controllo positivo (per loci di legame noti di proteine target positive e negative) devono anche essere inclusi per determinare il legame non specifico. Per risultati ottimali, il QC della cromatina IP tramite qPCR dovrebbe essere eseguito prima dell’analisi NGS a valle.
6.8 Come eluire la cromatina dalle microsfere di proteina A/G
La cromatina viene eluita dalle microsfere di proteina A/G utilizzando detergente e calore. Il “vortexing” a bassa velocità o la miscelazione sono necessari per mantenere le perline in sospensione e per aumentare l’eluizione della cromatina.
6.9 Come invertire i crosslink della cromatina
I crosslink sono invertiti da calore e sale elevati (entrambi questi sono componenti vitali). Viene anche aggiunta la proteinasi K per digerire le proteine associate alla cromatina e gli anticorpi aggiunti, permettendo una purificazione del DNA più efficiente a valle.
6.10 Come purificare il DNA
Dopo che i crosslink della cromatina vengono rimossi, il DNA viene purificato utilizzando i classici metodi di fenolo-cloroformio seguiti da precipitazione con etanolo o utilizzando kit di purificazione del DNA su colonna.
Come analizzare il DNA arricchito
Una volta che il DNA è purificato, diverse analisi a valle può essere condotto, tra cui ChIP-PCR, ChIP-qPCR, ChIP-chip, e ChIP-seq.
7.1 Analisi ChIP-PCR e ChIP-qPCR
Le analisi ChIP-PCR e ChIP-qPCR sono le migliori per l’analisi di un singolo gene e possono essere utilizzate per amplificare e quantificare specifici frammenti di DNA in modo rapido e conveniente.
7.2 Analisi ChIP-chip
L’analisi ChIP-chip utilizza chip microarray di DNA in tiling per creare una mappa genomica ad alta risoluzione del legame proteico e della modifica delle proteine.
7.3 Analisi ChIP-seq
L’analisi ChIP-seq utilizza la tecnologia NGS standard per allineare il DNA purificato con genomi interi precedentemente annotati per identificare profili di legame proteico a livello genomico.