¿Qué es la ChIP?
La inmunoprecipitación de la cromatina, o ChIP, es una tecnología basada en anticuerpos que se utiliza para enriquecer selectivamente proteínas específicas de unión al ADN junto con sus objetivos de ADN. La ChIP se utiliza para investigar una determinada interacción proteína-ADN, varias interacciones proteína-ADN o interacciones en todo el genoma o en un subconjunto de genes.
La ChIP utiliza anticuerpos que reconocen y se unen selectivamente a las proteínas, incluidas las histonas, las modificaciones de las histonas, los factores de transcripción y los cofactores, para proporcionar información sobre los estados de la cromatina y la transcripción de los genes. La combinación del análisis proteómico y las técnicas de biología molecular utilizadas en la ChIP permiten comprender la expresión y la regulación de los genes en las células o los tejidos de interés.
¿Cuándo se utiliza la ChIP?
Típicamente, la ChIP se utiliza para identificar la abundancia relativa de una proteína específica o de una modificación proteica específica en una determinada región del genoma. El ChIP puede utilizarse para responder a una multitud de preguntas científicas relacionadas con la interacción de las proteínas y la cromatina. Por ejemplo, el ChIP puede utilizarse para comparar la presencia de determinadas proteínas en varios loci, mapear las distintas proteínas en una región genómica de interés o cuantificar la unión de proteínas a un gen inducible en respuesta a un estímulo a lo largo del tiempo.
¿Cómo funciona el ChIP?
El principio en el que se basa la ChIP es relativamente sencillo y se basa en el uso de un anticuerpo para aislar, o precipitar, una determinada proteína, histona, factor de transcripción o cofactor y su cromatina unida a partir de una mezcla de proteínas extraída de células o tejidos. De ahí el nombre de la técnica: Inmunoprecipitación de la cromatina. En la ChIP-PCR o ChIP-seq, los fragmentos de ADN enriquecidos por la inmunidad pueden identificarse y cuantificarse utilizando reactivos de PCR o qPCR ampliamente disponibles y tecnologías de secuenciación de próxima generación (NGS).
¿Qué es la ChIP nativa (N-ChIP) frente a la ChIP reticulada (X-ChIP)?
Hay dos tipos de técnicas de ChIP que pueden llevarse a cabo dependiendo de la pregunta experimental y del material de partida para el experimento: 1) ChIP nativo (N-ChIP) y 2) ChIP reticulado (X-ChIP). Ambos tipos de ChIP tienen ventajas e inconvenientes:
- En el N-ChIP no se utiliza ningún agente de fijación para reticular las proteínas a la cromatina. En su lugar, la cromatina nativa se aísla de los núcleos celulares que se digieren con una nucleasa. Debido a que los anticuerpos se levantan contra antígenos no fijados, la N-ChIP ofrece la ventaja de un mejor reconocimiento y unión de los anticuerpos a sus antígenos objetivo. La PCR puede no ser necesaria para el análisis posterior debido a la gran abundancia de proteínas histónicas. Aunque estas ventajas hacen que el N-ChIP sea un método atractivo, sólo puede utilizarse para la detección de histonas. Además, la pérdida de la unión de la proteína durante los pasos de digestión de la cromatina e inmunoprecipitación puede sesgar los datos o impedir los análisis adecuados.
- En el X-ChIP, se utilizan fijadores químicos como el formaldehído para reticular la proteína de interés con el ADN y la fragmentación de la cromatina se consigue mediante sonicación o digestión con nucleasas. La ventaja de la X-ChIP es que puede utilizarse con proteínas histónicas y no histónicas y generalmente requiere menos material celular de partida que la N-ChIP. El X-ChIP también minimiza las posibilidades de pérdida de proteínas de la cromatina durante la extracción, permitiendo la detección de interacciones proteicas transitorias. Sin embargo, el paso de precipitación es menos eficiente y la amplificación del ADN por PCR es necesaria para los análisis posteriores.
¿Cuáles son los diferentes tipos de ensayos de ChIP?
Una vez que se ha completado la inmunoprecipitación de la cromatina en sí, se pueden llevar a cabo varios análisis posteriores sobre la cromatina purificada y las proteínas asociadas, histonas, factores de transcripción y cofactores. Los métodos más comunes para el análisis de un solo gen y de todo el genoma son la qPCR y la ChIP-seq, respectivamente. La PCR y el ChIP-chip también son opciones para el análisis posterior.
5.1 ¿Cuáles son las ventajas de la ChIP-PCR?
La ChIP-PCR se realiza para analizar las modificaciones de las histonas y/o la unión de proteínas a un subconjunto conocido de loci objetivo en el genoma. En la ChIP-PCR, se identifican y cuantifican fragmentos de ADN enriquecidos por el sistema inmunitario utilizando reactivos y tecnologías de PCR o qPCR ampliamente disponibles. Mediante la ChIP-qPCR se pueden realizar comparaciones rápidas y cuantitativas de regiones específicas del genoma en múltiples muestras. Esto es más barato y más eficiente en términos de tiempo que los métodos de secuenciación del genoma completo.
5.2 ¿Cuáles son las ventajas de ChIP-chip?
La tecnología ChIP-chip se refiere a la utilización de un chip de microarrays de ADN para analizar fragmentos de ADN enriquecidos con ChIP. El uso de la tecnología de microarrays de genoma permite un análisis de todo el genoma de las proteínas que se unen al ADN aislado y genera un mapa genómico de alta resolución de la unión de proteínas y de las modificaciones de las mismas. El ChIP-chip tiene múltiples usos en la investigación básica y en la investigación de enfermedades. Por ejemplo, puede utilizarse para identificar los sitios de unión de factores de transcripción, potenciadores y represores y para comparar estos tipos de proteínas unidas en muestras de control y patológicas. Sin embargo, dado que el coste de la NGS ha disminuido sustancialmente y se obtienen resultados similares utilizando ChIP-seq, cada vez más personas eligen realizar ChIP-seq en lugar de ChIP-chip.
5.3 ¿Cuáles son las ventajas de ChIP-seq?
De forma similar a ChIP-chip, ChIP-seq proporciona información sobre la unión de proteínas en todo el genoma. Sin embargo, a diferencia de ChIP-chip, ChIP-seq utiliza la tecnología NGS para identificar fragmentos de ADN y mapearlos con respecto a todo el genoma.
La tecnología de amplificación de ADN más contemporánea permite realizar análisis robustos en cuestión de días con una baja cantidad de ADN de entrada. Cuando el material de partida es escaso, estos avances tecnológicos en los métodos de preparación de bibliotecas han hecho posibles los experimentos de ChIP-seq.
Además, la nueva tecnología en la que las muestras de ADN se etiquetan de forma exclusiva con secuencias cortas, conocidas como códigos de barras, permite ahora agrupar fragmentos individuales en una única vía de secuenciación para realizar análisis multiplexados. Esto ha aumentado sustancialmente la eficiencia y ha disminuido el coste de los experimentos de secuenciación de ADN, apoyando aún más las aplicaciones de ChIP-seq.
En conjunto, debido a los avances en la tecnología de secuenciación de ADN, la ventaja de ChIP-seq es que se puede secuenciar un gran número de muestras de ADN enriquecido por ChIP de forma barata en un periodo de tiempo relativamente corto y con mayor sensibilidad y precisión que ChIP-chip.
¿Cuáles son los diferentes pasos del ensayo ChIP?
Los ensayos ChIP siguen un protocolo general:
- Encruzamiento de las proteínas con el ADN sólo para X-ChIP
- Lisis celular
- Fragmentación de la cromatina por digestión (para X-ChIP y N-ChIP) o cizallamiento basado en sonicación (para X-ChIP).(sólo para X-ChIP)
- Immunoprecipitación con anticuerpos específicos
- Limpieza del ADN para los análisis posteriores
- Análisis del ADN mediante PCR, qPCR, microarray, o NGS
Es importante que los controles positivos y negativos en cada paso sean integrales para determinar si un experimento de ChIP ha sido exitoso.
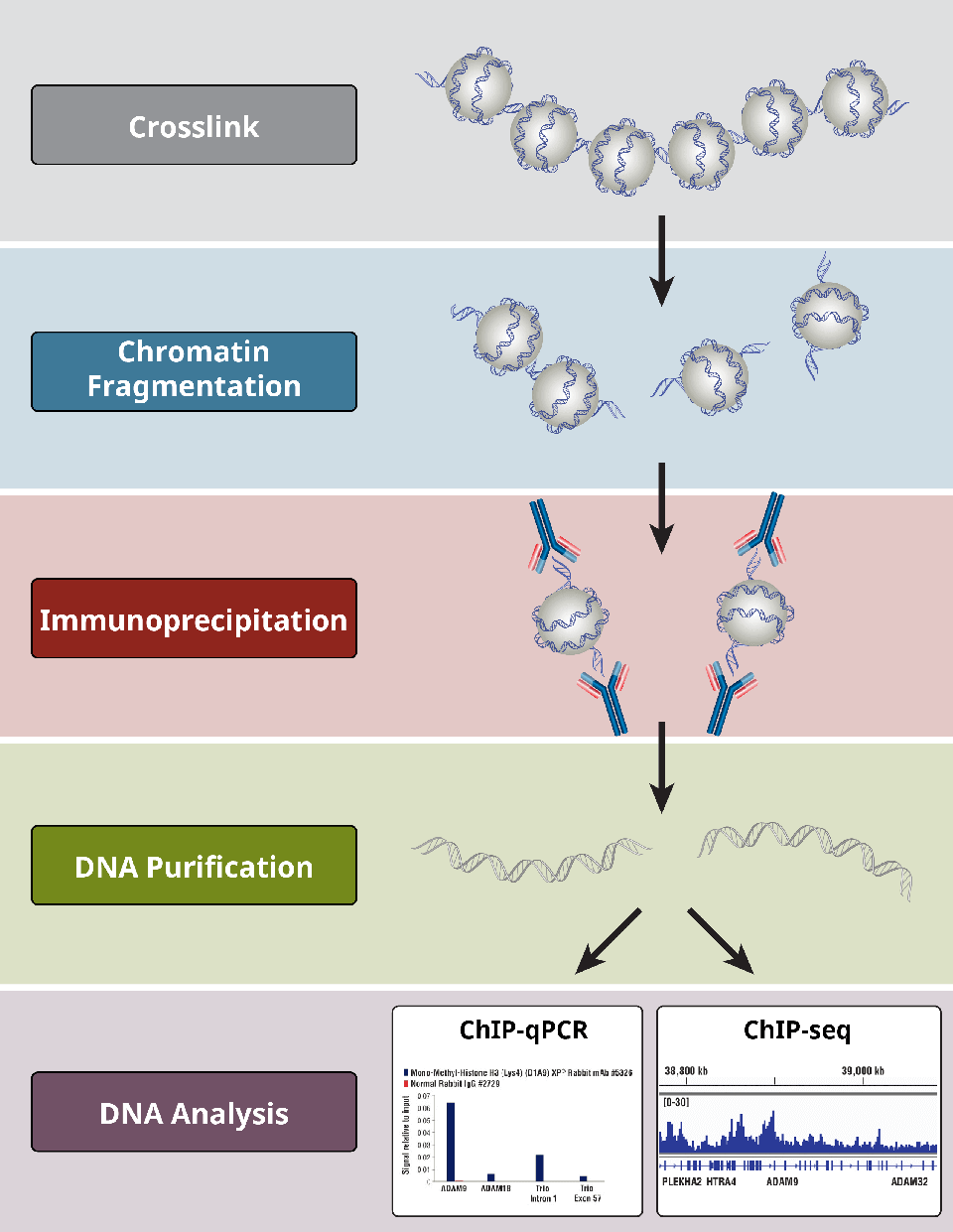
Resumen de los pasos más críticos de un protocolo de ChIP.
Resumen de los pasos más críticos de un protocolo de ChIP.
6.1 ¿Cómo se reticulan las células y los tejidos para el ChIP?
Los reactivos de reticulación se utilizan para «fijar» las proteínas al ADN al que se unen. Normalmente se utilizan reactivos basados en el formaldehído para lograr esta fijación. Las células y los tejidos generalmente se fijan de manera similar, pero los tejidos requieren un tiempo de fijación más largo y una entrega de fijación más rápida para impregnar rápidamente el tejido objetivo antes de que comience a degenerar.
La fijación excesiva de la cromatina puede reducir la eficiencia de la fragmentación por sonicación, además de inhibir la unión de los anticuerpos a sus objetivos proteicos. Por lo tanto, el tiempo de fijación debe determinarse empíricamente para permitir la máxima unión entre el anticuerpo y el antígeno a la vez que se consigue un entrecruzamiento ideal de las proteínas con su ADN diana.
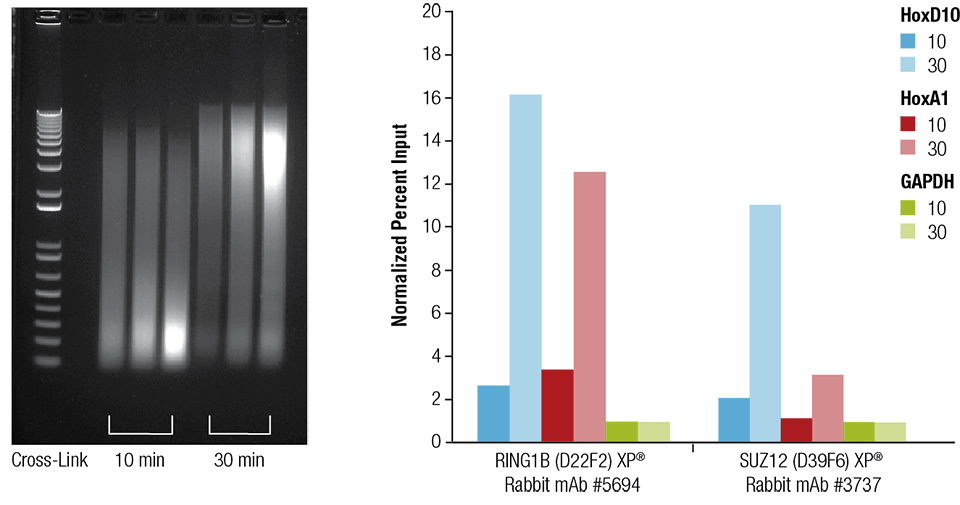
El corazón de ratón (H), el cerebro (B) y el hígado (L) se entrecruzaron durante 10 o 30 minutos según se indica (panel izquierdo). La cromatina se preparó y se sonicó durante 4 minutos. La ChIP se realizó utilizando la cromatina preparada a partir del tejido cardíaco con los anticuerpos indicados utilizando el kit SimpleChIP® Plus Sonication Chromatin IP Kit #56383 y el ADN enriquecido se cuantificó mediante PCR en tiempo real utilizando cebadores para los genes indicados (panel derecho). La cantidad de ADN inmunoprecipitado en cada muestra se representa como señal normalizada con respecto al loci negativo GAPDH, que es igual a 1.
Corazón de ratón (H), cerebro (B) e hígado (L) fueron reticulados durante 10 o 30 minutos según se indica (panel izquierdo). La cromatina se preparó y se sonicó durante 4 minutos. La ChIP se realizó utilizando la cromatina preparada a partir del tejido cardíaco con los anticuerpos indicados utilizando el kit SimpleChIP® Plus Sonication Chromatin IP Kit #56383 y el ADN enriquecido se cuantificó mediante PCR en tiempo real utilizando cebadores para los genes indicados (panel derecho). La cantidad de ADN inmunoprecipitado en cada muestra se representa como señal normalizada al loci negativo GAPDH, que es igual a 1.
6.2 ¿Cómo se fragmenta la cromatina?
La fragmentación de la cromatina es esencial para el éxito de un experimento de ChIP. La fragmentación de la cromatina es necesaria para solubilizar la cromatina y permite su coprecipitación. Además, la resolución del ensayo de ChIP depende de la fragmentación de la cromatina, ya que el tamaño de los fragmentos de ADN determina la resolución del ensayo de ChIP.
La digestión enzimática utiliza la nucleasa micrococcal (MNasa), que escinde el ADN de doble cadena entre los nucleosomas para generar fragmentos de cromatina. Mientras que una digestión completa con MNasa genera fragmentos de ADN de 150 pares de bases (mononucleosomas), una digestión incompleta genera fragmentos de ADN entre 150 y 750 pares de bases (mononucleosomas, dicucleosomas y trinucleosomas). La sonicación utiliza la fuerza mecánica para fragmentar la cromatina. La sonicación fragmenta la cromatina entre y dentro de los nucleosomas, generando un rango de fragmentos de cromatina entre 150 y 1000 pares de bases.
Para el X-ChIP, se utiliza la digestión enzimática o la sonicación para cizallar la cromatina. Las condiciones de sonicación en el protocolo de ChIP por sonicación deben determinarse empíricamente, ya que difieren según el tipo de célula y la condición experimental. Las condiciones de digestión son más consistentes en diferentes tipos de células y tejidos, pero el tamaño de los fragmentos de cromatina debe analizarse antes de la IP.
Para la N-ChIP, se utilizan nucleasas para fragmentar la cromatina con el fin de mantener la unión de proteínas en las muestras no fijadas. La fragmentación con nucleasas también debe determinarse empíricamente para minimizar la sobredigestión de la cromatina.
6.3 ¿Por qué utilizar la digestión enzimática para el ChIP?
La digestión con nucleasas debe utilizarse para el N-ChIP ya que las proteínas no están reticuladas al ADN y las duras condiciones asociadas a la fragmentación basada en la sonicación darían lugar a la disociación de las proteínas de la cromatina del ADN. La N-ChIP es ideal para analizar las interacciones histona-proteína-ADN, ya que la unión histona-ADN es muy fuerte y estable. Sin embargo, la N-ChIP no funciona bien para el análisis de la unión de factores de transcripción y cofactores a la cromatina.
Para fragmentar la cromatina en la X-ChIP se puede utilizar la digestión enzimática o la sonicación. Las ventajas de la digestión enzimática incluyen la consistencia de la fragmentación y unas condiciones de fragmentación suaves (menos calor y detergente) que preservan mejor la integridad de la cromatina y los epítopos de los anticuerpos, lo que resulta en un mayor enriquecimiento inmunológico de la cromatina unida al factor de transcripción y al cofactor.
6.4 ¿Por qué utilizar la sonicación para fragmentar la cromatina para ChIP?
A diferencia de la fragmentación de la cromatina que se consigue mediante la digestión enzimática, la sonicación se basa en fuerzas mecánicas para fragmentar la cromatina en trozos más pequeños. El tamaño ideal de los fragmentos de cromatina para el enriquecimiento inmunológico es de entre 200 y 1000 pares de bases. La sonicación es el método tradicional utilizado para fragmentar la cromatina y puede realizarse utilizando un sonicador de sonda tradicional o sonicadores de baño de agua más avanzados que proporcionan una sonicación más focalizada. La sonicación genera fragmentos de cromatina verdaderamente aleatorios; sin embargo, requiere una amplia optimización en diferentes líneas celulares y tejidos y es difícil de reproducir de un experimento a otro. El requerimiento de altos tampones detergentes y el calor generado durante la sonicación pueden dañar la integridad tanto de la cromatina como de los epítopos de anticuerpos en las proteínas de la cromatina.
6.5 Optimización de la sonicación de la cromatina para ChIP
La fragmentación de la cromatina basada en la sonicación utiliza tradicionalmente altos tampones detergentes y genera calor, y ambos pueden dañar la integridad de la cromatina y de los epítopos de anticuerpos. Por lo tanto, la cantidad de sonicación utilizada para fragmentar la cromatina debe determinarse experimentalmente para diferentes líneas celulares y tejidos. Se debe identificar y utilizar la cantidad mínima de sonicación requerida para generar fragmentos de ADN de 150 a 1000 pares de bases para minimizar el daño a la cromatina.
Antes de embarcarse en un ensayo ChIP completo con análisis posterior por qPCR, chip de ADN o NGS, se debe utilizar la electroforesis en gel para analizar las muestras de cromatina sonicadas durante varios tiempos. El tamaño del fragmento depende del tiempo de sonicación: el tamaño del fragmento disminuye a medida que aumenta el tiempo de sonicación. Sin embargo, los datos sugieren que los tiempos de sonicación más largos no conducen a mejores resultados. Por lo tanto, la ejecución del ADN inmunoprecipitado purificado en un gel y la determinación del tamaño ideal del fragmento es un método sencillo para determinar la cantidad mínima de sonicación necesaria para el tamaño deseado del ADN y para evitar daños innecesarios a la cromatina.

La cromatina digerida por la enzima se ejecutó en un gel de agarosa. El carril 1 muestra la cromatina poco digerida. El carril 2 muestra la cromatina correctamente digerida, y el carril 3 muestra la cromatina sobredigerida.
La cromatina digerida por enzimas se corrió en un gel de agarosa. El carril 1 muestra la cromatina poco digerida. El carril 2 muestra la cromatina correctamente digerida, y el carril 3 muestra la cromatina sobredigerida.
6.6 ¿Cómo se elige un anticuerpo para ChIP?
La elección de un anticuerpo apropiado para un experimento de ChIP es fundamental para su éxito. Los anticuerpos utilizados en un experimento de ChIP deben ser específicos para la proteína de interés y tener una alta afinidad por el antígeno. La mejor elección de anticuerpo para un experimento de ChIP o ChIP-seq es un anticuerpo validado para ChIP o ChIP-seq. Si no se dispone de un anticuerpo validado para ChIP para el gen de interés, la siguiente mejor opción es un anticuerpo que haya sido validado en IP. Es importante tener en cuenta que no todos los anticuerpos validados para IP funcionan en ChIP y no todos los anticuerpos validados para ChIP funcionan en ChIP-seq. Además, cuanto más se valide un anticuerpo en otras aplicaciones, como western, IP, IF, flow e IHC, más confianza se tendrá en el rendimiento y la especificidad del anticuerpo. La validación de un anticuerpo debe ir seguida de la determinación empírica de la concentración óptima del anticuerpo, junto con las condiciones de lavado de IP.
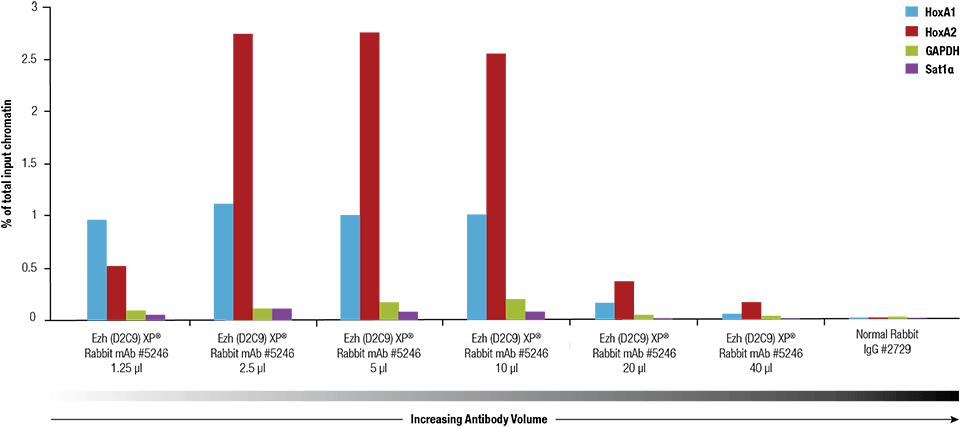
Ezh2 (D2C9) XP® Rabbit mAb #5246 se tituló utilizando el SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 en cromatina reticulada preparada a partir de 4 x 106 células NCCIT.
Ezh2 (D2C9) XP® Rabbit mAb #5246 se tituló utilizando el SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 en cromatina reticulada preparada a partir de 4 x 106 células NCCIT.
6.7 Cómo realizar la inmunoprecipitación
Los anticuerpos se utilizan para capturar una proteína de interés y su ADN unido. La concentración de anticuerpos debe determinarse empíricamente, con un punto de partida general de 0,5-2,0 μg de anticuerpos utilizados por 10 μg de ADN de cromatina (corresponde a aproximadamente 4 x 106 células). La rigurosidad de los tampones y los tiempos de lavado también deben determinarse empíricamente, ya que dependen de la afinidad del anticuerpo con su antígeno diana. Normalmente, las incubaciones anticuerpo:cromatina se realizan durante 2 horas o toda la noche.
Los complejos anticuerpo-antígeno (+ADN) se capturan por afinidad en una resina de unión a anticuerpos. En los experimentos de ChIP esta resina se compone típicamente de perlas magnéticas, de sefarosa o de agarosa de grado ChIP conjugadas con proteína A y/o proteína B. Los anticuerpos se unen a las perlas de proteína A y/o proteína G con varias afinidades dependiendo de la especie en la que se desarrollaron y del subtipo IgG de su cadena pesada. Las perlas se incuban típicamente con anticuerpo:cromatina durante 2 a 4 horas.
Se necesitan pasos de lavado para eliminar la cromatina no unida al anticuerpo, seguidos de la inversión de los enlaces cruzados (para X-ChIP) y la purificación del ADN. Además, debe realizarse una IP de control de IgG para determinar el fondo (señal:ruido). También deben incluirse anticuerpos de control positivo (es decir, histona H3 total) y/o cebadores qPCR de control positivo (para loci de unión de proteínas diana positivos y negativos conocidos) para determinar la unión no específica. Para obtener resultados óptimos, el control de calidad de la cromatina IP por qPCR debe realizarse antes del análisis NGS posterior.
6.8 Cómo eluir la cromatina de las perlas de proteína A/G
La cromatina se eluye de las perlas de proteína A/G utilizando detergente y calor. Se requiere un «vortex» o una mezcla a baja velocidad para mantener las perlas en suspensión y aumentar la elución de la cromatina.
6.9 Cómo invertir los enlaces cruzados de la cromatina
Los enlaces cruzados se invierten mediante calor y sal elevados (ambos son componentes vitales). También se añade proteinasa K para digerir las proteínas de la cromatina asociadas y se añaden anticuerpos, lo que permite una purificación más eficiente del ADN aguas abajo.
6.10 Cómo purificar el ADN
Después de eliminar los enlaces cruzados de la cromatina, el ADN se purifica utilizando los métodos clásicos de fenol-cloroformo seguidos de precipitación con etanol o utilizando kits de purificación de ADN en columna.
Cómo analizar el ADN enriquecido
Una vez purificado el ADN, se pueden realizar varios análisis posteriores, incluyendo ChIP-PCR, ChIP-qPCR, ChIP-chip y ChIP-seq.
7.1 Análisis de ChIP-PCR y ChIP-qPCR
Los análisis de ChIP-PCR y ChIP-qPCR son los mejores para el análisis de un solo gen y pueden utilizarse para amplificar y cuantificar fragmentos específicos de ADN de forma rápida y rentable.
7.2 Análisis ChIP-chip
El análisis ChIP-chip utiliza chips de microarrays de ADN en mosaico para crear un mapa de alta resolución de la unión de proteínas y de la modificación de proteínas en todo el genoma.
7.3 Análisis ChIP-seq
El análisis ChIP-seq utiliza la tecnología NGS estándar para alinear el ADN purificado con genomas completos previamente anotados para identificar perfiles de unión de proteínas en todo el genoma.