Ce este ChIP?
Imunoprecipitarea cromatinei, sau ChIP, este o tehnologie bazată pe anticorpi utilizată pentru a îmbogăți selectiv proteine specifice de legare a ADN-ului împreună cu țintele lor de ADN. ChIP este utilizată pentru a investiga o anumită interacțiune proteină-ADN, mai multe interacțiuni proteină-ADN sau interacțiuni la nivelul întregului genom sau al unui subset de gene.
ChIP utilizează anticorpi care recunosc și se leagă selectiv de proteine, inclusiv histone, modificări histonice, factori de transcripție și cofactori, pentru a furniza informații despre stările cromatinei și transcripția genelor. O combinație între analiza proteomică și tehnicile de biologie moleculară utilizate în ChIP permite capacitatea de a înțelege expresia și reglarea genelor în celulele sau țesuturile de interes.
Când se utilizează ChIP?
În mod obișnuit, ChIP este utilizată pentru a identifica abundența relativă a unei proteine specifice sau a unei modificări proteice specifice într-o anumită regiune a genomului. ChIP poate fi utilizată pentru a răspunde la o multitudine de întrebări științifice care implică interacțiunea dintre proteine și cromatină. De exemplu, ChIP poate fi utilizată pentru a compara prezența anumitor proteine la diferiți loci, pentru a cartografia diferitele proteine într-o regiune genomică de interes sau pentru a cuantifica legarea proteinelor la o genă inductibilă ca răspuns la un stimul în timp.
Cum funcționează ChIP?
Principiul din spatele ChIP este relativ simplu și se bazează pe utilizarea unui anticorp pentru a izola, sau precipita, o anumită proteină, histonă, factor de transcripție sau cofactor și cromatina legată de acesta dintr-un amestec de proteine care a fost extras din celule sau țesuturi. De aici și numele tehnicii: Imunoprecipitarea cromatinei. În ChIP-PCR sau ChIP-seq, fragmentele de ADN îmbogățite imunitar pot fi apoi identificate și cuantificate folosind reactivi PCR sau qPCR disponibili pe scară largă și tehnologii de secvențiere de generație următoare (NGS).
Ce este ChIP nativ (N-ChIP) vs ChIP reticulat (X-ChIP)?
Există 2 tipuri de tehnici ChIP care pot fi efectuate în funcție de întrebarea experimentală și de materialul de plecare pentru experiment: 1) ChIP nativ (N-ChIP) și 2) ChIP reticulat (X-ChIP). Ambele tipuri de ChIP au avantaje și dezavantaje:
- În N-ChIP, nu se utilizează niciun agent de fixare pentru a reticula proteinele de cromatină. În schimb, cromatina nativă este izolată din nuclee celulare care sunt digerate cu o nuclează. Deoarece anticorpii sunt crescuți împotriva antigenelor nefixate, N-ChIP oferă avantajul unei mai bune recunoașteri și legări a anticorpilor la antigenele lor țintă. Este posibil să nu fie necesară PCR pentru analiza în aval datorită abundenței ridicate a proteinelor histonice. Deși aceste avantaje fac din N-ChIP o metodă atractivă, aceasta poate fi utilizată doar pentru detectarea histonelor. În plus, pierderea legăturii proteinei în timpul etapelor de digestie a cromatinei și de imunoprecipitare poate distorsiona datele sau împiedica analizele corespunzătoare.
- În X-ChIP, se folosesc fixatori chimici, cum ar fi formaldehida, pentru a reticula proteina de interes la ADN, iar fragmentarea cromatinei se realizează prin sonicare sau digestie cu nucleaze. Avantajul X-ChIP este că poate fi utilizat cu proteine histone și non-histone și, în general, necesită mai puțin material celular de plecare decât N-ChIP. X-ChIP minimizează, de asemenea, șansele de pierdere a proteinelor din cromatină în timpul extracției, permițând detectarea interacțiunilor proteice tranzitorii. Cu toate acestea, etapa de precipitare este mai puțin eficientă și este necesară amplificarea ADN-ului prin PCR pentru analizele din aval.
Care sunt diferitele tipuri de teste ChIP?
După ce imunoprecipitarea cromatinei în sine este finalizată, pot fi efectuate mai multe analize în aval asupra cromatinei purificate și a proteinelor asociate, histone, factori de transcripție și cofactori. Cele mai comune metode pentru analiza unei singure gene și pentru analiza întregului genom sunt qPCR și, respectiv, ChIP-seq. PCR și ChIP-chip sunt, de asemenea, opțiuni pentru analiza în aval.
5.1 Care sunt avantajele ChIP-PCR?
ChIP-PCR se realizează pentru a analiza modificările histonice și/sau legarea proteinelor la un subset cunoscut de loci țintă din genom. În ChIP-PCR, fragmentele de ADN îmbogățite imunitar sunt identificate și cuantificate cu ajutorul reactivilor și tehnologiilor PCR sau qPCR disponibile pe scară largă. Cu ajutorul ChIP-qPCR se pot realiza comparații rapide și cantitative ale unor regiuni specifice din cadrul genomului în mai multe probe. Aceasta este mai ieftină și mai eficientă din punct de vedere al timpului decât metodele de secvențiere a întregului genom.
5.2 Care sunt avantajele ChIP-chip?
Tehnologia ChIP-chip se referă la utilizarea unui cip de microrețea de ADN pentru a analiza fragmente de ADN îmbogățite cu ChIP-imunitate. Utilizarea tehnologiei genome tiling microarray permite o analiză a întregului genom al proteinelor care sunt legate de ADN izolat și generează o hartă genomică de înaltă rezoluție a legăturilor cu proteinele și a modificărilor proteice. ChIP-chip are utilizări multiple în cercetarea de bază, precum și în cercetarea bazată pe boli. De exemplu, poate fi utilizat pentru a identifica situsurile de legare a factorilor de transcripție, a intensificatorilor și a reprimatorilor și pentru a compara aceste tipuri de proteine legate în eșantioane de control și patologice. Cu toate acestea, deoarece costul NGS a scăzut substanțial și se obțin rezultate similare folosind ChIP-seq, tot mai multe persoane aleg să efectueze ChIP-seq în locul ChIP-chip.
5.3 Care sunt avantajele ChIP-seq?
Similar cu ChIP-chip, ChIP-seq oferă informații despre legarea proteinelor la nivelul întregului genom. Cu toate acestea, spre deosebire de ChIP-chip, ChIP-seq utilizează tehnologia NGS pentru a identifica fragmente de ADN și pentru a le cartografia în raport cu întregul genom.
Tehnologia mai contemporană de amplificare a ADN-ului permite efectuarea unor analize robuste în câteva zile, cu o cantitate redusă de ADN de intrare. Atunci când materialul de plecare este rar, aceste progrese tehnologice în metodele de pregătire a bibliotecilor au făcut posibile experimentele ChIP-seq.
În plus, noua tehnologie în care probele de ADN sunt marcate în mod unic cu secvențe scurte, cunoscute sub numele de coduri de bare, permite acum ca fragmentele individuale să fie grupate într-un singur culoar de secvențiere pentru analize multiplexate. Acest lucru a crescut substanțial eficiența și a scăzut costul experimentelor de secvențiere a ADN-ului, sprijinind și mai mult aplicațiile ChIP-seq.
După toate acestea, datorită progreselor în tehnologia de secvențiere a ADN-ului, avantajul ChIP-seq-ului este că un număr mare de probe de ADN îmbogățit cu ChIP poate fi secvențiat ieftin într-o perioadă relativ scurtă de timp, cu o sensibilitate și o precizie mai mare decât ChIP-chip.
Care sunt diferitele etape ale testului ChIP?
Sesizările ChIP urmează un protocol general:
- Încrucișarea proteinelor cu ADN doar pentru X-ChIP
- Liza celulară
- Fragmentarea cromatinei prin digestie (pentru X-ChIP și N-ChIP) sau sonicare-.(numai pentru X-ChIP)
- Imunoprecipitare cu ajutorul anticorpilor specifici
- Curățarea ADN-ului pentru analizele din aval
- Analiza ADN-ului prin PCR, qPCR, microarray sau NGS
Important, controalele pozitive și negative la fiecare etapă sunt esențiale pentru a determina dacă un experiment ChIP a fost un succes.
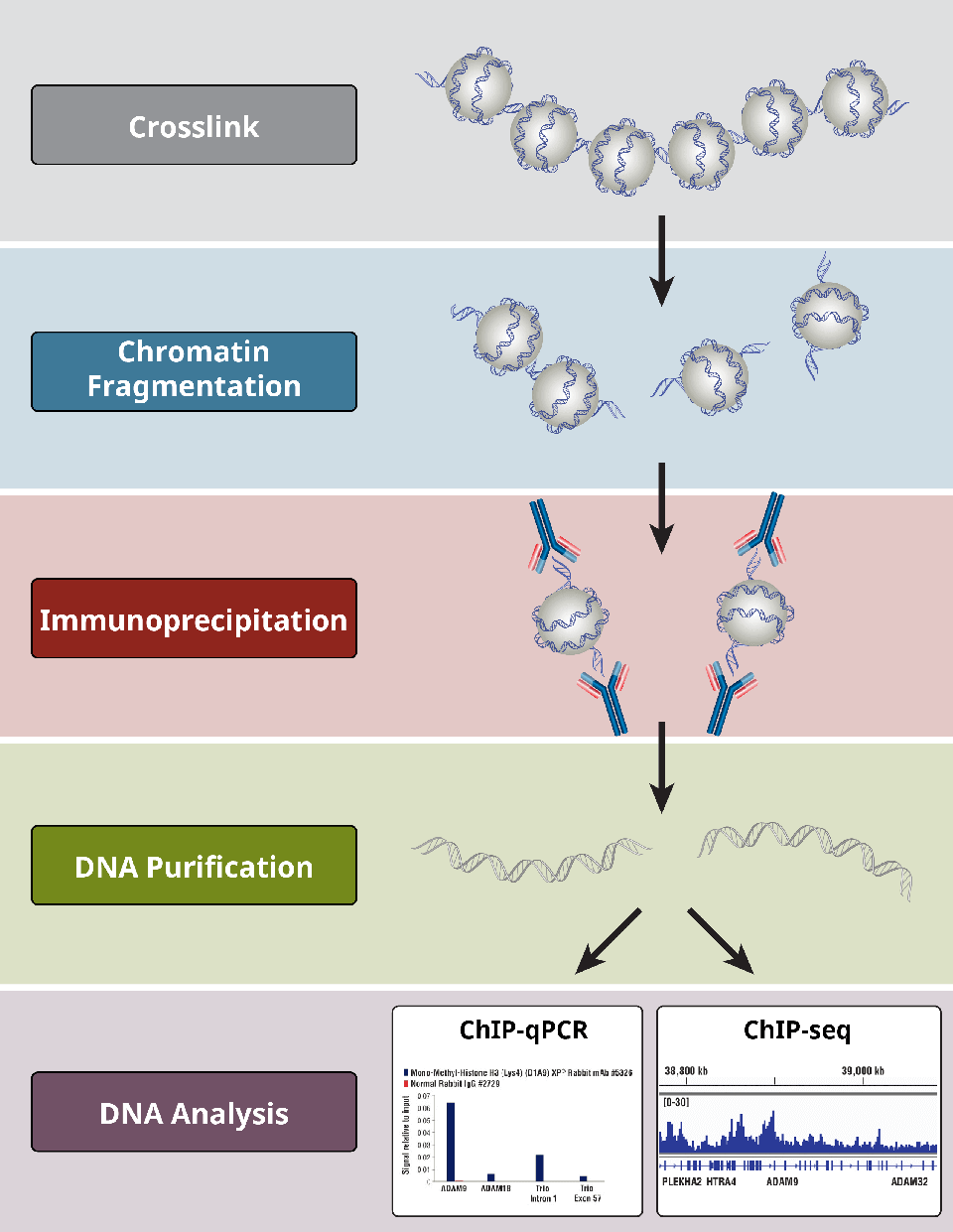
Vizualizare generală a celor mai critice etape ale unui protocol ChIP.
Vizualizare generală a celor mai critice etape ale unui protocol ChIP.
6.1 Cum se reticulează celulele și țesuturile pentru ChIP?
Reactivii de reticulare sunt utilizați pentru a „fixa” proteinele de ADN-ul pe care acestea îl leagă. Reactivii pe bază de formaldehidă sunt utilizați de obicei pentru a realiza această fixare. Celulele și țesuturile sunt, în general, fixate într-un mod similar, dar țesuturile necesită un timp de fixare mai lung și o livrare mai rapidă a fixării pentru a pătrunde rapid în țesutul țintă înainte ca acesta să înceapă să degenereze.
Fixarea excesivă a cromatinei poate reduce eficiența fragmentării prin sonicare, pe lângă faptul că inhibă legarea anticorpilor la țintele lor proteice. Prin urmare, timpul de fixare ar trebui să fie determinat empiric pentru a permite o legare maximă a anticorpului-antigen și, în același timp, pentru a obține o reticulare ideală a proteinelor la ADN-ul lor țintă.
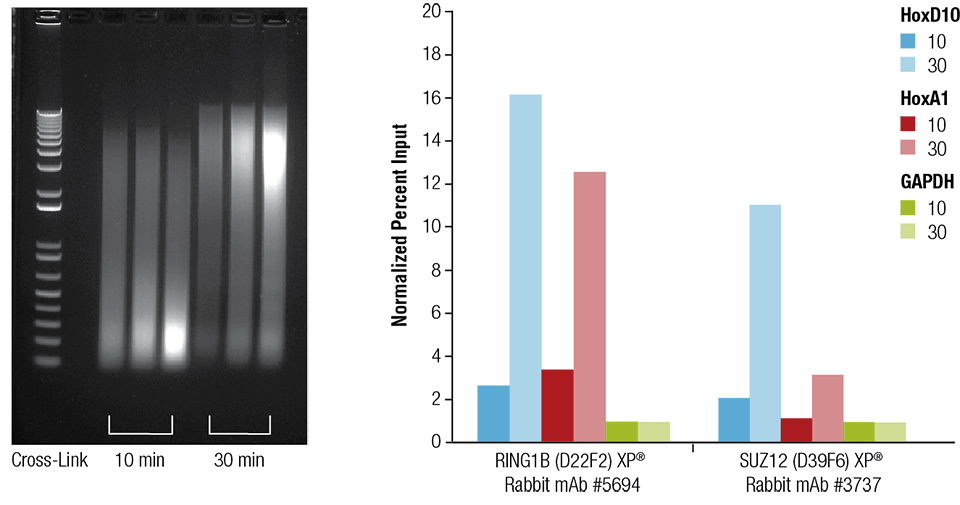
Inima de șoarece (H), creierul (B) și ficatul (L) au fost reticulate timp de 10 sau 30 de minute, după cum este indicat (panoul din stânga). Cromatina a fost preparată și sonicată timp de 4 minute. ChIP a fost efectuată folosind cromatina preparată din țesutul cardiac cu anticorpii indicați, utilizând kitul SimpleChIP® Plus Sonication Chromatin IP Kit #56383, iar ADN-ul îmbogățit a fost cuantificat prin PCR în timp real utilizând primeri pentru genele indicate (panoul din dreapta). Cantitatea de ADN imunoprecipitat în fiecare probă este reprezentată ca semnal normalizat în raport cu lociul negativ GAPDH, care este egal cu 1.
Carda de șoarece (H), creierul (B) și ficatul (L) au fost reticulate timp de 10 sau 30 de minute, după cum este indicat (panoul din stânga). Cromatina a fost preparată și sonicată timp de 4 minute. ChIP a fost efectuată folosind cromatina preparată din țesutul cardiac cu anticorpii indicați, utilizând kitul SimpleChIP® Plus Sonication Chromatin IP Kit #56383, iar ADN-ul îmbogățit a fost cuantificat prin PCR în timp real utilizând primeri pentru genele indicate (panoul din dreapta). Cantitatea de ADN imunoprecipitat în fiecare probă este reprezentată ca semnal normalizat față de lociul negativ GAPDH, care este egal cu 1.
6.2 Cum se fragmentează cromatina?
Fragmentarea cromatinei este esențială pentru succesul unui experiment ChIP. Fragmentarea cromatinei este necesară pentru a solubiliza cromatina și permite coprecipitarea acesteia. În plus, rezoluția testului ChIP depinde de fragmentarea cromatinei, deoarece dimensiunea fragmentelor de ADN determină rezoluția testului ChIP.
Digestia enzimatică utilizează nuclează micrococală (MNase), care clivează ADN bicatenar între nucleozomi pentru a genera fragmente de cromatină. În timp ce o digestie completă cu MNază generează fragmente de ADN de 150 de perechi de baze (mononucleozomi), o digestie incompletă generează fragmente de ADN între 150 și 750 de perechi de baze (mono-, di- și tri-nucleozomi). Sonicare utilizează forța mecanică pentru a fragmenta cromatina. Sonicația fragmentează cromatina între și în interiorul nucleozomilor, generând o gamă de fragmente de cromatină între 150 și 1000 de perechi de baze.
Pentru X-ChIP, se utilizează fie digestia enzimatică, fie sonicația pentru a forfeca cromatina. Condițiile de sonicare în protocolul ChIP prin sonicare ar trebui să fie determinate empiric, deoarece acestea diferă în funcție de tipul de celule și de condițiile experimentale. Condițiile de digestie sunt mai consecvente între diferite tipuri de celule și țesuturi, dar dimensiunea fragmentului de cromatină trebuie totuși analizată înainte de IP.
Pentru N-ChIP, se utilizează nucleaze pentru a fragmenta cromatina pentru a menține legarea proteinei în probele nefixate. Fragmentarea cu nucleaze ar trebui, de asemenea, să fie determinată empiric pentru a minimiza supradigestia cromatinei.
6.3 De ce să se utilizeze digestia enzimatică pentru ChIP?
Digestia cu nucleaze trebuie utilizată pentru N-ChIP deoarece proteinele nu sunt reticulate la ADN și condițiile dure asociate cu fragmentarea bazată pe sonicare ar duce la disocierea proteinelor de cromatină de ADN. N-ChIP este ideală pentru analiza interacțiunilor dintre proteinele histonice și ADN, deoarece legătura histonă-ADN este foarte puternică și stabilă. Cu toate acestea, N-ChIP nu funcționează bine pentru analiza legăturii de cromatină a factorilor de transcripție și a cofactorilor.
Pentru fragmentarea cromatinei în X-ChIP se poate utiliza fie digestia enzimatică, fie sonicația. Beneficiile digestiei enzimatice includ consistența fragmentării și condițiile blânde de fragmentare (mai puțină căldură și detergent) care păstrează mai bine integritatea cromatinei și a epitopilor anticorpilor, ceea ce duce la o mai mare îmbogățire imunologică a cromatinei legate de factorii de transcripție și cofactorii.
6.4 De ce să se utilizeze sonicare pentru fragmentarea cromatinei pentru ChIP?
În comparație cu fragmentarea cromatinei realizată prin digestie enzimatică, sonicare se bazează pe forțe mecanice pentru a fragmenta cromatina în bucăți mai mici. Dimensiunea ideală a fragmentelor de cromatină pentru îmbogățirea imună este între 200 și 1000 de perechi de baze. Sonicarea este metoda tradițională utilizată pentru fragmentarea cromatinei și poate fi realizată cu ajutorul unui sonicator tradițional cu sondă sau a unor sonicatoare cu baie de apă mai avansate care asigură o sonicare mai concentrată. Sonicarea generează fragmente de cromatină cu adevărat randomizate; cu toate acestea, necesită o optimizare extensivă pe diferite linii celulare și țesuturi și este dificil de reprodus de la un experiment la altul. Necesitatea unor tampoane cu conținut ridicat de detergenți și căldura generată în timpul sonicării pot afecta atât integritatea cromatinei, cât și epitopii anticorpilor de pe proteinele de cromatină.
6.5 Optimizarea sonicării cromatinei pentru ChIP
Fragmentarea cromatinei pe bază de sonicare utilizează în mod tradițional tampoane cu conținut ridicat de detergenți și generează căldură, ambele putând afecta integritatea cromatinei și a epitopilor anticorpilor. Prin urmare, cantitatea de sonicare utilizată pentru fragmentarea cromatinei trebuie să fie determinată experimental pentru diferite linii celulare și țesuturi. Trebuie să se identifice și să se utilizeze cantitatea minimă de sonicare necesară pentru a genera fragmente de ADN de 150 până la 1000 de perechi de baze pentru a minimiza deteriorarea cromatinei.
Înainte de a se lansa într-un test ChIP complet cu analiză în aval prin qPCR, cip de ADN sau NGS, ar trebui să se utilizeze electroforeza pe gel pentru a analiza eșantioanele de cromatină sonicate pentru diferite perioade de timp. Dimensiunea fragmentelor depinde de timpul de sonicare – dimensiunea fragmentelor scade pe măsură ce crește timpul de sonicare. Cu toate acestea, datele sugerează că un timp de sonicare mai lung nu duce la rezultate mai bune. Prin urmare, rularea ADN-ului purificat imunoprecipitat pe un gel și determinarea dimensiunii ideale a fragmentului este o metodă simplă pentru a determina cantitatea minimă de sonicare necesară pentru dimensiunea dorită a ADN-ului și pentru a evita deteriorarea inutilă a cromatinei.

Cromatina digerată enzimatic a fost rulată pe un gel de agaroză. Banda 1 arată cromatina care este subdigerată. Banda 2 arată cromatina corect digerată, iar banda 3 arată cromatina care este supradigerată.
Cromatina digerată enzimatic a fost rulată pe un gel de agaroză. Banda 1 arată cromatina care este subdigerată. Banda 2 arată cromatina corect digerată, iar banda 3 arată cromatina care este supradigerată.
6.6 Cum se alege un anticorp pentru ChIP?
Alegerea unui anticorp adecvat pentru un experiment ChIP este esențială pentru succesul acestuia. Anticorpii utilizați într-un experiment ChIP trebuie să fie specifici pentru proteina de interes și să aibă o afinitate ridicată pentru antigen. Cea mai bună alegere de anticorp pentru un experiment ChIP sau ChIP-seq este un anticorp validat pentru ChIP sau ChIP-seq. În cazul în care nu există un anticorp validat ChIP pentru gena de interes, cea mai bună alegere este un anticorp care a fost validat în IP. Este important de reținut că nu toți anticorpii validați IP funcționează în ChIP și nu toți anticorpii validați ChIP funcționează în ChIP-seq. În plus, cu cât un anticorp este mai mult validat în alte aplicații, cum ar fi western, IP, IF, IF, flux și IHC, cu atât mai multă încredere se poate avea în performanța și specificitatea anticorpului. Validarea unui anticorp trebuie să fie urmată de determinarea empirică a concentrației optime a anticorpului, împreună cu condițiile de spălare IP.
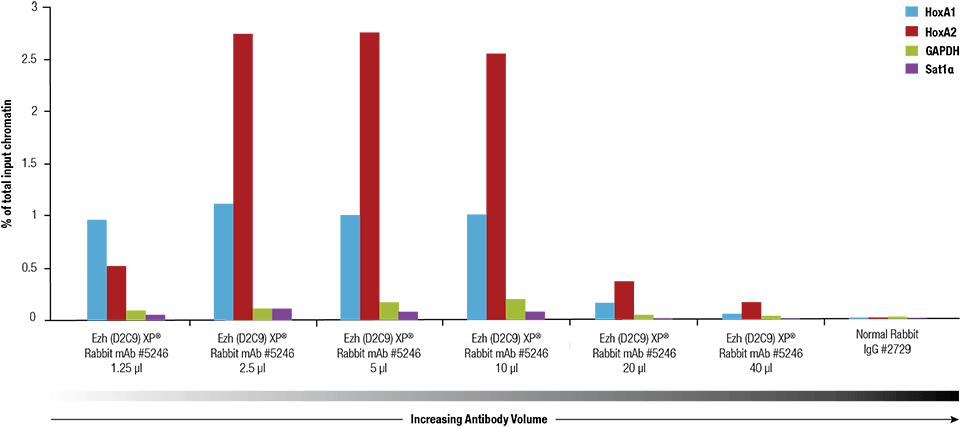
Ezh2 (D2C9) XP® Rabbit mAb #5246 a fost titrat folosind SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 pe cromatină reticulată preparată din 4 x 106 celule NCCIT.
Ezh2 (D2C9) XP® Rabbit mAb #5246 a fost titrat folosind SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 pe cromatină reticulată preparată din 4 x 106 celule NCCIT.
6.7 Cum se realizează imunoprecipitarea
Anticorpii sunt utilizați pentru a capta o proteină de interes și ADN-ul său legat. Concentrația anticorpilor trebuie determinată empiric, cu un punct de plecare general de 0,5-2,0 μg de anticorp utilizat la 10 μg de ADN de cromatină (corespunde la aproximativ 4 x 106 celule). Rigurozitatea tampoanelor și timpii de spălare trebuie, de asemenea, determinate empiric, deoarece acestea depind de afinitatea anticorpului față de antigenul său țintă. În mod obișnuit, incubările anticorp:cromatină se fac timp de 2 ore până la o noapte.
Complecșii anticorp-antigen (+ADN) sunt capturați prin afinitate pe o rășină de legare a anticorpilor. În experimentele ChIP, această rășină este compusă, de obicei, din perle magnetice, de sefaroză sau de agaroză de calitate ChIP conjugate cu proteina A și/sau proteina B. Anticorpii se leagă de perlele de proteină A și/sau de proteină G cu diferite afinități, în funcție de specia în care au fost dezvoltați și de subtipul IgG al lanțului lor greu. Bilele sunt de obicei incubate cu anticorp:cromatină timp de 2 până la 4 ore.
Sunt necesare etape de spălare pentru a elimina cromatina nelegată de anticorpi, urmate de inversarea legăturilor încrucișate (pentru X-ChIP) și purificarea ADN-ului. În plus, trebuie efectuată o IP de control IgG pentru a determina fondul (semnal:zgomot). Trebuie, de asemenea, să se includă anticorpi de control pozitiv (de exemplu, histona H3 totală) și/sau primeri qPCR de control pozitiv (pentru loci de legare a proteinelor țintă pozitive și negative cunoscute) pentru a determina legarea nespecifică. Pentru rezultate optime, QC al cromatinei IP prin qPCR trebuie efectuat înainte de analiza NGS din aval.
6.8 Cum se eluează cromatina de pe bilele de proteină A/G
Cromatina se eluează de pe bilele de proteină A/G folosind detergent și căldură. Este necesară „vortexarea” sau amestecarea la viteză mică pentru a menține perlele în suspensie și pentru a crește eluția cromatinei.
6.9 Cum se inversează legăturile încrucișate ale cromatinei
Legăturile încrucișate sunt inversate prin căldură mare și sare mare (ambele sunt componente vitale). Se adaugă, de asemenea, proteinaza K pentru a digera proteinele asociate cromatinei și se adaugă anticorpi, permițând o purificare mai eficientă a ADN-ului în aval.
6.10 Cum se purifică ADN-ul
După ce legăturile încrucișate ale cromatinei sunt îndepărtate, ADN-ul este purificat fie folosind metodele clasice de fenol-cloroform urmat de precipitare cu etanol, fie folosind kituri de purificare a ADN-ului pe bază de coloană.
Cum se analizează ADN-ul îmbogățit
După ce ADN-ul este purificat, pot fi efectuate mai multe analize în aval, inclusiv ChIP-PCR, ChIP-qPCR, ChIP-chip și ChIP-seq.
7.1 Analiza ChIP-PCR și ChIP-qPCR
Analizele ChIP-PCR și ChIP-qPCR sunt cele mai bune pentru analiza unei singure gene și pot fi utilizate pentru a amplifica și cuantifica fragmente specifice de ADN într-un mod rapid și eficient din punct de vedere al costurilor.
7.2 Analiza ChIP-chip
Analiza ChIP-chip utilizează cipuri de microrețele de ADN de tip „tiling” pentru a crea o hartă de înaltă rezoluție a legării și modificării proteinelor la nivelul întregului genom.
7.3 Analiza ChIP-seq
Analiza ChIP-seq utilizează tehnologia standard NGS pentru a alinia ADN-ul purificat cu genomurile întregi adnotate anterior pentru a identifica profilurile de legare a proteinelor la nivelul întregului genom.