O que é ChIP?
Imunoprecipitação da cromatina, ou ChIP, é uma tecnologia baseada em anticorpos utilizada para enriquecer selectivamente proteínas específicas de ligação ao ADN juntamente com os seus alvos de ADN. O ChIP é usado para investigar uma interação proteína-DNA particular, várias interações proteína-DNA, ou interações através de todo o genoma ou um subconjunto de genes.
ChIP utiliza anticorpos que reconhecem e ligam seletivamente proteínas, incluindo histones, modificações de histone, fatores de transcrição e cofatores, para fornecer informações sobre estados de cromatina e transcrição de genes. Uma combinação de análise proteômica e técnicas de biologia molecular usadas no ChIP permite a capacidade de compreender a expressão e regulação gênica em células ou tecidos de interesse.
Quando é usado o ChIP?
Tipicamente, o ChIP é usado para identificar a abundância relativa de uma proteína específica ou uma modificação proteica específica em uma determinada região do genoma. O ChIP pode ser usado para responder a uma série de questões científicas envolvendo a interação de proteínas e cromatina. Por exemplo, a PIQ pode ser usada para comparar a presença de certas proteínas em vários loci, mapear as várias proteínas em uma região genômica de interesse, ou quantificar a ligação de proteínas a um gene induzível em resposta a um estímulo ao longo do tempo.
Como funciona a PIQ?
O princípio por trás do ChIP é relativamente simples e depende do uso de um anticorpo para isolar, ou precipitar, uma certa proteína, histone, fator de transcrição, ou cofator e sua ligação com a cromatina de uma mistura de proteínas que foi extraída de células ou tecidos. Daí, o nome da técnica: Cromatina Imunoprecipitação. No ChIP-PCR ou ChIP-seq, os fragmentos de DNA imuno-enriquecido são então capazes de ser identificados e quantificados usando reagentes PCR ou qPCR amplamente disponíveis e tecnologias Next Generation Sequencing (NGS).
O que é ChIP nativo (N-ChIP) versus ChIP reticulado (X-ChIP)?
Existem 2 tipos de técnicas de ChIP que podem ser realizadas dependendo da questão experimental e do material de partida para o experimento: 1) ChIP nativo (N-ChIP) e 2) ChIP reticulado (X-ChIP). Ambos os tipos de ChIP têm vantagens e desvantagens:
- Em N-ChIP, nenhum agente fixador é usado para reticulação de proteínas à cromatina. Em vez disso, a cromatina nativa é isolada dos núcleos celulares que são digeridos com um nuclease. Como os anticorpos são criados contra antígenos não fixados, o N-ChIP oferece a vantagem de melhor reconhecimento e ligação dos anticorpos aos seus antígenos alvo. A PCR pode não ser necessária para a análise a jusante devido à elevada abundância de proteínas histoníacas. Embora estas vantagens façam da N-ChIP um método atractivo, ela só pode ser utilizada para a detecção de histonas. Além disso, a perda de ligação proteica durante a digestão da cromatina e as etapas de imunoprecipitação podem enviesar os dados ou impedir análises adequadas.
- No X-ChIP, fixadores químicos como o formaldeído são usados para cruzar a proteína de interesse para o DNA e a fragmentação da cromatina é obtida através da sonicação ou digestão por nuclease. A vantagem do X-ChIP é que ele pode ser usado com as proteínas histone e não-histone e geralmente requer menos material de partida celular do que o N-ChIP. O X-ChIP também minimiza as chances de perda de proteína cromatina durante a extração, permitindo a detecção de interações proteicas transitórias. Contudo, a etapa de precipitação é menos eficiente e a amplificação do DNA por PCR é necessária para análises a jusante.
Quais são os diferentes tipos de ensaios de ChIP?
Após a imunoprecipitação da cromatina estar completa, várias análises a jusante podem ser conduzidas sobre a cromatina purificada e as proteínas associadas, histonas, factores de transcrição e cofactores. Os métodos mais comuns para análise de um único gene e análise do genoma inteiro são qPCR e ChIP-seq, respectivamente. PCR e ChIP-chip também são opções para análise downstream.
5.1 Quais são as vantagens do ChIP-PCR?
ChIP-PCR é realizado para analisar modificações de histone e/ou ligação de proteínas a um subconjunto conhecido de loci alvo no genoma. No ChIP-PCR, fragmentos de DNA imuno-enriquecido são identificados e quantificados usando reagentes e tecnologias PCR ou qPCR amplamente disponíveis. Comparações rápidas e quantitativas de regiões específicas dentro do genoma através de múltiplas amostras podem ser obtidas usando o ChIP-qPCR. Isto é mais barato e mais eficiente em termos de tempo do que métodos de sequenciamento de genoma inteiro.
5.2 Quais são as vantagens do ChIP-chip?
A tecnologia ChIP-chip refere-se à utilização de um chip de microarranjo de ADN para analisar fragmentos de ADN enriquecidos com ChIP-immune. O uso da tecnologia de microarranjo do genoma permite uma análise de todo o genoma de proteínas que estão ligadas ao DNA isolado e gera um mapa genômico de alta resolução de ligação de proteínas e modificações protéicas. O ChIP-chip tem múltiplos usos na pesquisa básica, bem como na pesquisa baseada em doenças. Por exemplo, ele pode ser usado para identificar os locais de ligação dos fatores de transcrição, intensificadores e repressores e para comparar esses tipos de proteínas ligadas em amostras de controle e patológicas. Entretanto, como o custo do NGS diminuiu substancialmente e resultados similares são obtidos usando ChIP-seq, mais pessoas estão escolhendo realizar ChIP-seq ao invés de ChIP-chip.
5.3 Quais são as vantagens do ChIP-seq?
Similar ao ChIP-chip, o ChIP-seq fornece informações sobre a ligação de proteínas em todo o genoma. No entanto, ao contrário do ChIP-chip, ChIP-seq usa a tecnologia NGS para identificar fragmentos de DNA e mapeá-los contra todo o genoma.
Outra tecnologia de amplificação de DNA contemporânea permite que análises robustas sejam conduzidas em questão de dias com uma baixa quantidade de DNA de entrada. Quando o material inicial é escasso, estes avanços tecnológicos nos métodos de preparação de bibliotecas tornaram possíveis os experimentos ChIP-seq.
Além disso, a nova tecnologia na qual as amostras de DNA são marcadas exclusivamente com sequências curtas, conhecidas como códigos de barras, agora permite que fragmentos individuais sejam agrupados em uma única pista de seqüenciamento para análise multiplexada. Isso aumentou substancialmente a eficiência e diminuiu o custo dos experimentos de sequenciamento de DNA, suportando ainda mais as aplicações ChIP-seq.
Aparticular, devido aos avanços na tecnologia de sequenciamento de DNA, a vantagem do ChIP-seq é que um grande número de amostras de DNA enriquecidas com ChIP pode ser sequenciado a baixo custo em um período de tempo relativamente curto com maior sensibilidade e precisão do que o ChIP-chip.
Quais são as diferentes etapas do ensaio ChIP?
Os ensaios ChIP seguem um protocolo geral:
- Crosslinking de proteínas ao DNA apenas para X-ChIP
- Lise celular
- Fragmentação da cromatina por digestão (para X-ChIP e N-ChIP) ou sonicação…cisalhamento com base em X-ChIP (apenas para X-ChIP)
- Imunoprecipitação usando anticorpos específicos
- Purificação de ADN para análises a jusante
- Análise de ADN via PCR, qPCR, microarray, ou NGS
Importante, os controles positivos e negativos em cada etapa são parte integrante para determinar se uma experiência ChIP foi bem sucedida.
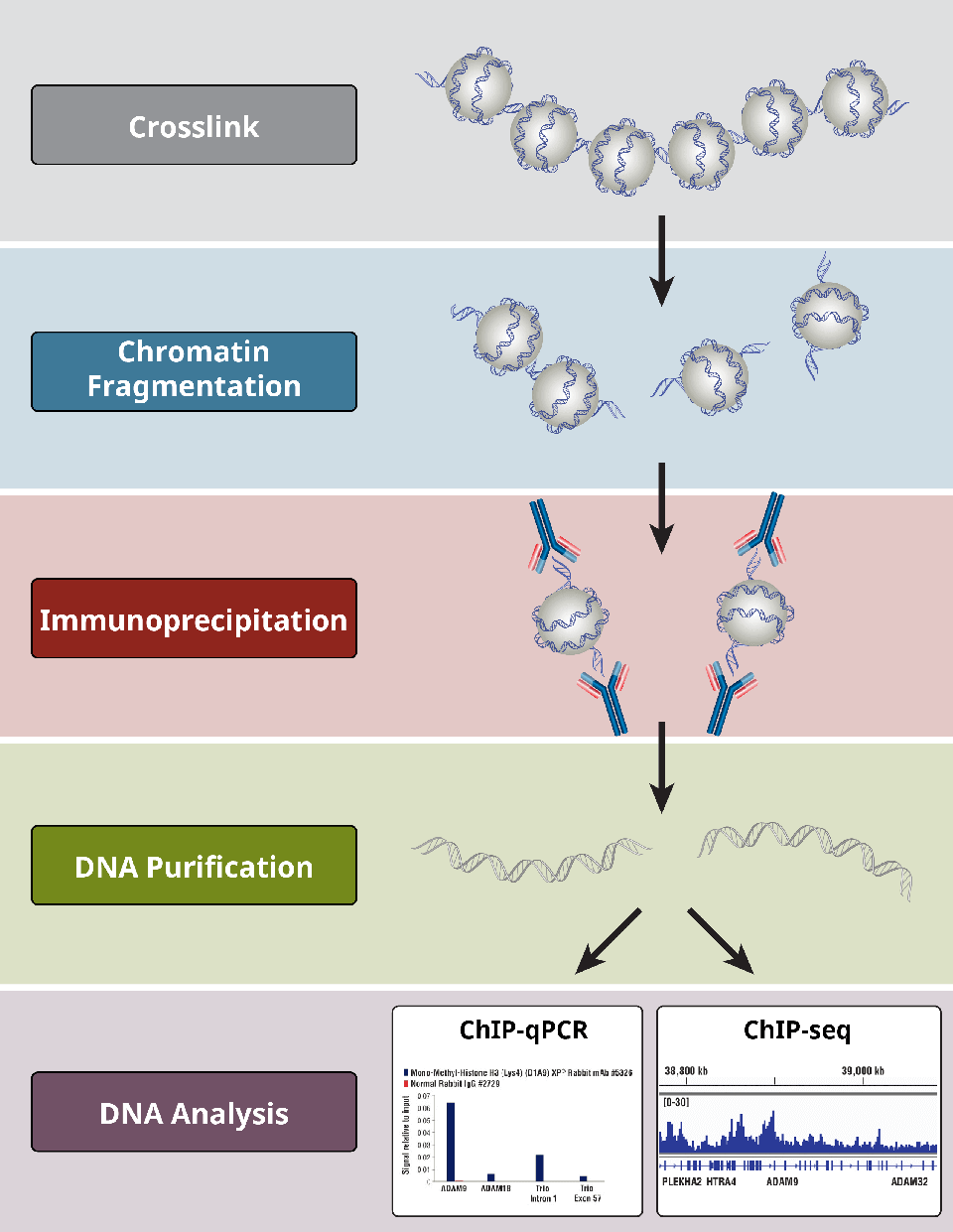
Visão geral dos passos mais críticos de um protocolo ChIP.
Visão geral dos passos mais críticos de um protocolo ChIP.
6.1 Como se interligam células e tecidos para ChIP?
Reagentes de interligação são usados para “fixar” proteínas ao ADN que elas ligam. Os reagentes à base de formaldeído são tipicamente usados para conseguir esta fixação. As células e tecidos são geralmente fixados de forma semelhante, mas os tecidos requerem um tempo de fixação mais longo e uma entrega de fixação mais rápida, de forma a permear rapidamente o tecido alvo antes que este comece a degenerar.
A sobrefixação da cromatina pode reduzir a eficiência da fragmentação por sonicação, além de inibir a ligação de anticorpos aos seus alvos proteicos. Portanto, o tempo de fixação deve ser determinado empiricamente a fim de permitir a máxima ligação anticorpo-antigênio, enquanto se obtém a ligação cruzada ideal das proteínas ao seu DNA alvo.
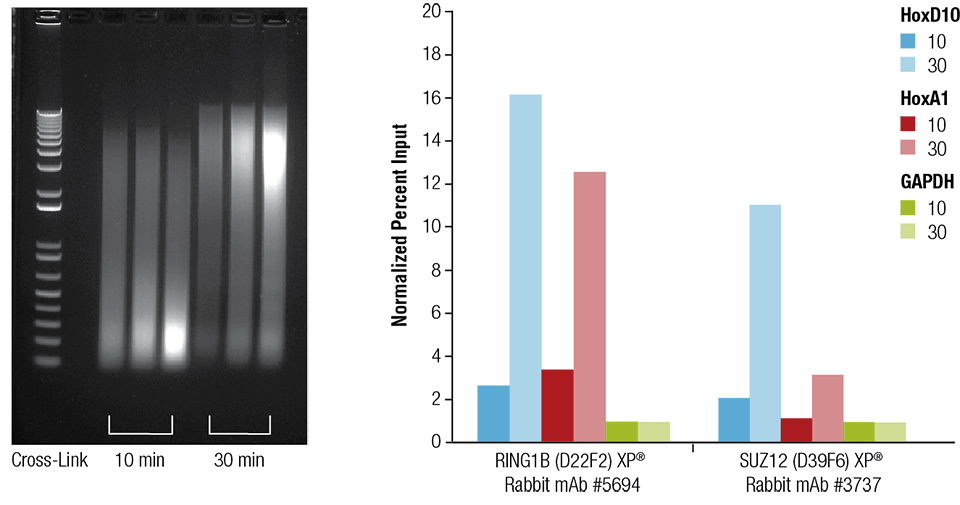
Rato coração (H), cérebro (B), e fígado (L) foram ligados transversalmente por 10 ou 30 minutos como indicado (painel esquerdo). A cromatina foi preparada e filtrada por 4 minutos. O ChIP foi realizado usando cromatina preparada a partir de tecido cardíaco com os anticorpos indicados usando o SimpleChIP® Plus Sonication Chromatin IP Kit #56383 e o DNA enriquecido foi quantificado por PCR em tempo real usando primers para os genes indicados (painel direito). A quantidade de DNA imunoprecipitado em cada amostra é representada como sinal normalizado para o GAPDH negativo dos loci, que é igual a 1,
Coração do rato (H), cérebro (B), e fígado (L) foram reticulados durante 10 ou 30 minutos como indicado (painel esquerdo). A cromatina foi preparada e filtrada por 4 minutos. O ChIP foi realizado usando cromatina preparada a partir de tecido cardíaco com os anticorpos indicados usando o SimpleChIP® Plus Sonication Chromatin IP Kit #56383 e o DNA enriquecido foi quantificado por PCR em tempo real usando primers para os genes indicados (painel direito). A quantidade de DNA imunoprecipitado em cada amostra é representada como sinal normalizado para o GAPDH negativo do loci, que é igual a 1,
6,2 Como se fragmenta a cromatina?
A fragmentação da cromatina é essencial para o sucesso de uma experiência ChIP. A fragmentação da cromatina é necessária para solubilizar a cromatina e permite a sua coprecipitação. Além disso, a resolução do ensaio ChIP depende da fragmentação da cromatina, já que o tamanho do fragmento de DNA determina a resolução do ensaio ChIP.
Digestão enzimática utiliza nuclease micrococal (MNase), que cliva o DNA de dupla cadeia entre os nucleossomos para gerar fragmentos de cromatina. Enquanto uma digestão MNase completa gera fragmentos de DNA de 150 pares de bases (mononucleosomas), uma digestão incompleta gera fragmentos de DNA entre 150 e 750 pares de bases (mononucleosomas, di-, e tri-nucleosomas). A sonicação utiliza força mecânica para fragmentar a cromatina. A sonicação fragmenta a cromatina entre e dentro dos núcleosomas, gerando uma gama de fragmentos de cromatina entre 150 e 1000 pares de bases.
Para X-ChIP, a digestão enzimática ou a sonicação é usada para cisalhar a cromatina. As condições de sonicação no protocolo ChIP de sonicação devem ser determinadas empiricamente, já que diferem por tipo celular e condição experimental. As condições de digestão são mais consistentes entre diferentes tipos celulares e tecidos, mas o tamanho do fragmento de cromatina ainda deve ser analisado antes da IP.
Para N-ChIP, as nucleases são usadas para fragmentar a cromatina a fim de manter a ligação protéica em amostras não fixadas. A fragmentação da nucleação também deve ser determinada empiricamente para minimizar a superdigestão da cromatina.
6.3 Por que usar a digestão enzimática para ChIP?
A digestão por N-ChIP deve ser usada, uma vez que as proteínas não são reticuladas ao DNA e as condições severas associadas à fragmentação baseada na sonicação resultariam na dissociação das proteínas da cromatina do DNA. O N-ChIP é ideal para analisar as interacções proteína histone-ADN, porque a ligação histone-ADN é muito forte e estável. Entretanto, o N-ChIP não funciona bem na análise do fator de transcrição e da ligação da cromatina co-fator.
A digestão enzimática ou a sonicação pode ser usada para fragmentar a cromatina no X-ChIP. Os benefícios da digestão enzimática incluem a consistência da fragmentação e condições de fragmentação suave (menor calor e detergente) que melhor preservam a integridade da cromatina e epitopos de anticorpos, resultando no aumento do enriquecimento imunológico do fator de transcrição e co-fator ligado à cromatina.
6.4 Por que usar a sonicação para fragmentar a cromatina para ChIP?
Não como a fragmentação da cromatina obtida através da digestão enzimática, a sonicação depende de forças mecânicas para fragmentar a cromatina em pedaços menores. O tamanho ideal de fragmentos de cromatina para o enriquecimento imunológico está entre 200 e 1000 pares de bases. A sonicação é o método tradicional utilizado para fragmentar a cromatina e pode ser realizada utilizando um sonicador de sonda tradicional ou sonicadores de banho de água mais avançados que proporcionam uma sonicação mais focalizada. A sonicação gera fragmentos de cromatina verdadeiramente aleatórios; no entanto, requer uma ampla otimização através de diferentes linhas celulares e tecidos e é difícil de reproduzir de experimento em experimento. A necessidade de tampões de alto detergente e o calor gerado durante a sonicação podem danificar a integridade tanto da cromatina quanto dos epitopos de anticorpos nas proteínas da cromatina.
6.5 Otimizar a sonicação da cromatina para ChIP
Fragmento de cromatina baseado na sonicação tradicionalmente utiliza tampões de alto detergente e gera calor, o que pode danificar a integridade dos epitopos de cromatina e anticorpos. Portanto, a quantidade de sonicação utilizada para fragmentar a cromatina deve ser determinada experimentalmente para diferentes linhas celulares e tecidos. Deve-se identificar e usar a quantidade mínima de sonicação necessária para gerar 150 a 1000 fragmentos de DNA de par de base para minimizar os danos à cromatina.
Antes de iniciar um ensaio ChIP completo com análise downstream por qPCR, chip de DNA ou NGS, a eletroforese em gel deve ser usada para analisar amostras de cromatina sonecadas por várias vezes. O tamanho do fragmento depende do tempo de sonicação – o tamanho do fragmento diminui à medida que o tempo de sonicação aumenta. Entretanto, os dados sugerem que tempos de sonicação mais longos não levam a melhores resultados. Portanto, executar o DNA purificado imunoprecipitado em um gel e determinar o tamanho ideal do fragmento é um método simples para determinar a quantidade mínima de sonicação necessária para o tamanho de DNA desejado e para evitar danos desnecessários à cromatina.

A cromatina digerida por enzimas foi executada em um gel de agarose. A pista 1 mostra a cromatina que está sub-digestada. A pista 2 mostra a cromatina digerida corretamente, e a pista 3 mostra a cromatina que é digerida em excesso.
Cromatina digerida por enzimas foi rodada em um gel de agarose. A pista 1 mostra a cromatina que está sub-digestada. A pista 2 mostra cromatina devidamente digerida, e a pista 3 mostra cromatina que está sobre digerida.
6.6 Como escolher um anticorpo para ChIP?
Optar por um anticorpo apropriado para uma experiência ChIP é parte integrante do seu sucesso. Os anticorpos usados em um experimento ChIP devem ser específicos para a proteína de interesse e ter alta afinidade com o antígeno. A melhor escolha de anticorpos para um experimento ChIP ou ChIP-seq é um anticorpo validado pelo ChIP ou ChIP-seq. Se não houver um anticorpo validado pelo ChIP disponível para o gene de interesse, a próxima melhor escolha é um anticorpo que tenha sido validado em IP. É importante notar que nem todos os anticorpos validados por PI funcionam no ChIP e nem todos os anticorpos validados por PI funcionam no ChIP-seq. Além disso, quanto mais um anticorpo é validado através de outras aplicações, tais como ocidental, IP, IF, fluxo e IHC, mais confiança se pode ter no desempenho e especificidade dos anticorpos. A validação de um anticorpo deve ser seguida pela determinação empírica da concentração ótima do anticorpo, juntamente com as condições de lavagem IP.
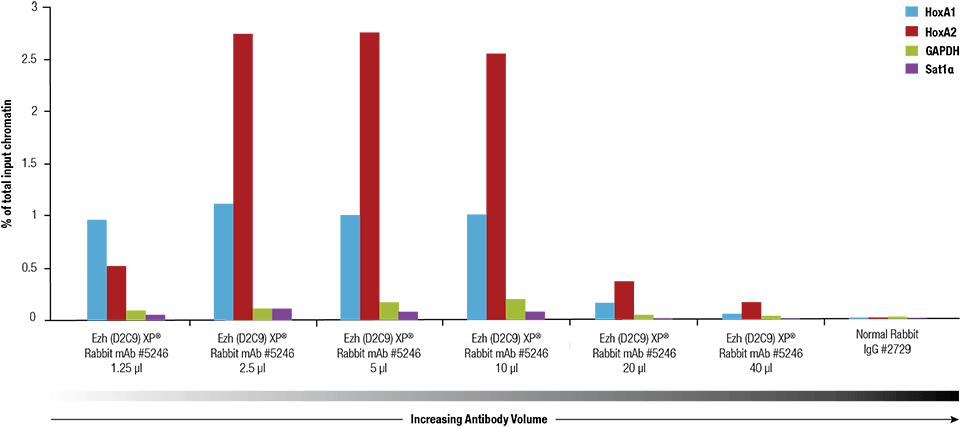
Ezh2 (D2C9) XP® Rabbit mAb #5246 foi titulado usando o SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 em cromatina reticulada preparada a partir de 4 x 106 células NCCIT.
Ezh2 (D2C9) XP® Rabbit mAb #5246 foi titulado usando o SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 em cromatina reticulada preparada a partir de 4 x 106 células NCCIT.
6.7 Como realizar a imunoprecipitação
Antibodies are used to capture a protein of interest and its bound DNA. A concentração de anticorpos deve ser determinada empiricamente, com um ponto de partida geral de 0,5-2,0 μg de anticorpos utilizados por 10 μg de ADN cromatinado (corresponde a aproximadamente 4 x 106 células). O rigor dos tampões e os tempos de lavagem também devem ser determinados empiricamente, pois dependem da afinidade do anticorpo com o seu antígeno alvo. Tipicamente, as incubações de anticorpos:cromatina são feitas durante 2 horas a uma noite.
Complexos de Anticorpo-antigénio (+DNA) são afinidades capturadas numa resina de ligação de anticorpos. Nas experiências ChIP esta resina é tipicamente composta de contas magnéticas, septosas ou agarose de grau ChIP conjugadas à proteína A e/ou proteína B. Os anticorpos ligam as contas da proteína A e/ou proteína G com várias afinidades dependendo da espécie em que foram desenvolvidos e do subtipo IgG da sua cadeia pesada. As contas são normalmente incubadas com anticorpos:cromatina durante 2 a 4 horas.
Passos de lavagem são necessários para remover a cromatina não ligada a anticorpos, seguidos pela inversão de ligações cruzadas (para X-ChIP) e purificação do ADN. Além disso, um IP de controle IgG deve ser realizado para determinar o fundo (sinal:ruído). Também devem ser incluídos anticorpos de controlo positivo (ou seja, histone total H3) e/ou primários de controlo positivo qPCR (para loci de ligação de proteínas-alvo positivas e negativas conhecidas) para determinar a ligação não específica. Para obter resultados óptimos, o QC da cromatina IP por qPCR deve ser realizado antes da análise NGS a jusante.
6.8 Como eluir a cromatina das esferas da proteína A/G
A cromatina é eluída das esferas da proteína A/G usando detergente e calor. Para manter as esferas em suspensão e aumentar a eluição da cromatina, é necessário um “vortexing” ou mistura a baixa velocidade.
6.9 Como inverter as ligações cruzadas da cromatina
As ligações cruzadas são invertidas por calor elevado e sal elevado (ambos são componentes vitais). A proteinase K também é adicionada para digerir as proteínas de cromatina associadas e adicionar anticorpos, permitindo uma purificação mais eficiente do DNA a jusante.
6.10 Como purificar o DNA
Após a remoção das ligações cruzadas de cromatina, o DNA é purificado usando o fenol-clorofórmio clássico seguido por métodos de precipitação de etanol ou usando kits de purificação de DNA em coluna.
Como analisar o DNA enriquecido
Once DNA é purificado, várias análises posteriores podem ser realizadas, incluindo ChIP-PCR, ChIP-qPCR, ChIP-chip, e ChIP-seq.
7.1 Análise ChIP-PCR e ChIP-qPCR
ChIP-PCR e análises ChIP-qPCR são melhores para análise de um único gene e podem ser usadas para amplificar e quantificar fragmentos específicos de DNA de forma rápida e econômica.
7.2 Análise ChIP-chip
Análise ChIP-chip utilizando chips de microarranjo de DNA para criar um mapa de ligação e modificação proteica em todo o genoma.
7.3 Análise ChIP-seq
Análise ChIP-seq utiliza tecnologia NGS padrão para alinhar o DNA purificado com genomas inteiros previamente anotados para identificar perfis de ligação proteica em todo o genoma.