Ce sont les notes de la conférence 4 du cours de biologie cellulaire de Harvard Extension.
La voie sécrétoire fait référence au réticulum endoplasmique, à l’appareil de Golgi et aux vésicules qui voyagent entre eux ainsi qu’à la membrane cellulaire et aux lysosomes. Elle est appelée « sécrétoire » parce qu’elle est la voie par laquelle la cellule sécrète des protéines dans l’environnement extracellulaire. Mais comme d’habitude, l’étymologie ne raconte qu’une partie de l’histoire. Cette voie traite également les protéines qui seront liées à la membrane (que ce soit dans la membrane cellulaire ou dans les membranes du RE ou du Golgi elles-mêmes), ainsi que les enzymes lysosomales, et aussi toutes les protéines qui vivront leur vie dans la voie sécrétoire elle-même. Il fait aussi d’autres choses que de traiter les protéines.
Le cytosol et le « lumen » (le liquide qui remplit la voie sécrétoire) sont des environnements chimiques différents, et ils ne se mélangent normalement jamais. Le cytosol est réducteur (lorsque vous êtes dans le cytosol, vous rencontrez sans cesse des molécules qui veulent vous offrir des électrons), et le RE, le Golgi et l’environnement extracellulaire sont oxydatifs (les molécules viennent sans cesse vers vous en demandant des électrons). Consultez la section sur l’oxydoréduction si vous ne comprenez toujours pas. Les conditions de repliement des protéines sont donc différentes : par exemple, les liaisons disulfure ne se forment généralement que dans des conditions oxydatives. En outre, différentes protéines peuvent vivre uniquement dans la voie de sécrétion ou uniquement dans le cytosol. La voie de sécrétion permet à la cellule de traiter des éléments qu’il ne serait pas bon d’avoir dans le cytoplasme et/ou qui sont plus utiles lorsqu’ils sont concentrés dans un compartiment spécialisé avec leurs partenaires d’interaction souhaités. Les hépatocytes (dans le foie) séquestrent les médicaments et les toxines dans le RE lisse et les décomposent pour les excréter de l’organisme à cet endroit. La voie de sécrétion n’est pas contiguë, mais chaque mouvement entre ses composants se fait dans de petits microcosmes bouillonnants de son propre monde chimique, appelés vésicules.
De nombreuses protéines qui passent par la voie de sécrétion ne touchent jamais le cytosol – sauf les parties des protéines membranaires qui dépassent du côté cytosolique. Beaucoup d’entre elles ont besoin de chaperons pour aider au repliement, et/ou de toute une série de modifications post-traductionnelles afin d’être prêtes pour leur fonction native, et la voie sécrétoire est spécialisée pour leur fournir tout cela.
La conférence d’aujourd’hui se concentrera sur la façon dont les protéines sont traduites dans le RE et comment elles voyagent (dans des vésicules) entre le RE, le Golgi et d’autres destinations. Ceci est magnifiquement décrit dans la vidéo Life of the Cell :
Le réticulum endoplasmique est la première étape de la voie sécrétoire. Sa membrane est continue avec la membrane nucléaire externe, bien que l’on ne sache pas très bien pourquoi cela est important, car ce n’est pas comme si les protéines commençaient leur vie dans le noyau. Les ARNm dérivent plutôt dans le cytoplasme jusqu’à ce qu’ils soient pris en charge par un ribosome désireux de les traduire. Dans le cas de la « translocation post-traductionnelle », la nouvelle protéine est déplacée dans le RE après avoir été traduite. Dans le phénomène plus intéressant appelé « translocation cotranslationnelle », le ribosome commence la traduction comme n’importe quelle autre protéine, mais quelque part dans les 16 à 30 premiers acides aminés, il rencontre un peptide signal (ou séquence signal). Le motif de ce signal est souvent constitué d’un acide aminé chargé positivement suivi de 6 à 12 acides aminés hydrophobes. Ce motif est reconnu par la particule de reconnaissance du signal (SRP, une « ribonucléoprotéine » ou molécule hybride ARN/protéine) qui s’y lie et empêche le ribosome de poursuivre la traduction. La traduction est arrêtée jusqu’à ce que le complexe ribosome/SRP rencontre un récepteur SRP sur la membrane du RE. Lorsqu’ils se rencontrent, la SRP et son récepteur se lient chacun à une molécule de GTP dans la membrane du RE, ce qui semble renforcer leur interaction. Par hasard, tout ceci se produit à proximité d’un translocon Sec61, un complexe protéique qui forme un canal traversant la membrane du RE. Le translocon est en fait un complexe de trois protéines différentes (gènes : SEC61A1 ou SEC61A2, SEC61B, SEC61G), dont la sous-unité Sec61a possède 10 hélices a de la membrane qui forment le canal. Une fois que le ribosome est amarré à la membrane, il continue la traduction, poussant le peptide signal et finalement la protéine entière à travers le canal dans la lumière du RE. Lorsque la traduction s’arrête, SRP et le récepteur SRP hydrolysent tous deux leur GTP pour se libérer mutuellement et libérer la cargaison du ribosome (cela doit nécessiter l’énergie du GTP, puisque la liaison initiale était descendante), une peptidase de signal clive le peptide de signal de la protéine naissante, et la protéine est libre de commencer à se plier dans le RE.
Un couple d’autres acteurs est impliqué pour certaines protéines du RE. L’oligosaccharide transférase, qui ajoute des groupes glycosyle aux asparagines de la protéine naissante, fait partie du complexe translocon et elle effectue effectivement la glycosylation alors que la nouvelle protéine est encore en cours de traduction. Ainsi, bien que nous appelions la glycosylation une « modification post-traductionnelle », elle est en fait effectuée pendant la traduction dans ce cas. De plus, pour obtenir leur structure correcte, certaines protéines doivent être entièrement traduites avant de pouvoir commencer à se plier. Si la partie N-terminale était autorisée à commencer à se plier dès qu’elle entre dans la lumière, elle se retrouverait avec une structure globale incorrecte. Pour éviter cela, il arrive que BiP, le chaperon, se lie à la protéine pour la maintenir dépliée pendant un certain temps. Imaginez BiP comme un autre Pac-Man qui mord sur la protéine pour la garder linéaire, comme Hsc70 dans le processus de ciblage mitochondrial (voir la semaine dernière).
Voici une vidéo à ce sujet :
Les deux premières minutes montrent le scénario de base décrit ci-dessus. Ensuite, on passe à un scénario plus complexe que je présenterai dans une minute. Pour info, la vidéo dépeint deux choses « controversées » non incluses dans la description ci-dessus : (1) le peptide signal qui est dégradé dans la membrane, et (2) une « protéine bouchon » qui bouche le canal avant/après la traduction. Tous les scientifiques ne sont pas encore d’accord sur ces deux choses.
Toutes les protéines dont nous savons qu’elles passent par la voie sécrétoire ont été repérées là par des personnes faisant des expériences de localisation pour voir où se trouve une protéine dans la cellule. Un fait étrange à propos du RE est que vous pouvez mettre la cellule dans un mixeur et qu’ensuite le RE va commencer à se reconnecter à lui-même, formant de petits « microsomes » qui ne sont pas attachés au noyau mais forment des bulles contiguës de RE. Vous pouvez alors commencer à jouer avec les protéases – qui dégradent les protéines – et les détergents – qui solubilisent la membrane du RE. En supposant que la protéine qui vous intéresse est traduite, vous pouvez vérifier si elle (1) survit au traitement par protéase mais (2) ne survit pas au traitement par protéase + détergent, alors il s’agit d’une protéine de la voie sécrétoire. La logique est que dans le cas (1), elle était protégée à l’intérieur du RE, mais dans le cas (2), vous avez dissous le RE, donc elle a été mangée par la protéase. Tout cela suppose que vous avez un anticorps ou un autre moyen de détecter si la protéine d’intérêt est là après ces traitements.
Les gens ont également utilisé ces techniques pour comprendre que seulement 70 acides aminés d’une nouvelle protéine peuvent être traduits avant qu’il ne devienne trop tard pour que cette protéine se retrouve dans le RE. Rappelez-vous, le peptide signal se trouve dans les 16-30 premiers acides aminés, et la translocation vers le RE dépend de la présence du SRP. Les ribosomes traduisent à un rythme prévisible, donc les gens ont fait en sorte que les ribosomes commencent à traduire certains ARNm et ont ensuite attendu des quantités de temps déterminées avant d’ajouter la SRP, pour voir combien de traduction pouvait se produire avant que la SRP ne puisse plus faire son travail.
Le récepteur SRP et les protéines Sec61 sont des protéines de la membrane du RE – et il y a beaucoup d’autres protéines de la membrane du RE, de la membrane du Golgi et de la membrane du lysosome également. En fait, même les protéines membranaires (voir classe 02) de la membrane cellulaire sont traitées dans la voie sécrétoire. Beaucoup d’entre elles possèdent plusieurs dizaines de domaines transmembranaires (20 à 25 acides aminés hydrophobes chacun) qui doivent être insérés dans l’ordre et l’orientation corrects (par exemple, vous voulez vraiment que vos canaux ioniques et vos transporteurs soient orientés dans la bonne direction, vers l’intérieur ou l’extérieur de la cellule). Par conséquent, il existe tout un tas de mécanismes biologiques sophistiqués permettant d’insérer correctement ces protéines dans la membrane. C’est ce que décrit la dernière moitié de la vidéo ci-dessus.
Donc voici une tautologie : certaines protéines ont une séquence topogénique qui détermine leur orientation dans la membrane. Cette séquence est constituée de deux types de séquences signal :
- une séquence stop-transfert (abrégée STA pour une raison quelconque) est une séquence de 22-25 acides aminés hydrophobes quelque part au milieu de la protéine qui forme une hélice alpha. Lorsqu’elle est rencontrée, elle est poussée dans la membrane, et la traduction du reste de la protéine se poursuit dans le cytosol. Donc, cela permet en quelque sorte de » défaire » la translocation vers le RE qui a été amorcée par le peptide signal au début (extrémité N) de la protéine.
- Une séquence d’ancrage du signal (abrégée SA) est également une hélice alpha hydrophobe de 22-25aa, mais avec une série de ~3 acides aminés chargés positivement à sa gauche ou à sa droite. Comme le peptide signal, elle est reconnue par SRP, qui amène le ribosome au RE. Mais contrairement au peptide signal, cette séquence hélicoïdale alpha sera insérée dans la membrane du RE. L’orientation de l’insertion est déterminée par les 3 acides aminés chargés positivement. Les charges positives doivent toujours se retrouver du côté cytosolique, donc s’ils viennent après (c’est-à-dire C-terminal de) la séquence hydrophobe, la protéine se retrouve avec son extrémité C terminale pointée dans le cytosol, mais s’ils viennent avant (c’est-à-dire N-terminal de) la séquence hydrophobe, la protéine se retrouve avec son extrémité N terminale pointée dans le cytosol.
Avec ces deux signaux comme blocs de construction, vous pouvez imaginer une protéine avec une série de séquences de transfert d’arrêt et d’ancrage de signal pour créer toute une série de domaines transmembranaires d’avant en arrière cousus dans la membrane comme par une machine à coudre. Les gens ont classé les protéines membranaires en cinq catégories:
- Le type I a juste un peptide signal et ensuite un transfert d’arrêt au milieu. Elle finit donc avec son extrémité N (hydrophile) dans la lumière, son milieu (hydrophobe) dans la membrane et son extrémité C (hydrophile) dans le cytosol.
- Le type II ne commence pas par un peptide signal. Il commence comme n’importe quelle autre protéine, mais au milieu, il a une séquence d’ancrage de signal avec les acides aminés +++ venant en premier et la série hydrophobe après. Cela fait que la protéine est transloquée à mi-chemin de la traduction, la partie N-terminale déjà traduite dépassant dans le cytosol (puisque les +++ doivent rester cytosoliques) et la partie C-terminale qui commence à être traduite étant traduite directement dans le RE. Il finit donc par être transmembranaire avec son extrémité C dans le RE et son extrémité N dans le cytosol – à l’opposé du type I.
- Le type III est comme le type II – pas de peptide signal, juste une ancre signal au milieu, mais dans ce cas les +++ viennent après la séquence hydrophobe, ce qui inverse l’orientation. Ainsi, la protéine se retrouve avec son extrémité N dans le RE et son extrémité C dans le cytosol. À l’opposé du type II et, au final, identique au type I, bien qu’il y soit arrivé d’une manière différente – il n’a pas de peptide signal qui est clivé dans le RE.
- Les protéines de type IV ou ‘multipass’ ont une série alternée de séquences signal et de séquences de transfert d’arrêt. Il s’agit clairement de plus d’un « type », mais elles sont loin d’être aussi diverses que votre imagination combinatoire pourrait le permettre. L’orientation de la première séquence signal détermine si l’extrémité N terminale se retrouvera dans le cytosol ou le RE, et le nombre total de séquences d’ancrage de transfert d’arrêt + signal détermine où l’extrémité C terminale se retrouvera : un nombre pair = même côté que l’extrémité N terminale, un nombre impair = côté opposé à l’extrémité N terminale. Les séquences STA et SA doivent strictement alterner, à l’exception du fait que vous pouvez commencer avec deux séquences d’ancrage de signal si la première est orientée avec l’extrémité N dans le cytosol. Pour tourner en dérision ce schéma de catégorisation, des personnes ont défini des sous-types de type IV incomplètement définis, où le type IVa est N-terminal dans le cytosol (il commence donc comme une protéine de type II) et le type IVb est N-terminal dans la lumière (il commence comme une protéine de type III mais possède ensuite une autre séquence SA qui le renvoie dans le RE). GLUT1 de la classe 02 est un type IVa.
- Les protéines à ancrage GPI, qui constituent le cinquième type mais ne sont pas appelées type V, commencent par un peptide signal et se terminent par une extrémité C hydrophobe qui reste encastrée dans la membrane. Cette extrémité hydrophobe est clivée et remplacée par du GPI, qui reste également ancré dans la membrane. La PrP est l’une de ces protéines – nous y reviendrons plus tard.
Nous avons maintenant discuté de la façon dont les protéines peuvent se retrouver dans la lumière du RE ou enjamber la membrane du RE. La plupart des protéines quittent le RE en quelques minutes, transportées dans des vésicules destinées au Golgi, puis plus tard à l’excrétion, aux lysosomes ou à la membrane cellulaire. Ce sens de déplacement vers l’avant est appelé antérograde ; revenir en arrière, du Golgi au RE, est un transport rétrograde.
Les deux types de transport ont lieu dans des vésicules liées à la membrane. Celles-ci bourgeonnent de la membrane de l’endroit d’où elles viennent et fusionnent ensuite avec la membrane de l’endroit où elles se dirigent – magnifiquement illustré à ~2:25 dans la vidéo Life of the Cell ci-dessus. Le corps à partir duquel les vésicules se forment est le « compartiment donneur », et la destination vers laquelle elles fusionnent plus tard est le « compartiment accepteur ».
Le processus de bourgeonnement nécessite que les protéines G de la membrane recrutent des protéines Coat. Plus précisément, pour le transport antérograde, la protéine G Sar1 (gène : SAR1A) recrute la COPII ( » cop two « ) ; pour le transport rétrograde, une protéine G ARF recrute la COPI (prononcé » cop one « ). Ces protéines G sont activées pour faire ce travail lorsque GEF les charge de GTP, en échangeant le GDP.
Donc les étapes du transport antérograde, par exemple, sont les suivantes :
- Sec12-GEF (Sec signifie sécrétoire) charge Sar1 de GTP. Lorsqu’elle est liée au PIB, Sar1 flotte simplement dans le compartiment donneur, mais lorsqu’elle est liée au GTP, elle subit un changement de conformation qui fait saillir sa queue hydrophobe N-terminale autrement brûlée, ce qui la fait adhérer à la membrane, où les protéines COPII commencent alors à s’accumuler parce qu’elles aiment vraiment cette queue.
- Les COPII commencent à se polymériser et, en raison de sa conformation, ont une préférence intrinsèque pour la courbure, donc leur accumulation commence à faire en sorte que le bourgeonnement se produise. Dans le même temps, les protéines liées à la membrane qui doivent être transportées – identifiées par une séquence d’acides aminés DXE (c’est-à-dire aspartate-anything-glutamate) qui forme un site de liaison dans leur partie cytosolique – sont recrutées dans la vésicule nouvellement formée. Les protéines liées à la membrane agissent comme des récepteurs, recrutant les protéines luménales qui sont liées au Golgi pour qu’elles s’accrochent dans l’espace concave où elles se retrouveront dans la vésicule une fois celle-ci formée.
- Une fois que suffisamment de COPII sont arrivés, la vésicule bourgeonne, à ce moment-là, Sar1 hydrolyse son GTP, fournissant l’énergie pour qu’il aspire sa queue hydrophobe en arrière de lui-même, coupant les COPII détachés. La vésicule est maintenant déconnectée du compartiment donneur.
- Maintenant, pour des raisons mal expliquées (ou mal comprises ?), le manteau des COPIIs vient de se désassembler, exposant des récepteurs sous le manteau qui dirigent le ciblage de la vésicule. Une fois la vésicule arrivée à destination, le Rab-GTP intégré à la membrane de la vésicule interagit avec un effecteur Rab intégré à la membrane du compartiment accepteur. Un regard en coin est échangé, l’intérêt est éveillé. Bientôt, la vésicule fusionnera avec la membrane.
- Les protéines SNARE présentes à la fois sur la vésicule et la membrane cible (respectivement V-SNARE et T-SNARE) interagissent pour rapprocher encore plus les membranes. Dans cet exemple, nous considérerons VAMP (les gènes VAMP_) comme le V-SNARE et Syntaxin (les gènes STX__) et SNAP25 (gène SNAP25) comme les T-SNARE. La Syntaxine et la SNAP25 sont toutes deux des protéines membranaires ; la Syntaxine possède une hélice alpha et la SNAP25 en possède deux, toutes situées du côté cytosolique. Les hélices alpha dirigent l’interaction avec le VAMP. Les hélices alpha des côtés opposés ont une affinité extrêmement forte l’une pour l’autre, ce qui rapproche suffisamment les membranes pour qu’elles fusionnent. Une fois que cela s’est produit, il faut deux protéines pour séparer à nouveau les V-SNARE et les T-SNARE : NSF (gène : NSF ; abréviation de NEM sensitive factor) et alpha-SNAP (gène : NAPA), une protéine soluble d’attachement NSF. NSF est une ATPase, et brûle l’ATP pour conduire le désassemblage énergétiquement ascendant du complexe.
Maintenant pour le transport rétrograde. Pourquoi y a-t-il un transport rétrograde ? Voici une liste non exhaustive de quelques raisons :
- Certaines protéines membranaires commencent leur vie dans le RE, doivent être modifiées dans le Golgi, mais doivent ensuite retourner dans le RE. Elles le font avec une séquence d’acides aminés KKXX.
- Il y a aussi une séquence d’acides aminés KDEL à l’extrémité C de certaines protéines luménales qui est supposée les maintenir dans le RE, mais ce n’est pas parfait – parfois elles finissent dans le Golgi, auquel cas elles sont ciblées de nouveau vers le RE par transport rétrograde dépendant de cette séquence KDEL pour la reconnaissance. Le mécanisme est assez net – les protéines qui reconnaissent et se lient à KDEL ne le font qu’à faible pH, et le pH du Golgi est inférieur à celui du RE, donc elles se lient à KDEL dans le Golgi, puis le libèrent lorsqu’elles sont de retour dans le pH plus neutre du RE.
- En outre, pensez-y, toutes les protéines qui participent au transport antérograde – les V-SNARES, Rab, etc. – doivent retourner au RE pour pouvoir recommencer, comme le bus doit retourner au dépôt de bus à la fin de la journée.
- Comme nous le verrons bientôt, le Golgi vient en plusieurs étapes qui dépendent de l’ajout d’enzymes de plus en aval.
Le processus de transport rétrograde n’est pas si différent de l’antérograde. Il utilise ARF au lieu de Sar1, COPI au lieu de COPII, mais il fonctionne de la même manière : ARF chargé de GTP laisse sa queue hydrophobe se coller dans la membrane, attirant l’attention des COPI. La COPI a deux composants, COPIalpha et COPIbeta, qui interagissent tous deux avec cette séquence KKXXX pour recruter des protéines liées à la membrane et destinées au transport rétrograde. Certaines protéines possèdent également une séquence RR (n’importe où dans la protéine) qui peut les signaler pour le transport rétrograde.
L’appareil de Golgi n’est pas contigu. C’est un ensemble empilé de sous-compartiments séparés appelés sacs ou cisternes. Les différents compartiments ont des propriétés différentes et les protéines les visitent dans un ordre particulier. Dans l’ordre, du RE à la membrane cellulaire, les compartiments de Golgi sont appelés réseau cis, médian, trans et trans-Golgi. Chaque compartiment possède différentes enzymes qui modifient les protéines, et les modifications doivent se produire dans un certain ordre, d’où la nécessité d’un ensemble de compartiments empilés.
Mais lorsque les protéines arrivent à maturité dans le Golgi, ce n’est pas comme si elles bourgeonnaient dans des vésicules d’un compartiment et passaient au suivant. Au contraire, le compartiment dans lequel elles se trouvent déjà se déplace vers l’extérieur et « mûrit » au fur et à mesure que de nouvelles enzymes y sont ajoutées (en provenance de plus loin dans la chaîne de Golgi) par transport rétrograde. Bizarre, non ? C’est un peu comme si, au lieu de passer d’une école primaire à un collège puis à un lycée, vous restiez dans le même bâtiment scolaire pendant toute votre enfance et votre adolescence, et qu’on vous apportait de nouveaux manuels et de nouveaux professeurs chaque année pour que l’école soit adaptée au niveau que vous et vos camarades avez atteint. Voici à quoi ressemblent les Golgi lorsqu’ils se déplacent et évoluent :
Il y a donc (peu ou) pas de transport antérograde au sein des Golgi, mais beaucoup de transport rétrograde pour amener chaque nouvelle série d’enzymes. Lorsque les protéines ont finalement terminé le cursus complet du réseau de Golgi, elles subissent un transport pour se rendre à leur destination finale. Elles bourgeonnent dans une vésicule qui ira à l’un des trois endroits suivants :
- Exocytose – fusion avec la membrane cellulaire. Ainsi, les protéines luménales seront sécrétées de manière extracellulaire, et les protéines membranaires deviendront des protéines de la membrane cellulaire.
- Vésicules sécrétoires – elles restent simplement en tant que vésicules dans la cellule jusqu’à ce qu’on en ait besoin – où « besoin » signifie qu’elles finissent par subir une exocytose. Dans les neurones, c’est là que les neurotransmetteurs sont stockés jusqu’à ce qu’un potentiel d’action exige leur sécrétion dans la synapse. Dans l’estomac, les cellules qui produisent les enzymes gastriques conservent ces enzymes dans des vésicules sécrétoires jusqu’à ce que la prise de nourriture déclenche leur libération dans l’estomac.
- Lysosomes – où les protéines mal repliées vont pour être dégradées.
Le transport du réseau trans-Golgi sur ces destinations est différent des autres transports discutés ci-dessus et implique souvent la clathrine (gènes CLT__). Les vésicules bourgeonnantes ont un revêtement à deux couches, avec des complexes de protéines adaptatrices (AP) comme couche interne et la clathrine comme couche externe. Les protéines adaptatrices ont un signal cible avec un motif YXXh (h = Φ = tout acide aminé hydrophobe). La clathrine forme ce qu’on appelle la formation « clathrine-triskelion » représentée ici :

(Image grâce à l’utilisateur Wikimedia Commons Phoebus87)
La clathrine est également responsable de l’endocytose – le bourgeonnement de vésicules de substance extracellulaire (et de protéines de la membrane cellulaire) pour entrer dans la cellule. C’est ce qu’on appelle l’endocytose médiée par la clathrine. Les récepteurs de la membrane cellulaire sont endocytés très fréquemment : la population entière des récepteurs hormonaux se renouvelle environ toutes les heures, en particulier lors de la réception d’hormones. L’absorption du récepteur dans une vésicule est une façon pour la cellule de couper le signal entrant jusqu’à ce qu’il puisse être traité.
Les notes sur la membrane plasmique abordent brièvement la mucoviscidose : Le CFTR est un transporteur ABC responsable du pompage du Cl- hors de la cellule (il laisse également entrer le Na+). Les mutants de perte de fonction ne pompent pas le Cl-, ce qui supprime la force motrice de l’osmose, épaississant le mucus et causant des problèmes respiratoires. Il existe au moins 127 mutants de perte de fonction CFTR différents (du moins, c’est le nombre de mutants testés par Natera) qui (si les deux allèles sont désactivés) provoquent la fibrose kystique. La mutation la plus courante est ΔF508, qui représente ~3 % de tous les allèles CFTR européens et environ 70 % des allèles mutants. La perte de cette seule phénylalanine modifie la conformation de CFTR de sorte que le code de sortie di-acide (acides aminés D565 et D567) qui cible CFTR pour les vésicules exocytotiques n’est plus correctement exposé et la protéine ne parvient jamais à la membrane cellulaire.
Section discussion
Dans la section, nous avons lu Hu 2009, qui a montré que les protéines atlastines sont impliquées dans la création du réseau tubulaire du RE. La preuve provenait presque entièrement des interactions protéine-protéine. J’ai été surpris que cet article soit si important, car il y a eu un million d’articles montrant des interactions protéine-protéine pour la huntingtine, et personne ne les croit vraiment tous et cela ne nous a pas nécessairement rapprochés de la connaissance de ce que fait la huntingtine ou de ce qui ne va pas dans la maladie de Huntington. Mais apparemment, Hu a été en mesure de démontrer clairement que les interactions des atlastines avec les réticulons impliquent un rôle dans la formation du RE. Le fait que Hu ait pu montrer une « interaction génétique » en plus d’une interaction physique (liaison) est utile. Une « interaction génétique » (j’ai dû chercher) signifie que « parfois, des mutations dans deux gènes produisent un phénotype qui est surprenant à la lumière des effets individuels de chaque mutation. Ce phénomène, qui définit l’interaction génétique, peut révéler des relations fonctionnelles entre les gènes et les voies. » .
PrP
Cela date d’une décennie, donc certaines choses peuvent être dépassées, mais j’ai trouvé la revue de Harris 2003 (ft) sur la biologie cellulaire de la PrP extrêmement claire et utile. Kim & Hegde 2002 a également été utile. La PrP est une protéine de la voie sécrétoire. Ses 22 premiers acides aminés (MANLGCWMLVLFVATWSDLGLC) sont un peptide signal qui provoque une translocation cotranslationnelle vers le RE. Normalement, la PrP est simplement liée à la GPI à son extrémité C et est ancrée sur le côté exoplasmique de la membrane. Mais les acides aminés 111-134 (HMAGAAAAGAVVGGLGGYMLGSAM) sont une sorte de séquence d’ancrage de signal faible (type II, les acides aminés +++ venant avant l’ancrage de signal) qui devient parfois, mais pas toujours, un domaine transmembranaire, inversant l’extrémité C dans la lumière. Plus déroutant encore, cette séquence peut parfois se transformer en domaine transmembranaire sans inversion, de sorte que l’extrémité N se trouve dans la lumière. Il existe donc trois topologies membranaires de la PrP : l’ancien ancrage GPI ordinaire et deux orientations transmembranaires, comme le montre la figure 3 de Harris 2003 :
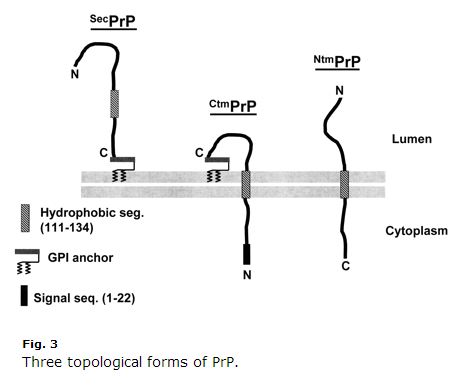
Notez à quel point la CtmPrP est bizarre. Elle est transmembranaire mais aussi ancrée GPI, et le peptide signal N-terminal n’est jamais clivé. Normalement, les formes transmembranaires représentent <10% de la PrP totale. Dans certaines conditions de laboratoire, le pourcentage est plus élevé, et deux des mutations causant le GSS (A117V et P105L) augmentent également la fraction de CtmPrP à 20-30% de toute la PrP. Parmi ces trois formes, il existe un bon nombre de preuves que la CtmPrP est toxique, et qu’elle pourrait jouer un rôle dans la formation des prions, bien que la plupart des mutations génétiques des maladies à prions (y compris la FFI D178N) ne semblent pas affecter la topologie membranaire de la PrP ou la fraction de CtmPrP.
Après avoir traversé le Golgi, la PrP est ciblée pour la membrane cellulaire. Mais selon Harris, elle ne reste pas là – elle passe fréquemment par une endocytose médiée par la clathrine et effectue des cycles dans la cellule toutes les ~60 minutes, certaines molécules étant clivées à chaque cycle. Le cuivre stimule cette endocytose de la PrP. La plupart des mutations génétiques des maladies à prions modifient la localisation de la PrP – en général, lorsqu’une mutation est présente, on trouve moins de PrP à la surface des cellules, une plus grande quantité s’accumulant dans le RE.