Mikä on ChIP?
Kromatiinin immunoprecipitointi eli ChIP on vasta-ainepohjainen tekniikka, jota käytetään tiettyjen DNA:ta sitovien proteiinien ja niiden DNA-kohteiden selektiiviseen rikastamiseen. ChIP:tä käytetään tietyn proteiini-DNA-vuorovaikutuksen, useiden proteiini-DNA-vuorovaikutusten tai koko genomin tai geenien osajoukon vuorovaikutusten tutkimiseen.
ChIP:ssä hyödynnetään vasta-aineita, jotka tunnistavat ja sitovat valikoivasti proteiineja, mukaan lukien histoneja, histonimodifikaatioita, transkriptiotekijöitä ja kofaktoreja, saadakseen tietoa kromatiinin tiloista ja geenien transkriptiosta. ChIP:ssä käytettävien proteomianalyysin ja molekyylibiologisten tekniikoiden yhdistelmä antaa mahdollisuuden ymmärtää geenien ilmentymistä ja säätelyä kiinnostavissa soluissa tai kudoksissa.
Milloin ChIP:tä käytetään?
Tyypillisesti ChIP:tä käytetään tietyn proteiinin tai tietyn proteiinimodifikaation suhteellisen runsauden tunnistamiseen tietyllä alueella genomissa. ChIP:n avulla voidaan vastata moniin tieteellisiin kysymyksiin, jotka liittyvät proteiinien ja kromatiinin vuorovaikutukseen. ChIP:n avulla voidaan esimerkiksi verrata tiettyjen proteiinien esiintymistä eri lokuksissa, kartoittaa eri proteiineja kiinnostavalla genomialueella tai kvantifioida proteiinien sitoutumista indusoituvaan geeniin vasteena ärsykkeeseen ajan kuluessa.
Miten ChIP toimii?
ChIP:n periaate on suhteellisen yksinkertainen, ja se perustuu vasta-aineen käyttöön tietyn proteiinin, histonin, transkriptiotekijän tai kofaktorin ja siihen sitoutuneen kromatiinin eristämiseksi tai saostamiseksi soluista tai kudoksista uutetusta proteiiniseoksesta. Tästä juontaa juurensa tekniikan nimi: Kromatiinin immunoprecipitointi. ChIP-PCR:ssä tai ChIP-seq:ssä immuunirikastetut DNA-fragmentit voidaan sitten tunnistaa ja kvantifioida käyttämällä laajalti saatavilla olevia PCR- tai qPCR-reagensseja ja seuraavan sukupolven sekvensointitekniikoita (Next Generation Sequencing, NGS).
Mitä on natiivi ChIP (N-ChIP) vs. ristiinkytketty ChIP (X-ChIP)?
Kokeellisesta tutkimuskysymyksestä ja kokeen lähtöaineistosta riippuen on olemassa kahta erityyppistä ChIP-menetelmää, jotka voidaan suorittaa: 1) natiivi ChIP (N-ChIP) ja 2) ristisilloitettu ChIP (X-ChIP). Molemmilla ChIP-tyypeillä on etuja ja haittoja:
- N-ChIP:ssä ei käytetä kiinnitysainetta proteiinien ristisidontaan kromatiiniin. Sen sijaan natiivia kromatiinia eristetään solun ytimistä, jotka pilkotaan nukleaasilla. Koska vasta-aineet kasvatetaan kiinnittämättömiä antigeenejä vastaan, N-ChIP:n etuna on vasta-aineiden parempi tunnistaminen ja sitoutuminen kohdeantigeeneihinsa. PCR:ää ei välttämättä tarvita jatkoanalyysissä, koska histoniproteiineja on runsaasti. Vaikka nämä edut tekevät N-ChIP:stä houkuttelevan menetelmän, sitä voidaan käyttää vain histonien osoittamiseen. Lisäksi proteiinien sitoutumisen häviäminen kromatiinin pilkkomis- ja immunoprecipitointivaiheiden aikana voi vääristää tietoja tai vaikeuttaa asianmukaisia analyysejä.
- X-ChIP:ssä käytetään kemiallisia fiksatiiveja, kuten formaldehydiä, kiinnostavan proteiinin ristiinsidontaan DNA:han, ja kromatiinin fragmentoituminen saavutetaan sonikaatiolla tai nukleaasien pilkkomisella. X-ChIP:n etuna on, että sitä voidaan käyttää histoniproteiinien ja muiden kuin histoniproteiinien kanssa ja että se vaatii yleensä vähemmän solujen lähtöaineistoa kuin N-ChIP. X-ChIP minimoi myös kromatiiniproteiinien häviämisen mahdollisuudet uuton aikana, mikä mahdollistaa ohimenevien proteiinien vuorovaikutusten havaitsemisen. Saostusvaihe on kuitenkin tehottomampi, ja DNA:n monistaminen PCR:llä on tarpeen jatkoanalyysejä varten.
Mitä erityyppisiä ChIP-määrityksiä on olemassa?
Kun itse kromatiinin immunoprecipitointi on valmis, puhdistetulle kromatiinille ja siihen liittyville proteiineille, histoneille, transkriptiofaktoreille ja kofaktoreille, voidaan tehdä useita jatkoanalyysejä. Yleisimmät menetelmät yksittäisen geenin analyysiin ja koko genomin analyysiin ovat qPCR ja ChIP-seq. PCR ja ChIP-chip ovat myös vaihtoehtoja jatkoanalyysejä varten.
5.1 Mitkä ovat ChIP-PCR:n edut?
ChIP-PCR suoritetaan histonimodifikaatioiden ja/tai proteiinien sitoutumisen analysoimiseksi tunnettuun osajoukkoon kohdelokuksia genomissa. ChIP-PCR:ssä immuunilla rikastetut DNA-fragmentit tunnistetaan ja kvantifioidaan käyttämällä yleisesti saatavilla olevia PCR- tai qPCR-reagensseja ja -tekniikoita. ChIP-qPCR:n avulla voidaan tehdä nopeita ja kvantitatiivisia vertailuja genomin tiettyjen alueiden välillä useissa näytteissä. Tämä on halvempaa ja ajallisesti tehokkaampaa kuin koko genomin sekvensointimenetelmät.
5.2 Mitkä ovat ChIP-chip-tekniikan edut?
ChIP-chip-tekniikalla tarkoitetaan DNA-mikrosirun hyödyntämistä ChIP-immuunilla rikastettujen DNA-fragmenttien analysointiin. Genomilaatoitus-mikrosirutekniikan käyttö mahdollistaa eristettyyn DNA:han sitoutuneiden proteiinien koko genomin analyysin ja tuottaa korkean resoluution genomikartan proteiineihin sitoutumisesta ja proteiinimodifikaatioista. ChIP-sirulla on useita käyttötarkoituksia sekä perustutkimuksessa että sairauksiin perustuvassa tutkimuksessa. Sen avulla voidaan esimerkiksi tunnistaa transkriptiotekijöiden, tehostimien ja repressorien sitoutumiskohdat ja vertailla tämäntyyppisiä sitoutuneita proteiineja kontrolli- ja patologisissa näytteissä. Koska NGS:n kustannukset ovat kuitenkin laskeneet huomattavasti ja ChIP-seq:lla saadaan samankaltaisia tuloksia, yhä useammat valitsevat ChIP-seq:n ChIP-chip:n sijaan.
5.3 Mitä etuja ChIP-seq:llä on?
Samoin kuin ChIP-chip:llä, myös ChIP-seq:llä saadaan tietoa proteiinien sidonnaisuudesta genomin laajuisesti. Toisin kuin ChIP-chip, ChIP-seq käyttää kuitenkin NGS-tekniikkaa DNA-fragmenttien tunnistamiseen ja niiden kartoittamiseen koko genomia vasten.
Nykyaikaisempi DNA:n monistustekniikka mahdollistaa vankan analyysin tekemisen muutamassa päivässä pienellä syötettävän DNA:n määrällä. Kun lähtöaineistoa on niukasti, nämä teknologiset edistysaskeleet kirjastonvalmistusmenetelmissä ovat mahdollistaneet ChIP-seq-kokeet.
Lisäksi uusi teknologia, jossa DNA-näytteet merkitään yksiselitteisesti lyhyillä sekvensseillä, niin sanotuilla viivakoodeilla, mahdollistaa nyt yksittäisten fragmenttien yhdistämisen yhdelle sekvensointikaistalle multipleksoitua analyysia varten. Tämä on lisännyt huomattavasti DNA-sekvensointikokeiden tehokkuutta ja alentanut niiden kustannuksia, mikä tukee entisestään ChIP-seq-sovelluksia.
Kokonaisuudessaan DNA-sekvensointitekniikan kehittymisen ansiosta ChIP-seq:n etuna on se, että suuri määrä ChIP-rikastettuja DNA-näytteitä voidaan sekvensoida halvalla suhteellisen lyhyessä ajassa ja suuremmalla herkkyydellä ja tarkkuudella kuin ChIP-chipin näytteet.
Mitkä ovat ChIP-määrityksen eri vaiheet?
ChIP-määritykset noudattavat yleistä protokollaa:
- Proteiinien ristikytkentä DNA:han vain X-ChIP:n osalta
- Solujen lyysi
- Kromatiinin fragmentointi digestion (X-ChIP:n ja N-ChIP:n osalta) tai sonikaation avulla-(vain X-ChIP:n osalta)
- Immunoprecipitointi spesifisten vasta-aineiden avulla
- DNA:n puhdistus jatkoanalyysejä varten
- DNA:n analysointi PCR:n avulla, qPCR:n, mikrosirujen tai NGS:n avulla
Positiiviset ja negatiiviset kontrollit jokaisessa vaiheessa ovat olennaisen tärkeitä määritettäessä, onko ChIP-kokeilu onnistunut.
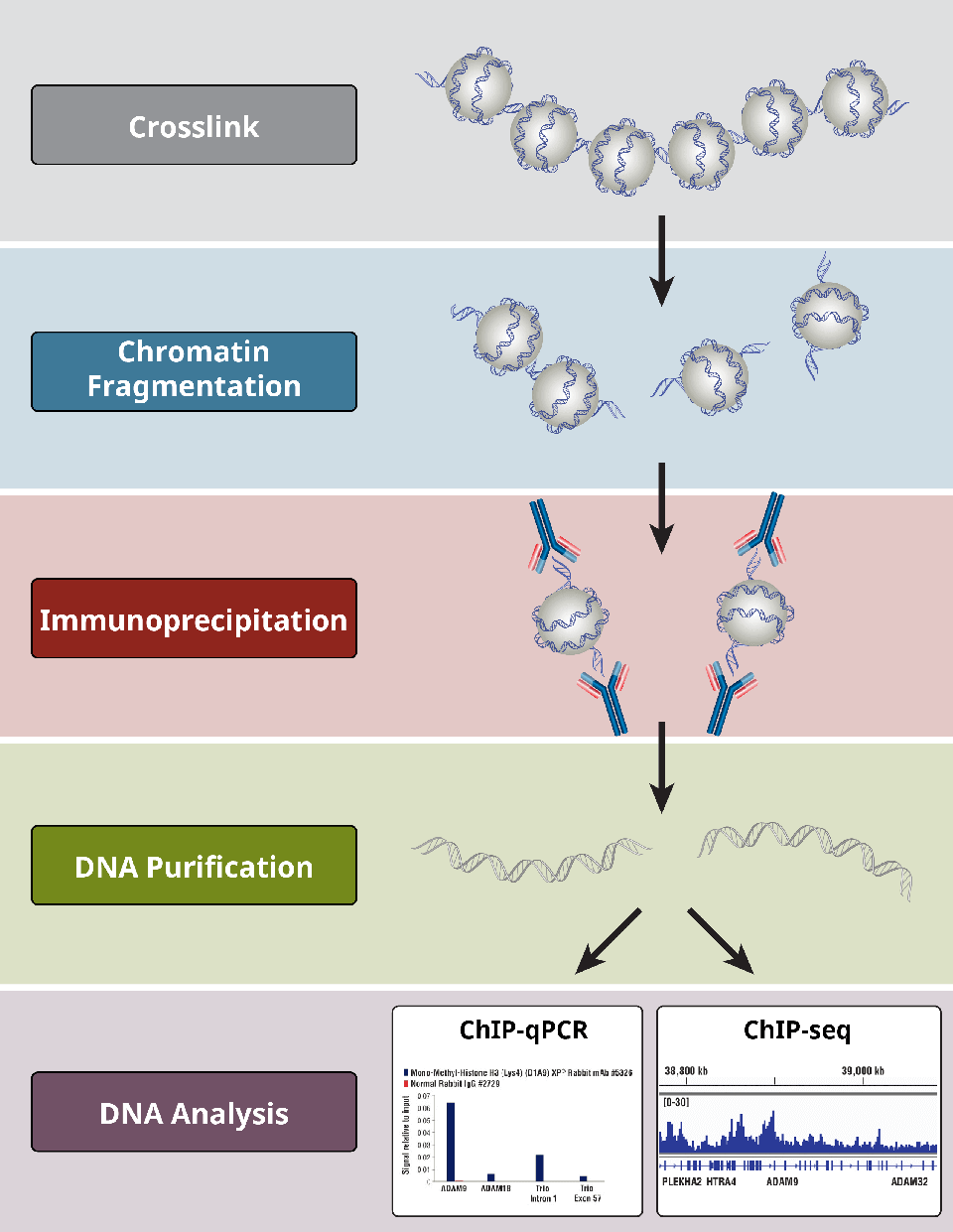
Yleiskatsaus ChIP-protokollan kriittisimpiin vaiheisiin.
Yleiskatsaus ChIP-protokollan kriittisimpiin vaiheisiin.
6.1 Miten solut ja kudokset ristisidotaan ChIP:tä varten?
Ristisilloittavia reagensseja käytetään proteiinien ”kiinnittämiseen” DNA:han, jota ne sitovat. Formaldehydipohjaisia reagensseja käytetään tyypillisesti tämän kiinnityksen aikaansaamiseksi. Solut ja kudokset kiinnitetään yleensä samalla tavalla, mutta kudos vaatii pidemmän kiinnitysajan ja nopeamman kiinnitystoimituksen, jotta kohdekudos saadaan nopeasti läpäistyä ennen kuin se alkaa degeneroitua.
Kromatiinin liiallinen kiinnittäminen voi heikentää fragmentoinnin tehokkuutta sonikoimalla, minkä lisäksi se estää vasta-aineiden sitoutumisen proteiinikohteisiinsa. Siksi fiksaatioaika olisi määritettävä empiirisesti, jotta mahdollistetaan maksimaalinen vasta-aine-antigeeni-sitoutuminen ja saavutetaan samalla proteiinien ihanteellinen ristisilloittuminen kohde-DNA:han.
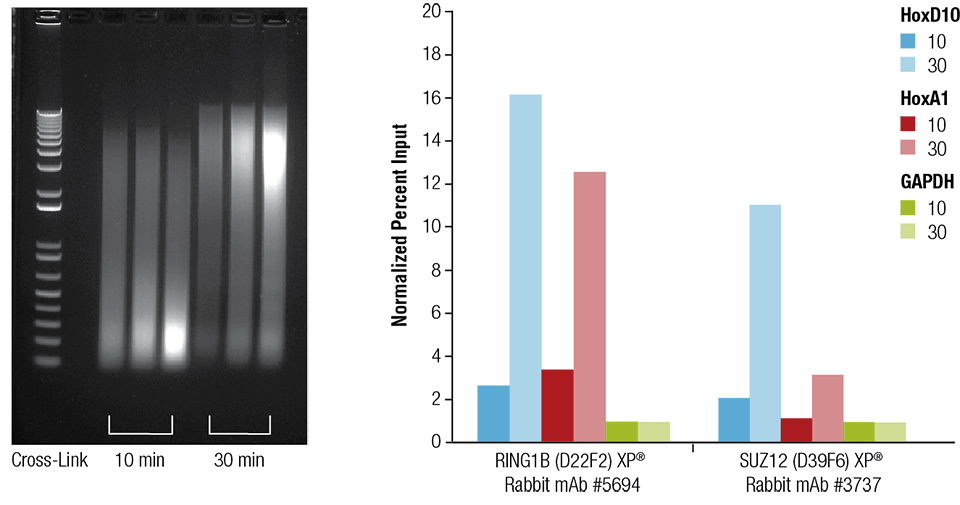
Hiiren sydäntä (H), aivoja (B) ja maksaa (L) ristisilloitettiin 10 tai 30 minuuttia, kuten ilmoitettu (vasen paneeli). Kromatiini valmistettiin ja sonikoitiin 4 minuuttia. ChIP suoritettiin sydänkudoksesta valmistetusta kromatiinista ilmoitetuilla vasta-aineilla käyttäen SimpleChIP® Plus Sonication Chromatin IP Kit #56383 -pakettia, ja rikastunut DNA kvantifioitiin reaaliaikaisella PCR:llä käyttäen ilmoitettujen geenien alukkeita (oikea paneeli). Immunoprecipitoidun DNA:n määrä kussakin näytteessä esitetään normalisoituna signaalina negatiiviseen lokukseen GAPDH, joka on yhtä suuri kuin 1.
Hiiren sydän (H), aivot (B) ja maksa (L) ristisilloitettiin 10 tai 30 minuutin ajaksi kuten ilmoitettu (vasen paneeli). Kromatiini valmistettiin ja sonikoitiin 4 minuuttia. ChIP suoritettiin sydänkudoksesta valmistetusta kromatiinista ilmoitetuilla vasta-aineilla käyttäen SimpleChIP® Plus Sonication Chromatin IP Kit #56383:a. Rikastettu DNA kvantifioitiin reaaliaikaisella PCR:llä käyttäen ilmoitettujen geenien alukkeita (oikea paneeli). Immunoprecipitoidun DNA:n määrä kussakin näytteessä esitetään normalisoituna signaalina negatiiviseen lokukseen GAPDH, joka on yhtä kuin 1.
6.2 Miten kromatiini fragmentoidaan?
Kromatiinin fragmentointi on välttämätöntä ChIP-kokeen onnistumisen kannalta. Kromatiinin fragmentointi on välttämätöntä kromatiinin liuottamiseksi ja mahdollistaa sen koprezipitaation. Lisäksi ChIP-määrityksen resoluutio riippuu kromatiinin fragmentoinnista, sillä DNA-fragmentin koko määrittää ChIP-määrityksen resoluution.
Ensymaattisessa digestoinnissa käytetään mikrokokkinukleaasia (MNaasi), joka pilkkoo kaksijuosteista DNA:ta nukleosomien välistä kromatiinifragmenttien tuottamiseksi. Täydellinen MNaasi-digestio tuottaa 150 emäsparin DNA-fragmentteja (mono-nukleosomeja), mutta epätäydellinen digestio tuottaa 150-750 emäsparin DNA-fragmentteja (mono-, di- ja tri-nukleosomeja). Sonikointi käyttää mekaanista voimaa kromatiinin pirstomiseen. Sonikaatio pirstoo kromatiinia nukleosomien välistä ja sisällä, jolloin syntyy kromatiinifragmentteja 150-1000 emäsparin välillä.
X-ChIP:ssä kromatiinin leikkaamiseen käytetään joko entsymaattista digestiota tai sonikaatiota. Sonikaatio-ChIP-protokollan sonikaatio-olosuhteet on määritettävä empiirisesti, koska ne vaihtelevat solutyypeittäin ja koeolosuhteittain. Digestio-olosuhteet ovat yhdenmukaisempia eri solutyypeissä ja kudoksissa, mutta kromatiinifragmenttien koko olisi silti analysoitava ennen IP:tä.
N-ChIP:ssä kromatiinin pirstomiseen käytetään nukleaaseja proteiinien sitoutumisen ylläpitämiseksi kiinnittymättömissä näytteissä. Nukleaasifragmentointi olisi myös määriteltävä empiirisesti, jotta kromatiinin ylidigestoituminen voidaan minimoida.
6.3 Miksi käyttää entsymaattista digestointia ChIP:ssä?
Nukleaasifragmentointia on käytettävä N-ChIP:ssä, koska proteiinit eivät ole ristisilloittuneet DNA:han ja sonikaatioon perustuvassa fragmentoinnissa käytettävät ankarat olosuhteet johtaisivat siihen, että kromatiinivalkuaisproteiineja irtoaisi dissosiaatioon DNA:sta. N-ChIP on ihanteellinen histoniproteiinien ja DNA:n välisten vuorovaikutusten analysointiin, koska histonin ja DNA:n välinen sitoutuminen on hyvin vahvaa ja vakaata. N-ChIP ei kuitenkaan sovellu hyvin transkriptiotekijöiden ja kofaktoreiden kromatiinin sitoutumisen analysointiin.
Kromatiinin fragmentointiin X-ChIP:ssä voidaan käyttää joko entsymaattista pilkkomista tai sonikointia. Entsymaattisen digestion etuihin kuuluvat fragmentaation johdonmukaisuus ja miedot fragmentointiolosuhteet (vähemmän lämpöä ja detergenttiä), jotka säilyttävät paremmin kromatiinin ja vasta-aineepitooppien eheyden, jolloin transkriptiotekijä- ja kofaktorisidonnaisen kromatiinin immuunirikastuminen lisääntyy.
6.4 Miksi käyttää sonikointia kromatiinin fragmentoimiseksi ChIP:iä varten?
Toisin kuin kromatiinin fragmentoiminen, joka saavutetaan entsymaattisen digestion avulla, sonikointi perustuu mekaanisiin voimiin, joiden avulla voidaan fragmentoida kromatiini pienempiin kappaleisiin. Kromatiinifragmenttien ihanteellinen koko immuunirikastusta varten on 200 ja 1000 emäsparin välillä. Sonikointi on perinteinen menetelmä, jota käytetään kromatiinin pirstomiseen, ja se voidaan suorittaa perinteisellä koettimen sonikaattorilla tai kehittyneemmillä vesikylpy-sonikaattoreilla, jotka mahdollistavat tarkemmin kohdennetun sonikoinnin. Sonikointi tuottaa todella satunnaistettuja kromatiinifragmentteja; se vaatii kuitenkin laajaa optimointia eri solulinjoissa ja kudoksissa, ja sitä on vaikea toistaa kokeesta toiseen. Vaatimus korkeista detergenttipuskureista ja sonikaation aikana syntyvä lämpö voivat vahingoittaa sekä kromatiinin että vasta-aineepitooppien eheyttä kromatiiniproteiineissa.
6.5 Kromatiinin sonikaation optimointi ChIP:tä varten
Sonikaatioon perustuvassa kromatiinifragmentoinnissa käytetään perinteisesti korkeita detergenttipuskureita ja syntyy lämpöä, jotka molemmat voivat vahingoittaa sekä kromatiinin eheyttä että vasta-aineiden epitooppeja. Siksi kromatiinin fragmentointiin käytettävän sonikaation määrä on määritettävä kokeellisesti eri solulinjoille ja kudoksille. Kromatiinin vaurioitumisen minimoimiseksi on määriteltävä ja käytettävä 150-1000 emäsparin DNA-fragmenttien tuottamiseen tarvittava vähimmäismäärä sonikointia.
Ennen kuin ryhdytään täydelliseen ChIP-määritykseen, johon liittyy myöhemmän vaiheen analyysi qPCR:llä, DNA-sirulla tai NGS:llä, kromatiininäytteiden, joita on sonikoitu eri pituisia aikoja, analyysiin olisi käytettävä geelielektroforeesia. Fragmenttien koko riippuu sonikointiajasta – fragmenttien koko pienenee sonikointiajan kasvaessa. Tiedot viittaavat kuitenkin siihen, että pidemmät sonikointiajat eivät johda parempiin tuloksiin. Siksi puhdistetun immunoprecipitoidun DNA:n ajaminen geelillä ja ihanteellisen fragmenttikoon määrittäminen on suoraviivainen menetelmä, jolla voidaan määrittää halutun DNA-koon saavuttamiseksi tarvittava vähimmäissonikointimäärä ja välttää kromatiinin tarpeetonta vaurioitumista.

Entsyymillä pilkottu kromatiini ajettiin agaroosigeelillä. Kaistalla 1 näkyy kromatiini, joka on alidigestoitunut. Kaistalla 2 näkyy oikein sulatettu kromatiini ja kaistalla 3 kromatiini, joka on ylidigestoitu.
Ensi-entsyymillä sulatettu kromatiini ajettiin agaroosigeelillä. Kaistalla 1 näkyy kromatiini, joka on vajaasti sulatettu. Lane 2 näyttää oikein sulatettua kromatiinia, ja Lane 3 näyttää kromatiinia, joka on ylidigestoitu.
6.6 Miten valitset vasta-aineen ChIP:tä varten?
Sopivan vasta-aineen valitseminen ChIP-kokeeseen on olennainen osa sen onnistumista. ChIP-kokeessa käytettävien vasta-aineiden tulisi olla spesifisiä kiinnostavalle proteiinille ja niillä tulisi olla korkea affiniteetti antigeeniin. Paras valinta vasta-aineeksi ChIP- tai ChIP-seq-kokeeseen on ChIP- tai ChIP-seq-validoitu vasta-aine. Jos kiinnostavalle geenille ei ole saatavilla ChIP-validoitua vasta-ainetta, seuraavaksi paras valinta on vasta-aine, joka on validoitu IP:ssä. On tärkeää huomata, että kaikki IP-validoidut vasta-aineet eivät toimi ChIP:ssä eivätkä kaikki ChIP-validoidut vasta-aineet toimi ChIP-seq:ssä. Lisäksi mitä enemmän vasta-ainetta on validoitu muissa sovelluksissa, kuten western-, IP-, IF-, flow- ja IHC-menetelmissä, sitä varmemmin voidaan luottaa vasta-aineen suorituskykyyn ja spesifisyyteen. Vasta-aineen validointia on seurattava vasta-aineen optimaalisen konsentraation empiirinen määrittäminen sekä IP:n pesuolosuhteet.
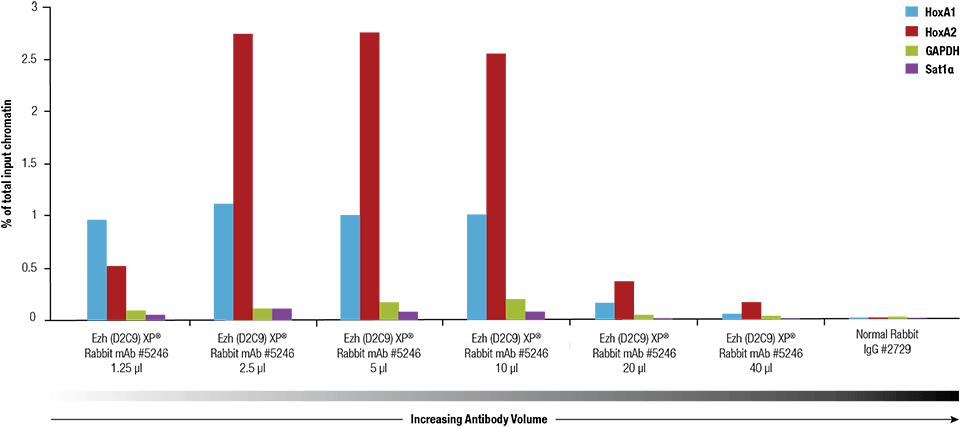
Ezh2 (D2C9) XP® Rabbit mAb #5246 titrattiin SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005:llä ristisilloitetulle kromatiinille, joka oli valmistettu 4 x 106:sta NCCIT-solusta.
Ezh2 (D2C9) XP® Rabbit mAb #5246 titrattiin käyttämällä SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005:tä ristisilloitetulla kromatiinilla, joka oli valmistettu 4 x 106 NCCIT-solusta.
6.7 Immunoprecipitaation suorittaminen
Avasta-aineita käytetään kiinnostuksen kohteena olevan proteiinin ja siihen sidotun DNA:n vangitsemiseen. Vasta-ainekonsentraatio on määritettävä empiirisesti; yleinen lähtökohta on 0,5-2,0 μg vasta-ainetta, jota käytetään 10 μg kromatiini-DNA:ta kohti (vastaa noin 4 x 106 solua). Myös puskurien tiukkuus ja pesuajat on määritettävä empiirisesti, koska ne riippuvat vasta-aineen affiniteetista kohdeantigeeniinsä. Tyypillisesti vasta-aine:kromatiini -inkubaatioita tehdään 2 tunnista yön yli.
Vasta-aine-antigeeni (+DNA) -kompleksit kiinnitetään affiniteetin avulla vasta-ainetta sitovaan hartsiin. ChIP-kokeissa tämä hartsi koostuu tyypillisesti ChIP-luokan magneetti-, sefaroosi- tai agaroosihelmistä, jotka on konjugoitu proteiiniin A ja/tai proteiiniin B. Vasta-aineet sitoutuvat proteiini A ja/tai proteiini G -helmiin eri affiniteeteilla riippuen lajista, jossa ne on kehitetty, ja niiden raskaan ketjun IgG-alatyypistä. Helmet inkuboidaan tyypillisesti vasta-aine:kromatiinin kanssa 2-4 tuntia.
Pesuvaiheita tarvitaan ei-vasta-aineeseen sitoutuneen kromatiinin poistamiseksi, minkä jälkeen ristisidokset käännetään (X-ChIP:tä varten) ja DNA puhdistetaan. Lisäksi on suoritettava IgG-kontrolli-IP taustan (signaali:kohina) määrittämiseksi. Positiiviset kontrollivasta-aineet (esim. histoni H3:n kokonaismäärä) ja/tai positiiviset kontrolli-qPCR-alukkeet (tunnettujen positiivisten ja negatiivisten kohdeproteiinien sitoutumislokusten osalta) on myös otettava mukaan epäspesifisen sitoutumisen määrittämiseksi. Optimaalisten tulosten saamiseksi kromatiinin IP:n QC qPCR:llä olisi suoritettava ennen myöhemmän NGS-analyysin suorittamista.
6.8 Miten kromatiini eluoidaan proteiini A/G -helmistä
Kromatiini eluoidaan proteiini A/G -helmistä käyttämällä detergenttiä ja lämpöä. Matalan nopeuden ”vorteksointia” tai sekoittamista tarvitaan helmien pitämiseksi suspensiossa ja kromatiinin eluoitumisen lisäämiseksi.
6.9 Miten kromatiinin ristisidokset käännetään
Ristisidokset käännetään korkealla lämmöllä ja korkealla suolapitoisuudella (molemmat ovat elintärkeitä komponentteja). Lisäksi lisätään proteinaasi K:ta siihen liittyvien kromatiiniproteiinien pilkkomiseksi ja lisätään vasta-aineita, mikä mahdollistaa tehokkaamman DNA:n puhdistamisen myöhemmässä vaiheessa.
6.10 Miten DNA:ta puhdistetaan
Kromatiinin ristisilloitusten poistamisen jälkeen DNA puhdistetaan joko klassisilla fenoli-kloroformi-menetelmillä, joita seuraa etanolin saostusmenetelmä, tai pylvääseen perustuvilla DNA:n puhdistussarjoilla.
Miten rikastettua DNA:ta analysoidaan
Kun DNA on puhdistettu, voidaan suorittaa useita jatkoanalyysejä, kuten ChIP-PCR, ChIP-qPCR, ChIP-chip ja ChIP-seq.
7.1 ChIP-PCR- ja ChIP-qPCR-analyysit
ChIP-PCR- ja ChIP-qPCR-analyysit soveltuvat parhaiten yhden geenin analyyseihin, ja niiden avulla voidaan monistaa ja kvantifioida spesifisiä DNA-fragmentteja nopeasti ja kustannustehokkaasti.
7.2 ChIP-chip-analyysi
ChIP-chip-analyysi, jossa hyödynnetään laatoitettuja DNA-mikrosirusiruja koko genomin laajuisen, korkean resoluution kartan luomiseksi proteiinien sitoutumisesta ja proteiinimodifikaatiosta.
7.3 ChIP-seq-analyysi
ChIP-seq-analyysissä käytetään tavanomaista NGS-tekniikkaa, jolla puhdistettua DNA:ta sovitetaan yhteen aiemmin annotoitujen kokonaisten genomien kanssa, jotta voidaan yksilöidä koko genomin laajuiset proteiinien sitoutumisprofiilit.