Dies sind Notizen aus der Vorlesung 4 des Zellbiologie-Kurses von Harvard Extension.
Der sekretorische Weg bezieht sich auf das endoplasmatische Retikulum, den Golgi-Apparat und die Vesikel, die zwischen ihnen verkehren, sowie auf die Zellmembran und die Lysosomen. Er wird als „sekretorisch“ bezeichnet, weil er der Weg ist, über den die Zelle Proteine in die extrazelluläre Umgebung abgibt. Aber wie üblich erzählt die Etymologie nur einen Bruchteil der Geschichte. Dieser Weg verarbeitet auch Proteine, die membrangebunden werden (sei es in der Zellmembran oder in den ER- oder Golgi-Membranen selbst), sowie lysosomale Enzyme und auch alle Proteine, die ihr Leben im sekretorischen Weg selbst verbringen werden. Das Cytosol und das „Lumen“ (die Flüssigkeit, die den Sekretionsweg füllt) sind unterschiedliche chemische Umgebungen, die sich normalerweise nicht vermischen. Das Zytosol ist reduktiv (wenn man sich im Zytosol befindet, trifft man ständig auf Moleküle, die einem Elektronen anbieten wollen), und das ER, die Golgi und die extrazelluläre Umgebung sind oxidativ (Moleküle kommen ständig auf einen zu und verlangen nach Elektronen). Siehe Redox, falls Sie immer noch verwirrt sind. Daraus ergeben sich unterschiedliche Bedingungen für die Proteinfaltung: Disulfidbindungen bilden sich z. B. normalerweise nur unter oxidativen Bedingungen. Außerdem können verschiedene Proteine nur im sekretorischen Weg oder nur im Zytosol leben. Der Sekretionsweg bietet der Zelle einen Weg, um Dinge zu handhaben, die im Zytoplasma nicht gut aufgehoben sind und/oder am nützlichsten sind, wenn sie in einem spezialisierten Kompartiment mit ihren gewünschten Interaktionspartnern konzentriert gehalten werden. Hepatozyten (in der Leber) lagern Medikamente und Toxine im glatten ER ab und bauen sie dort ab, um sie aus dem Körper auszuscheiden. Der sekretorische Weg ist nicht zusammenhängend, sondern jede Bewegung zwischen seinen Komponenten findet in kleinen aufgeblasenen Mikrokosmen seiner eigenen chemischen Welt statt, die Vesikel genannt werden.
Viele Proteine, die den sekretorischen Weg durchlaufen, kommen nie mit dem Cytosol in Berührung – mit Ausnahme der Teile von Membranproteinen, die auf der cytosolischen Seite herausragen. Viele von ihnen benötigen Chaperone, die bei der Faltung helfen, und/oder eine ganze Reihe von posttranslationalen Modifikationen, um für ihre native Funktion bereit zu sein, und der sekretorische Weg ist darauf spezialisiert, ihnen all das zu bieten.
Der heutige Vortrag wird sich darauf konzentrieren, wie Proteine in das ER übersetzt werden und wie sie (in Vesikeln) zwischen dem ER, Golgi und anderen Zielen reisen. Dies wird im Video Life of the Cell sehr schön dargestellt:
Das endoplasmatische Retikulum ist die erste Stufe des sekretorischen Weges. Seine Membran schließt sich an die äußere Kernmembran an, aber es ist nicht klar, warum das wichtig ist, denn Proteine beginnen ihr Leben nicht im Kern. Vielmehr treiben die mRNAs im Zytoplasma umher, bis sie von einem Ribosom aufgenommen werden, das sie übersetzen möchte. Bei der „posttranslationalen Translokation“ wird das neue Protein nach der Übersetzung in das ER verschoben. Bei dem interessanteren Phänomen der „kotranslationalen Translokation“ beginnt das Ribosom mit der Übersetzung wie bei jedem anderen Protein, stößt aber irgendwo in den ersten 16 bis 30 Aminosäuren auf ein Signalpeptid (auch Signalsequenz genannt). Dieses Signalmotiv besteht häufig aus einer positiv geladenen Aminosäure, gefolgt von 6-12 hydrophoben Aminosäuren. Dieses Motiv wird von einem Signalerkennungspartikel (SRP, ein „Ribonukleoprotein“ oder hybrides RNA/Protein-Molekül) erkannt, das daran bindet und das Ribosom an der Fortsetzung der Übersetzung hindert. Die Translation wird gestoppt, bis der Ribosom/SRP-Komplex auf der ER-Membran auf einen SRP-Rezeptor trifft. Beim Zusammentreffen binden SRP und sein Rezeptor jeweils ein GTP-Molekül in der ER-Membran, was ihre Interaktion offenbar verstärkt. Zufälligerweise geschieht dies alles in der Nähe eines Sec61-Translocons – eines Proteinkomplexes, der einen Kanal bildet, der die ER-Membran durchquert. Der Translocon ist eigentlich ein Komplex aus drei verschiedenen Proteinen (Gene: SEC61A1 oder SEC61A2, SEC61B, SEC61G), von denen die Untereinheit Sec61a zehn membranüberspannende a-Helices besitzt, die den Kanal bilden. Sobald das Ribosom an der Membran angedockt ist, setzt es die Translation fort und schiebt das Signalpeptid und schließlich das gesamte Protein durch den Kanal in das ER-Lumen. Wenn die Translation stoppt, hydrolisieren SRP und SRP-Rezeptor ihr GTP, um sich gegenseitig und die Ribosomenfracht freizugeben (dies erfordert die Energie von GTP, da die ursprüngliche Bindung bergab erfolgte), eine Signalpeptidase spaltet das Signalpeptid vom entstehenden Protein ab, und das Protein ist frei, um mit der Faltung im ER zu beginnen.
Bei einigen ER-Proteinen sind noch weitere Akteure beteiligt. Die Oligosaccharid-Transferase, die Glykosylgruppen an Asparagine im naszierenden Protein anhängt, ist Teil des Translocon-Komplexes und führt die Glykosylierung durch, während das neue Protein noch übersetzt wird. Obwohl wir die Glykosylierung also als „posttranslationale Modifikation“ bezeichnen, wird sie in diesem Fall tatsächlich während der Translation vorgenommen. Außerdem müssen einige Proteine, um ihre richtige Struktur zu erhalten, vollständig übersetzt werden, bevor sie mit der Faltung beginnen können – würde der N-terminale Teil mit der Faltung beginnen, sobald er in das Lumen eintritt, hätte er die falsche Gesamtstruktur. Um dies zu verhindern, bindet das Chaperon BiP manchmal das Protein, um es für eine Weile ungefaltet zu halten. Stellen Sie sich BiP als einen weiteren Pac-Man vor, der auf das Protein beißt, um es linear zu halten, wie Hsc70 beim mitochondrialen Targeting-Prozess (siehe letzte Woche).
Hier ist ein Video davon:
Die ersten paar Minuten zeigen das oben beschriebene grundlegende Szenario. Dann geht es weiter zu einem komplexeren Szenario, das ich in einer Minute vorstellen werde. Zu Ihrer Information: Das Video zeigt zwei „kontroverse“ Dinge, die in der obigen Beschreibung nicht enthalten sind: (1) das Signalpeptid, das in der Membran abgebaut wird, und (2) ein „Pfropfenprotein“, das den Kanal vor/nach der Translation verschließt. Über diese beiden Dinge sind sich noch nicht alle Wissenschaftler einig.
Alle Proteine, von denen wir wissen, dass sie den sekretorischen Weg durchlaufen, wurden dort durch Lokalisierungsexperimente ermittelt, um zu sehen, wo in der Zelle ein Protein liegt. Eine merkwürdige Tatsache über das ER ist, dass man die Zelle in einen Mixer geben kann und sich das ER danach einfach wieder mit sich selbst verbindet und kleine „Mikrosomen“ bildet, die nicht mit dem Zellkern verbunden sind, sondern zusammenhängende Blasen aus ER bilden. Dann können Sie mit Proteasen – die Proteine abbauen – und Detergenzien – die die ER-Membran auflösen – spielen. Unter der Annahme, dass das Protein, das Sie interessiert, translatiert wird, können Sie prüfen, ob es (1) die Behandlung mit Proteasen überlebt, aber (2) die Behandlung mit Proteasen und Detergenzien nicht überlebt, dann handelt es sich um ein Protein des sekretorischen Weges. Die Logik ist, dass es im Fall (1) im ER geschützt war, aber im Fall (2) haben Sie das ER aufgelöst, so dass es von der Protease gefressen wurde. All dies setzt voraus, dass man über einen Antikörper oder eine andere Möglichkeit verfügt, um festzustellen, ob das betreffende Protein nach diesen Behandlungen noch vorhanden ist.
Mit Hilfe solcher Techniken hat man auch herausgefunden, dass nur 70 Aminosäuren eines neuen Proteins übersetzt werden können, bevor es für dieses Protein zu spät ist, im ER zu landen. Zur Erinnerung: Das Signalpeptid befindet sich in den ersten 16-30 Aminosäuren, und die Translokation in das ER hängt vom Vorhandensein des SRP ab. Ribosomen übersetzen mit einer vorhersagbaren Geschwindigkeit, also hat man die Ribosomen dazu gebracht, einige mRNA zu übersetzen und dann eine bestimmte Zeit gewartet, bevor man SRP hinzufügte, um zu sehen, wie viel Übersetzung stattfinden konnte, bevor SRP seine Aufgabe nicht mehr erfüllen konnte.
Der SRP-Rezeptor und die Sec61-Proteine sind ER-Membranproteine – und es gibt viele andere ER-Membran-, Golgi-Membran- und Lysosomen-Membranproteine. Tatsächlich werden sogar die Membranproteine (siehe Klasse 02) der Zellmembran im sekretorischen Weg prozessiert. Viele dieser Proteine haben mehrere Dutzend Transmembrandomänen (jeweils 20-25 hydrophobe Aminosäuren), die in der richtigen Reihenfolge und Ausrichtung eingefügt werden müssen (z. B. müssen die Ionenkanäle und Transporter in die richtige Richtung zeigen, d. h. in die Zelle hinein und aus der Zelle heraus). Dementsprechend gibt es eine Reihe von ausgeklügelten biologischen Mechanismen, um diese Proteine korrekt in die Membran einzubauen. Dies wird in der zweiten Hälfte des obigen Videos dargestellt.
Hier also eine Tautologie: Einige Proteine haben eine topogene Sequenz, die ihre Ausrichtung in der Membran bestimmt. Diese Sequenz besteht aus zwei Arten von Signalsequenzen:
- eine Stop-Transfer-Sequenz (aus irgendeinem Grund mit STA abgekürzt) ist eine 22-25 hydrophobe Aminosäuresequenz irgendwo in der Mitte des Proteins, die eine Alpha-Helix bildet. Wenn sie angetroffen wird, wird sie in die Membran geschoben, und die Translation des restlichen Proteins wird im Zytosol fortgesetzt. Dadurch wird die Translokation in das ER, die durch das Signalpeptid am Anfang (N-Terminus) des Proteins eingeleitet wurde, gewissermaßen „rückgängig“ gemacht.
- Eine Signalankersequenz (abgekürzt SA) ist ebenfalls eine 22-25aa hydrophobe Alpha-Helix, jedoch mit einer Reihe von ~3 positiv geladenen Aminosäuren links oder rechts davon. Wie das Signalpeptid wird sie von SRP erkannt, das das Ribosom zum ER bringt. Doch im Gegensatz zum Signalpeptid wird diese alpha-helikale Sequenz in die ER-Membran eingebaut. Die Ausrichtung der Insertion wird durch die 3 positiv geladenen Aminosäuren bestimmt. Die positiven Ladungen müssen immer auf der zytosolischen Seite enden, d.h. wenn sie nach (d.h. C-terminal von) der hydrophoben Sequenz kommen, endet das Protein mit seinem C-terminalen Ende in Richtung Zytosol, aber wenn sie vor (d.h. N-terminal von) der hydrophoben Sequenz kommen, endet das Protein mit seinem N-Terminus in Richtung Zytosol.
Mit diesen beiden Signalen als Bausteinen kann man sich ein Protein mit einer Reihe von Stop-Transfer- und Signalankersequenzen vorstellen, um eine ganze Reihe von hin- und hergehenden Transmembrandomänen zu schaffen, die wie von einer Nähmaschine in die Membran eingenäht werden. Man hat die Membranproteine in fünf Kategorien eingeteilt:
- Typ I hat nur ein Signalpeptid und dann einen Stop-Transfer in der Mitte. Daher endet es mit seinem (hydrophilen) N-Terminus im Lumen, seiner (hydrophoben) Mitte in der Membran und seinem (hydrophilen) C-Terminus im Cytosol.
- Typ II beginnt nicht mit einem Signalpeptid. Es beginnt wie jedes andere Protein, hat aber in der Mitte eine Signalanker-Sequenz, bei der die +++ Aminosäuren zuerst kommen und die hydrophobe Reihe danach. Dies führt dazu, dass das Protein auf halbem Weg durch die Translation transloziert wird, wobei der bereits translozierte N-terminale Teil in das Cytosol ragt (da die +++ im Cytosol bleiben müssen) und der nun beginnende, zu translozierende C-terminale Teil direkt in das ER übersetzt wird. So endet es transmembral mit dem C-Terminus im ER und dem N-Terminus im Zytosol – im Gegensatz zu Typ I.
- Typ III ist wie Typ II – kein Signalpeptid, nur ein Signalanker in der Mitte, aber in diesem Fall kommen die +++ nach der hydrophoben Sequenz, was die Orientierung umkehrt. Somit befindet sich der N-Terminus im ER und der C-Terminus im Zytosol. Das Gegenteil von Typ II und letztlich dasselbe wie Typ I, obwohl es auf andere Weise dorthin gelangt ist – es hat kein Signalpeptid, das im ER abgespalten wird.
- Typ IV oder „Multipass“-Proteine haben eine abwechselnde Reihe von Signalsequenzen und Stopp-Transfersequenzen. Das sind eindeutig mehr als ein „Typ“, aber sie sind nicht annähernd so vielfältig, wie es Ihre kombinatorische Vorstellungskraft zulassen würde. Die Ausrichtung der ersten Signalsequenz bestimmt, ob der N-Terminus im Zytosol oder im ER landet, und die Gesamtzahl der Stop-Transfer- und Signalankersequenzen bestimmt, wo der C-Terminus landet: eine gerade Zahl = auf der gleichen Seite wie der N-Terminus, eine ungerade Zahl = auf der gegenüberliegenden Seite wie der N-Terminus. Die STA- und SA-Sequenzen müssen sich strikt abwechseln, mit der Ausnahme, dass man mit zwei Signalankersequenzen beginnen kann, wenn die erste mit dem N-Terminus in das Zytosol ausgerichtet ist. Um dieses Kategorisierungsschema ins Lächerliche zu ziehen, hat man einige unvollständig definierte Subtypen des Typs IV definiert, wobei Typ IVa N-terminal im Zytosol liegt (also wie ein Typ-II-Protein beginnt) und Typ IVb N-terminal im Lumen liegt (es beginnt wie ein Typ-III-Protein, hat dann aber eine weitere SA-Sequenz, die es zurück ins ER bringt). GLUT1 aus Klasse 02 ist ein Typ IVa.
- GPI-verankerte Proteine, die den fünften Typ darstellen, aber nicht als Typ V bezeichnet werden, beginnen mit einem Signalpeptid und enden mit einem hydrophoben C-Terminus, der in der Membran eingebettet bleibt. Dieses hydrophobe Ende wird abgespalten und durch GPI ersetzt, das ebenfalls in der Membran eingebettet bleibt. PrP ist eines dieser Proteine – mehr dazu später.
Wir haben jetzt besprochen, wie Proteine in das ER-Lumen gelangen oder die ER-Membran überspannen können. Die meisten Proteine verlassen das ER innerhalb weniger Minuten und werden in Vesikeln zum Golgi und später zur Ausscheidung, zu den Lysosomen oder zur Zellmembran transportiert. Die Vorwärtsbewegung wird als anterograder Transport bezeichnet, die Rückwärtsbewegung vom Golgi zum ER als retrograder Transport.
Beide Arten des Transports finden in membrangebundenen Vesikeln statt. Diese lösen sich von der Membran des Ortes, von dem sie kommen, und verschmelzen später mit der Membran des Ortes, zu dem sie gehen – wunderschön dargestellt bei ~2:25 im obigen Life of the Cell Video. Der Körper, aus dem sich die Vesikel bilden, ist das „Donor-Kompartiment“, und das Ziel, mit dem sie später verschmelzen, ist das „Akzeptor-Kompartiment“.
Der Knospungsprozess erfordert, dass G-Proteine in der Membran Hüllproteine rekrutieren. Konkret rekrutiert das G-Protein Sar1 (Gen: SAR1A) für den anterograden Transport COPII („cop two“); für den retrograden Transport rekrutiert ein ARF-G-Protein COPI (ausgesprochen „cop one“). Diese G-Proteine werden für diese Aufgabe aktiviert, wenn GEF sie mit GTP belädt und GDP ersetzt.
Die Schritte des anterograden Transports sind also zum Beispiel folgende:
- Sec12-GEF (Sec steht für sekretorisch) belädt Sar1 mit GTP. Wenn Sar1 an GDP gebunden ist, schwimmt es einfach im Donor-Kompartiment herum, aber wenn es an GTP gebunden ist, erfährt es eine Konformationsänderung, die bewirkt, dass sein sonst vergrabener N-terminaler hydrophober Schwanz herausragt, wodurch es in der Membran stecken bleibt, wo sich dann COPII-Proteine anzusammeln beginnen, weil sie diesen Schwanz wirklich mögen.
- Die COPIIs beginnen zu polymerisieren und haben aufgrund ihrer Konformation eine intrinsische Vorliebe für Krümmung, so dass ihre Anhäufung beginnt, die Knospung zu ermöglichen. Gleichzeitig werden membrangebundene Proteine, die transportiert werden müssen – identifiziert durch eine DXE-Aminosäuresequenz (d.h. Aspartat-irgendwas-Glutamat), die eine Bindungsstelle in ihrem zytosolischen Teil bildet -, in das neu entstehende Vesikel rekrutiert. Membrangebundene Proteine fungieren als Rezeptoren und rekrutieren lumenale Proteine, die für den Golgi gebunden sind, um sich in den konkaven Raum zu begeben, wo sie in das Vesikel gelangen, sobald es sich bildet.
- Wenn genügend COPII angekommen sind, springt das Vesikel ab, woraufhin Sar1 sein GTP hydrolysiert und damit die Energie bereitstellt, um seinen hydrophoben Schwanz in sich selbst zurückzusaugen und die COPII abzuschneiden. Das Vesikel ist nun vom Donor-Kompartiment getrennt.
- Aus schlecht erklärlichen (oder schlecht verstandenen?) Gründen zerfällt nun die Hülle der COPIIs und legt Rezeptoren unter der Hülle frei, die die Ausrichtung des Vesikels steuern. Sobald das Vesikel an seinem Ziel angekommen ist, interagiert das in der Vesikelmembran eingebettete Rab-GTP mit einem Rab-Effektor, der in der Membran des Akzeptorkompartiments eingebettet ist. Ein Seitenblick wird ausgetauscht, das Interesse ist geweckt. Bald wird das Vesikel mit der Membran verschmelzen.
- SNARE-Proteine, die sowohl auf der Vesikel- als auch auf der Zielmembran vorhanden sind (V-SNARE bzw. T-SNARE), interagieren, um die Membranen noch näher zusammenzubringen. In diesem Beispiel betrachten wir VAMP (die VAMP_-Gene) als V-SNARE und Syntaxin (die STX__-Gene) und SNAP25 (SNAP25-Gen) als die T-SNAREs. Syntaxin und SNAP25 sind beides Membranproteine; Syntaxin hat eine Alpha-Helix und SNAP25 hat zwei, alle auf der zytosolischen Seite. Die Alpha-Helices sind für die Interaktion mit VAMP verantwortlich. Die Alpha-Helices der gegenüberliegenden Seiten haben eine extrem starke Affinität zueinander und bringen die Membranen so nahe zusammen, dass sie verschmelzen. Sobald dies geschehen ist, sind zwei Proteine erforderlich, um die V-SNAREs und T-SNAREs wieder voneinander zu trennen: NSF (Gen: NSF; steht für NEM sensitive factor) und alpha-SNAP (Gen: NAPA), ein lösliches NSF-Anhängeprotein. NSF ist eine ATPase und verbrennt ATP, um den energetisch aufwändigen Abbau des Komplexes voranzutreiben.
Nun zum retrograden Transport. Warum gibt es überhaupt einen retrograden Transport? Hier ist eine nicht erschöpfende Liste einiger Gründe:
- Einige Membranproteine beginnen ihr Leben im ER, müssen im Golgi modifiziert werden, aber dann wieder zurück ins ER gelangen. Sie tun dies mit einer KKXX-Aminosäuresequenz.
- Es gibt auch eine KDEL-Aminosäuresequenz am C-Terminus einiger Lumenproteine, die dafür sorgen soll, dass sie im ER bleiben, aber das ist nicht perfekt – manchmal landen sie im Golgi, und in diesem Fall werden sie über einen retrograden Transport, der von der KDEL-Sequenz zur Erkennung abhängt, zurück in den ER gebracht. Der Mechanismus ist ziemlich raffiniert – die Proteine, die KDEL erkennen und daran binden, tun dies nur bei niedrigem pH-Wert, und der pH-Wert des Golgi ist niedriger als der des ER, so dass sie KDEL im Golgi binden und dann freisetzen, wenn sie sich wieder im neutraleren pH-Wert des ER befinden.
- Überlegen Sie auch, dass alle Proteine, die am anterograden Transport beteiligt sind – die V-SNARES, Rab, usw. – müssen zum ER zurückkehren, damit sie alles noch einmal machen können, so wie der Bus am Ende des Tages zum Busdepot zurückkehren muss.
- Wie wir gleich sehen werden, gibt es im Golgi mehrere Stufen, die von der Hinzufügung von Enzymen von weiter unten abhängen.
Der Prozess des retrograden Transports unterscheidet sich nicht so sehr vom anterograden. Er verwendet ARF anstelle von Sar1, COPI anstelle von COPII, aber er funktioniert genauso: Mit GTP beladenes ARF lässt seinen hydrophoben Schwanz in die Membran einkleben und erregt so die Aufmerksamkeit von COPIs. COPI hat zwei Komponenten, COPIalpha und COPIbeta, die beide mit dieser KKXXX-Sequenz interagieren, um membrangebundene Proteine für den retrograden Transport zu rekrutieren. Einige Proteine haben auch eine RR-Sequenz (irgendwo im Protein), die sie für den retrograden Transport kennzeichnen kann.
Der Golgi-Apparat ist nicht zusammenhängend. Er besteht aus einer Reihe von getrennten Unterkompartimenten, die als Säcke oder Zisternen bezeichnet werden. Die verschiedenen Kompartimente haben unterschiedliche Eigenschaften, und die Proteine besuchen sie in einer bestimmten Reihenfolge. In der Reihenfolge vom ER zur Zellmembran werden die Golgi-Kompartimente als cis-, medial-, trans- und trans-Golgi-Netzwerk bezeichnet. Jedes Kompartiment verfügt über verschiedene Enzyme, die Proteine modifizieren, und die Modifikationen müssen in einer bestimmten Reihenfolge erfolgen, daher die Notwendigkeit einer Reihe von gestapelten Kompartimenten.
Aber wenn Proteine im Golgi reifen, ist es nicht so, dass sie in Vesikeln aus einem Kompartiment austreten und in das nächste wandern. Vielmehr bewegt sich das Kompartiment, in dem sie sich bereits befinden, nach außen und „reift“, während neue Enzyme (von weiter unten in der Golgi-Kette) durch retrograden Transport hinzugefügt werden. Seltsam, oder? Es ist in etwa so, als ob man, anstatt von der Grundschule über die Mittelschule zur Oberschule zu wechseln, während der gesamten Kindheit und Jugend in einem Schulgebäude bleiben würde und jedes Jahr neue Lehrbücher und Lehrer hinzukämen, um die Schule an das Niveau anzupassen, das man und seine Klassenkameraden inzwischen erreicht haben. So sehen die Golgi aus, wenn sie sich bewegen und entwickeln:
Es gibt also (wenig oder) keinen anterograden Transport innerhalb der Golgi, aber viel retrograden Transport, um jede neue Runde von Enzymen hineinzubringen. Wenn Proteine schließlich den gesamten K-12-Lehrplan des Golgi-Netzwerks absolviert haben, werden sie transportiert, um zu ihrem endgültigen Bestimmungsort zu gelangen. Sie werden in einem Vesikel ausgeschieden, das einen der folgenden drei Wege einschlägt:
- Exozytose – Verschmelzung mit der Zellmembran. So werden die Lumenproteine extrazellulär sezerniert, und die Membranproteine werden zu Zellmembranproteinen.
- Sekretorische Vesikel – diese bleiben einfach als Vesikel in der Zelle, bis sie gebraucht werden – wobei „gebraucht“ bedeutet, dass sie schließlich exozytiert werden. In Neuronen werden hier Neurotransmitter gespeichert, bis ein Aktionspotential ihre Ausschüttung in die Synapse erfordert. Im Magen bewahren die Zellen, die Magenenzyme produzieren, diese Enzyme in sekretorischen Vesikeln auf, bis die Nahrungsaufnahme ihre Freisetzung in den Magen auslöst.
- Lysosomen – wohin fehlgefaltete Proteine gehen, um abgebaut zu werden.
Der Transport vom trans-Golgi-Netzwerk zu diesen Zielen unterscheidet sich von den anderen oben besprochenen Transporten und beinhaltet oft Clathrin (CLT__-Gene). Die abspringenden Vesikel haben eine zweischichtige Hülle, mit Adapterprotein-Komplexen (AP) als innerer Schicht und Clathrin als äußerer Schicht. Die Adapterproteine haben ein Zielsignal mit einem YXXh-Motiv (h = Φ = eine beliebige hydrophobe Aminosäure). Clathrin bildet die hier gezeigte sogenannte „Clathrin-Triskelion“-Formation:

(Bild dank Wikimedia Commons Benutzer Phoebus87)
Clathrin ist auch für die Endozytose verantwortlich – das Abspalten von Vesikeln mit extrazellulären Stoffen (und Zellmembranproteinen), die in die Zelle gelangen. Dies wird als Clathrin-vermittelte Endozytose bezeichnet. Rezeptoren in der Zellmembran werden sehr häufig endozytiert: Die gesamte Population der Hormonrezeptoren wechselt etwa jede Stunde, vor allem wenn Hormone aufgenommen werden. Die Aufnahme des Rezeptors in ein Vesikel ist eine Möglichkeit für die Zelle, das eingehende Signal zu unterbrechen, bis es verarbeitet werden kann.
In den Anmerkungen zur Plasmamembran wird kurz auf die zystische Fibrose eingegangen: CFTR ist ein ABC-Transporter, der für das Herauspumpen von Cl- aus der Zelle verantwortlich ist (er lässt auch Na+ hinein). Funktionsverlustmutanten pumpen kein Cl-, wodurch die treibende Kraft für die Osmose entfällt, der Schleim verdickt wird und Atemprobleme entstehen. Es gibt mindestens 127 verschiedene CFTR-Mutanten mit Funktionsverlust (zumindest sind es so viele, auf die Natera testet), die (wenn beide Allele deaktiviert sind) Mukoviszidose verursachen. Die häufigste Mutation ist ΔF508, die ~3 % aller europäischen CFTR-Allele und etwa 70 % der mutierten Allele ausmacht. Der Verlust dieses einen Phenylalanins verändert die Konformation von CFTR, so dass der di-saure Exit-Code (Aminosäuren D565 und D567), der CFTR für exozytotische Vesikel ausrichtet, nicht mehr korrekt exponiert ist und das Protein es nie bis zur Zellmembran schafft.
Diskussionsabschnitt
Im Abschnitt lesen wir Hu 2009, der zeigte, dass Atlastin-Proteine an der Bildung des tubulären ER-Netzwerks beteiligt sind. Der Nachweis erfolgte fast ausschließlich über Protein-Protein-Wechselwirkungen. Ich war überrascht, dass diese Arbeit so viel Aufsehen erregt hat, denn es gibt eine Million Arbeiten, die Protein-Protein-Wechselwirkungen für Huntingtin zeigen, und niemand glaubt ihnen wirklich, und sie haben uns nicht unbedingt näher gebracht, was Huntingtin tut oder was bei der Huntington-Krankheit falsch läuft. Aber anscheinend konnte Hu die Wechselwirkungen der Atlastine mit den Retikulonen ziemlich eindeutig nachweisen, was auf eine Rolle bei der ER-Bildung hindeutet. Es ist hilfreich, dass Hu zusätzlich zu einer physikalischen (bindenden) Interaktion auch eine „genetische Interaktion“ nachweisen konnte. Eine „genetische Interaktion“ (ich musste es nachschlagen) bedeutet, dass „manchmal Mutationen in zwei Genen einen Phänotyp erzeugen, der angesichts der individuellen Auswirkungen jeder Mutation überraschend ist. Dieses Phänomen, das die genetische Interaktion definiert, kann funktionelle Beziehungen zwischen Genen und Signalwegen aufzeigen.“
PrP
Dies ist ein Jahrzehnt alt, so dass einiges veraltet sein mag, aber ich fand Harris 2003 (ft)’s Überblick über die Zellbiologie von PrP äußerst klar und hilfreich. Kim & Hegde 2002 war ebenfalls hilfreich. PrP ist ein Protein des sekretorischen Weges. Seine ersten 22 Aminosäuren (MANLGCWMLVLFVATWSDLGLC) sind ein Signalpeptid, das eine kotranslationale Translokation in das ER bewirkt. Normalerweise wird PrP nur an seinem C-Terminus GPI-verknüpft und ist an der exoplasmatischen Seite der Membran verankert. Die Aminosäuren 111-134 (HMAGAAAAGAVVGGLGGYMLGSAM) sind jedoch eine Art schwache Signalankersequenz (Typ II, wobei die +++ Aminosäuren vor dem Signalanker stehen), die manchmal, aber nicht immer, zu einer Transmembrandomäne wird und den C-Terminus in das Lumen verlagert. Noch verwirrender ist, dass diese Sequenz manchmal einfach als Transmembrandomäne ohne die Inversion enden kann, so dass sich der N-Terminus im Lumen befindet. Es gibt also drei Membrantopologien von PrP: die normale alte GPI-Verankerung und zwei Transmembranausrichtungen, wie in Harris 2003 Abb. 3 dargestellt:
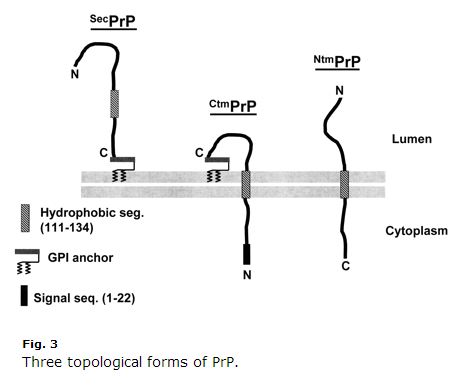
Beachten Sie, wie seltsam CtmPrP ist. Es ist transmembranös, aber auch GPI-verankert, und das N-terminale Signalpeptid wird nie abgespalten. Normalerweise machen die Transmembranformen < 10 % des gesamten PrP aus. Unter bestimmten Laborbedingungen ist der Anteil höher, und zwei der GSS-verursachenden Mutationen (A117V und P105L) erhöhen den Anteil von CtmPrP auf 20-30 % des gesamten PrP. Von diesen drei Formen gibt es zahlreiche Hinweise darauf, dass CtmPrP toxisch ist und dass es bei der Prionenbildung eine Rolle spielen könnte, obwohl die meisten genetischen Mutationen bei Prionenerkrankungen (einschließlich FFI D178N) die Membrantopologie von PrP oder den Anteil von CtmPrP nicht zu beeinträchtigen scheinen.
Nachdem PrP den Golgi passiert hat, ist es für die Zellmembran bestimmt. Harris zufolge bleibt es dort aber nicht einfach sitzen, sondern durchläuft häufig die Clathrin-vermittelte Endozytose und durchläuft die Zelle alle ~60 Minuten, wobei bei jedem Zyklus einige Moleküle abgespalten werden. Kupfer stimuliert diese Endozytose von PrP. Die meisten genetischen Mutationen bei Prionenkrankheiten verändern die Lokalisierung von PrP – bei Vorliegen einer Mutation befindet sich in der Regel weniger PrP auf der Zelloberfläche, während sich mehr im ER ansammelt.