Was ist ChIP?
Die Chromatin-Immunpräzipitation (ChIP) ist eine auf Antikörpern basierende Technologie zur selektiven Anreicherung spezifischer DNA-bindender Proteine zusammen mit ihren DNA-Zielen. ChIP wird eingesetzt, um eine bestimmte Protein-DNA-Interaktion, mehrere Protein-DNA-Interaktionen oder Interaktionen im gesamten Genom oder in einer Untergruppe von Genen zu untersuchen.
ChIP nutzt Antikörper, die selektiv Proteine erkennen und binden, darunter Histone, Histonmodifikationen, Transkriptionsfaktoren und Kofaktoren, um Informationen über den Chromatinzustand und die Gentranskription zu erhalten. Eine Kombination aus Proteomanalyse und molekularbiologischen Techniken, die bei ChIP zum Einsatz kommen, ermöglicht es, die Genexpression und -regulation in Zellen oder Geweben von Interesse zu verstehen.
Wann wird ChIP eingesetzt?
Typischerweise wird ChIP eingesetzt, um die relative Häufigkeit eines bestimmten Proteins oder einer bestimmten Proteinmodifikation in einer bestimmten Region im Genom zu ermitteln. ChIP kann zur Beantwortung einer Vielzahl wissenschaftlicher Fragen verwendet werden, die die Interaktion von Proteinen und Chromatin betreffen. ChIP kann beispielsweise dazu verwendet werden, das Vorhandensein bestimmter Proteine an verschiedenen Loci zu vergleichen, die verschiedenen Proteine in einer interessierenden Genomregion zu kartieren oder die Proteinbindung an ein induzierbares Gen als Reaktion auf einen Stimulus im Laufe der Zeit zu quantifizieren.
Wie funktioniert ChIP?
Das Prinzip der ChIP ist relativ einfach und beruht auf der Verwendung eines Antikörpers zur Isolierung oder Ausfällung eines bestimmten Proteins, Histons, Transkriptionsfaktors oder Cofaktors und des daran gebundenen Chromatins aus einer Proteinmischung, die aus Zellen oder Geweben extrahiert wurde. Daher auch der Name der Technik: Chromatin-Immunopräzipitation. Bei der ChIP-PCR oder ChIP-seq können die immunangereicherten DNA-Fragmente dann mit Hilfe von weithin verfügbaren PCR- oder qPCR-Reagenzien und Next Generation Sequencing (NGS)-Technologien identifiziert und quantifiziert werden.
Was ist natives ChIP (N-ChIP) im Vergleich zu vernetztem ChIP (X-ChIP)?
Es gibt 2 Arten von ChIP-Techniken, die je nach Fragestellung und Ausgangsmaterial für das Experiment durchgeführt werden können: 1) natives ChIP (N-ChIP) und 2) vernetztes ChIP (X-ChIP). Beide Arten von ChIP haben Vor- und Nachteile:
- Bei N-ChIP wird kein Fixierungsmittel verwendet, um Proteine mit dem Chromatin zu vernetzen. Stattdessen wird das native Chromatin aus Zellkernen isoliert, die mit einer Nuklease verdaut werden. Da die Antikörper gegen unfixierte Antigene gebildet werden, bietet N-ChIP den Vorteil einer besseren Erkennung und Bindung der Antikörper an ihre Zielantigene. Aufgrund der hohen Abundanz von Histonproteinen ist für die nachgeschaltete Analyse möglicherweise keine PCR erforderlich. Diese Vorteile machen N-ChIP zwar zu einer attraktiven Methode, doch kann sie nur zum Nachweis von Histonen eingesetzt werden. Darüber hinaus kann der Verlust der Proteinbindung während des Chromatindigestions und der Immunpräzipitation die Daten verfälschen oder die ordnungsgemäße Analyse behindern.
- Bei X-ChIP werden chemische Fixierungsmittel wie Formaldehyd verwendet, um das Protein von Interesse mit der DNA zu vernetzen, und die Fragmentierung des Chromatins wird durch Sonikation oder Nukleaseverdauung erreicht. Der Vorteil von X-ChIP besteht darin, dass es mit Histon- und Nicht-Histon-Proteinen durchgeführt werden kann und im Allgemeinen weniger zelluläres Ausgangsmaterial als N-ChIP benötigt. X-ChIP minimiert auch die Gefahr des Verlusts von Chromatinproteinen während der Extraktion und ermöglicht so den Nachweis vorübergehender Proteininteraktionen. Der Fällungsschritt ist jedoch weniger effizient, und für die nachgeschalteten Analysen ist eine DNA-Amplifikation mittels PCR erforderlich.
Welche Arten von ChIP-Assays gibt es?
Nach Abschluss der Chromatin-Immunpräzipitation können verschiedene nachgeschaltete Analysen des gereinigten Chromatins und der assoziierten Proteine, Histone, Transkriptionsfaktoren und Cofaktoren durchgeführt werden. Die gebräuchlichsten Methoden für die Einzelgenanalyse und die Analyse des gesamten Genoms sind qPCR und ChIP-seq. PCR und ChIP-Chip sind ebenfalls Optionen für die nachgeschaltete Analyse.
5.1 Was sind die Vorteile der ChIP-PCR?
ChIP-PCR wird durchgeführt, um Histonmodifikationen und/oder Proteinbindungen an eine bekannte Untergruppe von Zielloci im Genom zu analysieren. Bei der ChIP-PCR werden mit dem Immunsystem angereicherte DNA-Fragmente identifiziert und mit Hilfe weithin verfügbarer PCR- oder qPCR-Reagenzien und -Technologien quantifiziert. Mit der ChIP-qPCR lassen sich schnelle und quantitative Vergleiche spezifischer Regionen innerhalb des Genoms über mehrere Proben hinweg durchführen. Dies ist kostengünstiger und zeitsparender als Ganzgenom-Sequenzierungsmethoden.
5.2 Welche Vorteile bietet die ChIP-Chip-Technologie?
Die ChIP-Chip-Technologie bezieht sich auf die Verwendung eines DNA-Mikroarray-Chips zur Analyse von mit ChIP-Immunzellen angereicherten DNA-Fragmenten. Die Verwendung der Genom-Tiling-Microarray-Technologie ermöglicht eine Ganzgenomanalyse von Proteinen, die an isolierte DNA gebunden sind, und erzeugt eine hochauflösende Genomkarte von Proteinbindungen und Proteinmodifikationen. Der ChIP-Chip kann sowohl in der Grundlagenforschung als auch in der krankheitsbezogenen Forschung eingesetzt werden. Beispielsweise können damit die Bindungsstellen von Transkriptionsfaktoren, Enhancern und Repressoren identifiziert und diese Arten von gebundenen Proteinen in Kontroll- und pathologischen Proben verglichen werden. Da die Kosten für NGS jedoch erheblich gesunken sind und mit ChIP-seq ähnliche Ergebnisse erzielt werden können, entscheiden sich immer mehr Menschen für die Durchführung von ChIP-seq anstelle von ChIP-chip.
5.3 Was sind die Vorteile von ChIP-seq?
Ähnlich wie ChIP-chip liefert ChIP-seq Informationen über genomweite Proteinbindung. Im Gegensatz zum ChIP-Chip verwendet ChIP-seq jedoch die NGS-Technologie, um DNA-Fragmente zu identifizieren und sie mit dem gesamten Genom abzugleichen.
Moderne DNA-Amplifikationstechnologien ermöglichen robuste Analysen, die innerhalb weniger Tage mit einer geringen Menge an Ausgangs-DNA durchgeführt werden können. Wenn das Ausgangsmaterial knapp ist, haben diese technologischen Fortschritte bei den Bibliotheksvorbereitungsmethoden ChIP-seq-Experimente möglich gemacht.
Darüber hinaus ermöglicht eine neue Technologie, bei der DNA-Proben mit kurzen Sequenzen, den so genannten Barcodes, eindeutig markiert werden, die Zusammenführung einzelner Fragmente in einer einzigen Sequenzierspur für die Multiplexanalyse. Dies hat die Effizienz erheblich gesteigert und die Kosten für DNA-Sequenzierungsexperimente gesenkt, was ChIP-seq-Anwendungen weiter begünstigt.
Aufgrund der Fortschritte in der DNA-Sequenzierungstechnologie besteht der Vorteil von ChIP-seq darin, dass eine große Anzahl von ChIP-angereicherten DNA-Proben in relativ kurzer Zeit kostengünstig sequenziert werden kann, und zwar mit höherer Sensitivität und Genauigkeit als bei ChIP-Chips.
Welche sind die verschiedenen Schritte im ChIP-Assay?
ChIP-Assays folgen einem allgemeinen Protokoll:
- Vernetzung von Proteinen mit DNA nur für X-ChIP
- Zelllyse
- Chromatinfragmentierung durch Verdauung (für X-ChIP und N-ChIP) oder sonikations-Scherung (nur für X-ChIP)
- Immunopräzipitation mit spezifischen Antikörpern
- DNA-Reinigung für nachfolgende Analysen
- DNA-Analyse mittels PCR, qPCR, Microarray oder NGS
Wichtig ist, dass in jedem Schritt positive und negative Kontrollen durchgeführt werden, um festzustellen, ob ein ChIP-Experiment erfolgreich war.
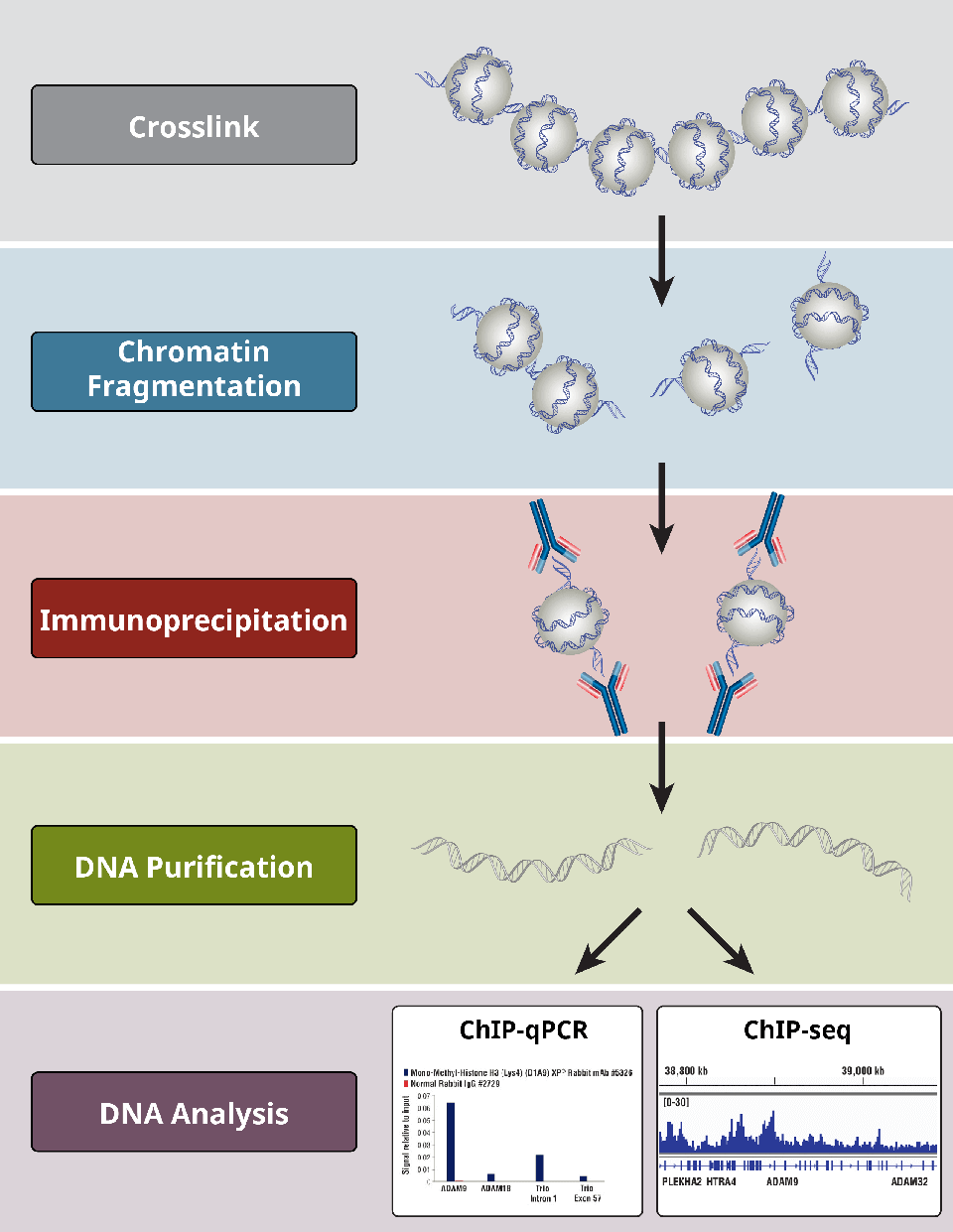
Übersicht über die kritischsten Schritte eines ChIP-Protokolls.
Übersicht über die kritischsten Schritte eines ChIP-Protokolls.
6.1 Wie vernetzt man Zellen und Gewebe für ChIP?
Vernetzungsreagenzien werden verwendet, um Proteine an die DNA zu „fixieren“, die sie binden. Für diese Fixierung werden in der Regel Reagenzien auf Formaldehydbasis verwendet. Zellen und Gewebe werden in der Regel auf ähnliche Weise fixiert, aber Gewebe erfordert eine längere Fixierungszeit und eine schnellere Fixierung, um das Zielgewebe schnell zu durchdringen, bevor es zu degenerieren beginnt.
Eine Überfixierung von Chromatin kann die Effizienz der Fragmentierung durch Beschallung verringern und außerdem die Bindung von Antikörpern an ihre Zielproteine hemmen. Daher sollte die Fixierungszeit empirisch bestimmt werden, um eine maximale Antikörper-Antigen-Bindung zu ermöglichen und gleichzeitig eine ideale Vernetzung der Proteine mit ihrer Ziel-DNA zu erreichen.
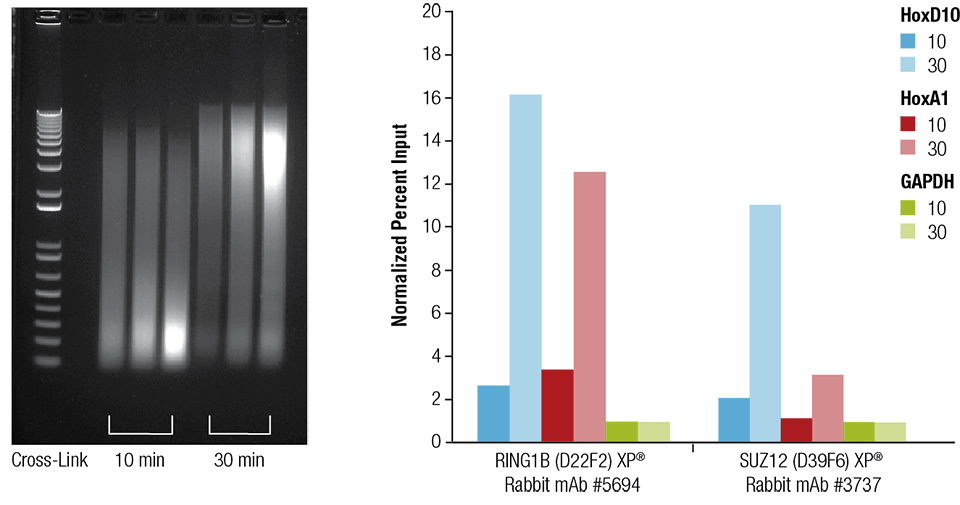
Mausherz (H), Gehirn (B) und Leber (L) wurden wie angegeben 10 oder 30 Minuten lang vernetzt (linkes Feld). Das Chromatin wurde präpariert und 4 Minuten lang beschallt. ChIP wurde mit Chromatin aus Herzgewebe mit den angegebenen Antikörpern unter Verwendung des SimpleChIP® Plus Sonication Chromatin IP Kit #56383 durchgeführt, und die angereicherte DNA wurde durch Echtzeit-PCR mit Primern für die angegebenen Gene quantifiziert (rechtes Feld). Die Menge der immunpräzipitierten DNA in jeder Probe ist als normalisiertes Signal im Vergleich zu den negativen Loci GAPDH dargestellt, was gleich 1 ist.
Mausherz (H), Gehirn (B) und Leber (L) wurden wie angegeben 10 oder 30 Minuten lang vernetzt (linkes Feld). Das Chromatin wurde präpariert und 4 Minuten lang beschallt. ChIP wurde mit Chromatin aus Herzgewebe mit den angegebenen Antikörpern unter Verwendung des SimpleChIP® Plus Sonication Chromatin IP Kit #56383 durchgeführt, und die angereicherte DNA wurde durch Echtzeit-PCR mit Primern für die angegebenen Gene quantifiziert (rechtes Feld). Die Menge der immunpräzipitierten DNA in jeder Probe ist als normalisiertes Signal auf den negativen Locus GAPDH dargestellt, was 1 entspricht.
6.2 Wie fragmentiert man Chromatin?
Die Fragmentierung von Chromatin ist für den Erfolg eines ChIP-Experiments unerlässlich. Die Fragmentierung des Chromatins ist notwendig, um das Chromatin zu solubilisieren und seine Kopräzipitation zu ermöglichen. Darüber hinaus hängt die Auflösung des ChIP-Assays von der Fragmentierung des Chromatins ab, da die Größe der DNA-Fragmente die Auflösung des ChIP-Assays bestimmt.
Beim enzymatischen Verdau wird Mikrokokzennuklease (MNase) verwendet, die doppelsträngige DNA zwischen Nukleosomen spaltet und so Chromatinfragmente erzeugt. Während ein vollständiger MNase-Verdau DNA-Fragmente von 150 Basenpaaren (Mono-Nukleosomen) erzeugt, entstehen bei einem unvollständigen Verdau DNA-Fragmente zwischen 150 und 750 Basenpaaren (Mono-, Di- und Tri-Nukleosomen). Bei der Sonikation wird zur Fragmentierung des Chromatins mechanische Kraft eingesetzt. Die Sonikation fragmentiert das Chromatin zwischen und innerhalb der Nukleosomen und erzeugt eine Reihe von Chromatinfragmenten zwischen 150 und 1000 Basenpaaren.
Beim X-ChIP wird das Chromatin entweder durch enzymatischen Verdau oder durch Sonikation abgeschert. Die Beschallungsbedingungen im Beschallungs-ChIP-Protokoll sollten empirisch ermittelt werden, da sie sich je nach Zelltyp und Versuchsbedingungen unterscheiden. Die Verdauungsbedingungen sind bei verschiedenen Zelltypen und Geweben konsistenter, aber die Größe der Chromatinfragmente sollte dennoch vor der IP analysiert werden.
Bei N-ChIP werden Nukleasen zur Fragmentierung des Chromatins verwendet, um die Proteinbindung in unfixierten Proben zu erhalten. Die Nuklease-Fragmentierung sollte ebenfalls empirisch bestimmt werden, um eine Überdigestion des Chromatins zu minimieren.
6.3 Warum enzymatischer Verdau für ChIP?
Für N-ChIP muss ein Nuklease-Verdau verwendet werden, da die Proteine nicht mit der DNA vernetzt sind und die rauen Bedingungen, die mit der Fragmentierung auf der Basis von Sonikation verbunden sind, zu einer Dissoziation der Chromatinproteine von der DNA führen würden. N-ChIP ist ideal für die Analyse von Histonprotein-DNA-Interaktionen, da die Histon-DNA-Bindung sehr stark und stabil ist. N-ChIP eignet sich jedoch nicht für die Analyse der Chromatinbindung von Transkriptionsfaktoren und Cofaktoren.
Zur Fragmentierung des Chromatins bei X-ChIP kann entweder ein enzymatischer Verdau oder eine Beschallung verwendet werden. Zu den Vorteilen des enzymatischen Verdaus gehören die Konsistenz der Fragmentierung und milde Fragmentierungsbedingungen (geringere Hitze und Detergenz), die die Integrität des Chromatins und der Antikörperepitope besser bewahren, was zu einer erhöhten Immunanreicherung von Transkriptionsfaktor- und Cofaktor-gebundenem Chromatin führt.
6.4 Warum wird Chromatin für ChIP durch Beschallung fragmentiert?
Im Gegensatz zur Chromatinfragmentierung, die durch enzymatischen Verdau erreicht wird, beruht die Beschallung auf mechanischen Kräften, um Chromatin in kleinere Stücke zu fragmentieren. Die ideale Größe von Chromatinfragmenten für die Immunanreicherung liegt zwischen 200 und 1000 Basenpaaren. Die Sonikation ist die traditionelle Methode zur Fragmentierung von Chromatin und kann mit einem herkömmlichen Sondensonicator oder mit moderneren Wasserbadsonicatoren durchgeführt werden, die eine gezieltere Sonikation ermöglichen. Die Sonikation erzeugt wirklich randomisierte Chromatinfragmente; sie erfordert jedoch eine umfassende Optimierung für verschiedene Zelllinien und Gewebe und ist von Experiment zu Experiment schwer zu reproduzieren. Der Bedarf an stark waschaktiven Puffern und die bei der Beschallung erzeugte Wärme können die Integrität des Chromatins und der Antikörperepitope auf Chromatinproteinen schädigen.
6.5 Optimierung der Chromatinsonikation für ChIP
Bei der auf Beschallung basierenden Chromatinfragmentierung werden traditionell stark waschaktive Puffer verwendet und Wärme erzeugt, die beide die Integrität des Chromatins und der Antikörperepitope schädigen können. Daher muss die zur Fragmentierung des Chromatins verwendete Beschallungsmenge für verschiedene Zelllinien und Gewebe experimentell bestimmt werden. Es muss die minimale Beschallungsmenge ermittelt und verwendet werden, die erforderlich ist, um DNA-Fragmente mit 150 bis 1000 Basenpaaren zu erzeugen, um die Schädigung des Chromatins zu minimieren.
Bevor ein vollständiger ChIP-Assay mit nachgeschalteter Analyse durch qPCR, DNA-Chip oder NGS durchgeführt wird, sollte die Gelelektrophorese zur Analyse von Chromatinproben verwendet werden, die für verschiedene Zeiten beschallt wurden. Die Fragmentgröße hängt von der Beschallungsdauer ab – die Fragmentgröße nimmt mit zunehmender Beschallungsdauer ab. Die Daten legen jedoch nahe, dass längere Beschallungszeiten nicht zu besseren Ergebnissen führen. Daher ist es eine einfache Methode, gereinigte immunpräzipitierte DNA auf einem Gel laufen zu lassen und die ideale Fragmentgröße zu bestimmen, um die minimale Beschallungsdauer zu ermitteln, die für die gewünschte DNA-Größe erforderlich ist, und um unnötige Schäden am Chromatin zu vermeiden.

Enzymverdautes Chromatin wurde auf einem Agarosegel laufen gelassen. Spur 1 zeigt Chromatin, das nicht ausreichend verdaut ist. Spur 2 zeigt richtig verdautes Chromatin, und Spur 3 zeigt überverdautes Chromatin.
Enzymverdautes Chromatin wurde auf einem Agarosegel laufen gelassen. Spur 1 zeigt Chromatin, das nicht ausreichend verdaut ist. Spur 2 zeigt richtig verdautes Chromatin, und Spur 3 zeigt Chromatin, das überverdaut ist.
6.6 Wie wählt man einen Antikörper für ChIP?
Die Wahl eines geeigneten Antikörpers für ein ChIP-Experiment ist entscheidend für dessen Erfolg. Die in einem ChIP-Experiment verwendeten Antikörper sollten für das betreffende Protein spezifisch sein und eine hohe Affinität für das Antigen aufweisen. Die beste Wahl eines Antikörpers für ein ChIP- oder ChIP-seq-Experiment ist ein ChIP- oder ChIP-seq-validierter Antikörper. Steht kein ChIP-validierter Antikörper für das betreffende Gen zur Verfügung, ist die nächstbeste Wahl ein Antikörper, der in IP validiert wurde. Es ist wichtig zu beachten, dass nicht alle IP-validierten Antikörper auch bei ChIP funktionieren und nicht alle ChIP-validierten Antikörper auch bei ChIP-seq. Je mehr ein Antikörper für andere Anwendungen wie Western, IP, IF, Flow und IHC validiert ist, desto mehr Vertrauen kann man in die Leistung und Spezifität des Antikörpers haben. Auf die Validierung eines Antikörpers muss die empirische Bestimmung der optimalen Konzentration des Antikörpers zusammen mit den IP-Waschbedingungen folgen.
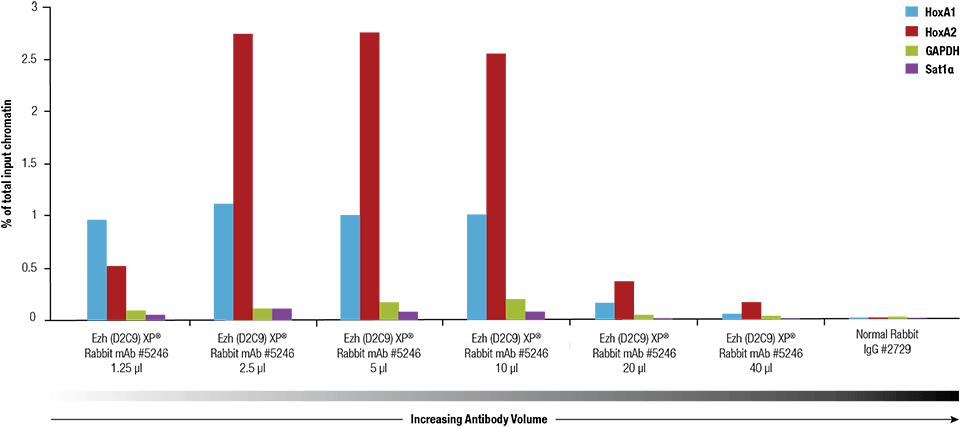
Ezh2 (D2C9) XP® Rabbit mAb #5246 wurde mit dem SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 auf quervernetztem Chromatin titriert, das aus 4 x 106 NCCIT-Zellen hergestellt wurde.
Ezh2 (D2C9) XP® Rabbit mAb #5246 wurde mit dem SimpleChIP® Plus Enzymatic Chromatin IP Kit #9005 auf vernetztes Chromatin titriert, das aus 4 x 106 NCCIT-Zellen präpariert wurde.
6.7 Durchführung der Immunpräzipitation
Antikörper werden verwendet, um ein Protein von Interesse und die daran gebundene DNA einzufangen. Die Antikörperkonzentration sollte empirisch bestimmt werden, wobei ein allgemeiner Ausgangspunkt 0,5-2,0 μg Antikörper pro 10 μg Chromatin-DNA (entspricht etwa 4 x 106 Zellen) ist. Die Stringenz der Puffer und die Waschzeiten sollten ebenfalls empirisch bestimmt werden, da sie von der Affinität des Antikörpers zu seinem Zielantigen abhängen. Üblicherweise werden Antikörper-Chromatin-Inkubationen 2 Stunden bis über Nacht durchgeführt.
Antikörper-Antigen (+DNA)-Komplexe werden durch Affinität an ein Antikörper-bindendes Harz gebunden. Bei ChIP-Experimenten besteht dieses Harz in der Regel aus magnetischen, Sepharose- oder Agarose-Beads in ChIP-Qualität, die mit Protein A und/oder Protein B konjugiert sind. Antikörper binden Protein A- und/oder Protein G-Beads mit unterschiedlicher Affinität, je nach der Spezies, in der sie entwickelt wurden, und dem IgG-Subtyp ihrer schweren Kette. Die Beads werden in der Regel 2 bis 4 Stunden lang mit Antikörper-Chromatin inkubiert.
Waschschritte sind erforderlich, um nicht an Antikörper gebundenes Chromatin zu entfernen, gefolgt von der Umkehrung von Querverbindungen (für X-ChIP) und der Reinigung der DNA. Darüber hinaus muss eine IgG-Kontroll-IP durchgeführt werden, um den Hintergrund (Signal:Rauschen) zu bestimmen. Positivkontroll-Antikörper (z. B. Gesamt-Histon H3) und/oder Positivkontroll-qPCR-Primer (für bekannte positive und negative Zielprotein-Bindungsorte) müssen ebenfalls einbezogen werden, um unspezifische Bindungen zu bestimmen. Um optimale Ergebnisse zu erzielen, sollte die QC der Chromatin-IP durch qPCR vor der nachgeschalteten NGS-Analyse durchgeführt werden.
6.8 Elution des Chromatins von den Protein-A/G-Beads
Das Chromatin wird mit Hilfe von Detergenz und Hitze von den Protein-A/G-Beads eluiert. Langsames „Vortexen“ oder Mischen ist erforderlich, um die Beads in Suspension zu halten und die Elution des Chromatins zu verstärken.
6.9 Umkehrung der Chromatin-Crosslinks
Die Crosslinks werden durch hohe Hitze und hohe Salzkonzentration umgekehrt (beides sind wichtige Komponenten). Außerdem wird Proteinase K hinzugefügt, um die assoziierten Chromatinproteine zu verdauen, und es werden Antikörper hinzugefügt, die eine effizientere DNA-Reinigung ermöglichen.
6.10 Wie wird die DNA gereinigt
Nachdem die Chromatin-Crosslinks entfernt wurden, wird die DNA entweder mit klassischen Phenol-Chloroform-Methoden und anschließender Ethanol-Präzipitation oder mit säulenbasierten DNA-Reinigungskits gereinigt.
Analyse der angereicherten DNA
Nach der Aufreinigung der DNA können verschiedene nachgeschaltete Analysen durchgeführt werden, darunter ChIP-PCR, ChIP-qPCR, ChIP-Chip und ChIP-seq.
7.1 ChIP-PCR- und ChIP-qPCR-Analyse
ChIP-PCR- und ChIP-qPCR-Analysen eignen sich am besten für die Analyse einzelner Gene und können zur Amplifikation und Quantifizierung spezifischer DNA-Fragmente auf schnelle und kostengünstige Weise verwendet werden.
7.2 ChIP-Chip-Analyse
Die ChIP-Chip-Analyse nutzt Tiling-DNA-Microarray-Chips, um eine genomweite, hochauflösende Karte der Proteinbindung und -modifikation zu erstellen.
7.3 ChIP-seq-Analyse
Die ChIP-seq-Analyse nutzt Standard-NGS-Technologie, um gereinigte DNA mit zuvor annotierten ganzen Genomen abzugleichen, um genomweite Proteinbindungsprofile zu identifizieren.